第三章 DNA 的优化
3.1 试验材料与方法
3.1.1 试验材料和药品
3.1.1.1 试验材料
试验材料同 2.1.1.1。
3.1.1.2 试剂
CTAB、5mol/L NaCl、氯仿:异戊醇(24:1)、无水乙醇、异丙醇、TE 缓冲液、蒸馏水、液氮、Golden view(艾德莱生物试剂有限公司)、Loading Buffer(天根生物试剂有限公司)、Marker DL2000(天根生物试剂有限公司)、PVP、β-巯基乙醇、RnaseA(10mg/mL).
3.1.2 试验方法
对 CTAB 法进行改良,优化提取的 DNA,主要从以下几方面改良:
3.1.2.1 多酚类物质的去除
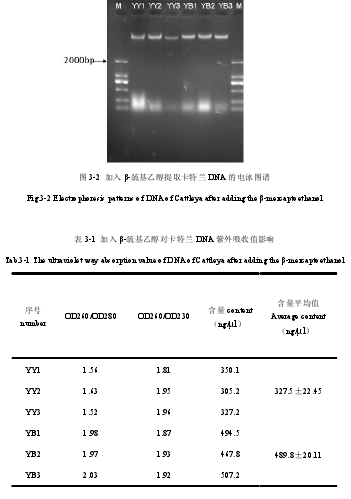
在加入 CTAB 提取液时迅速加入 14?l β-巯基乙醇,进行水浴加热,观察现象,拍照得到图 3-1,提取 DNA 之后进行电泳检测得到电泳图,见图 3-2。
3.1.2.2 多糖等的去除
卡特兰叶片多汁,含糖量较高,紫外吸收值检测也显示含有糖类盐类等小分子。对于多糖等小分子杂质的去除,前人的研究主要有加入 PVP 去多糖法和无水乙醇沉淀阶段加入 NaCl 溶液去多糖法。所以本次主要设置以下几组试验:(1)只在 CTAB 溶液中加入2g PVP,沉淀阶段方法无变化,同 2.1.2。(2)提取液中不加 PVP,在无水乙醇沉淀 DNA阶段,加入 0.5 倍体积的 5mol/L NaCl 溶液,混匀后,再加入预冷的无水乙醇。(3)既在提取缓冲液中加入 PVP,又在无水乙醇沉淀之前加入 0.5 倍体积的 5mol/L NaCl 溶液。(4)用异丙醇代替无水乙醇沉淀 DNA。试验结果见图 3-3 和图 3-4。
3.1.2.3 蛋白的去除
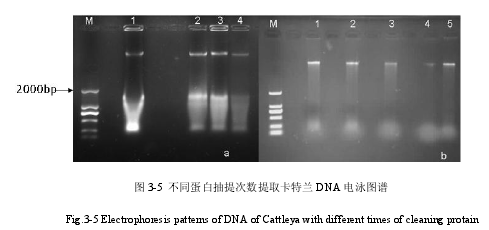
对于蛋白的去除主要是采用氯仿异戊醇抽提次数增加的方式,进行两次抽提。设置两组试验,一组为用氯仿异戊醇抽提一次,另一组为用氯仿异戊醇抽提两次,将提取的DNA 溶于 TE,进行电泳检测,拍照,得到图 3-5。
3.1.2.4RNA 的去除
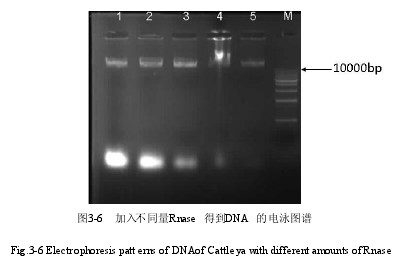
卡特兰 RNA 污染严重,对于 RNA 的去除采用加入 RnaseA 的方法,设计试验,在上述方法提取出来的 DNA 中分别加入 0.1?l,0.2?l,0.3?l,0.4?l,0.5?l 的 RnaseA, 37℃水浴 30min,对 DNA 进行电泳检测,试验后将胶放在紫外透射仪下拍照得到图 3-6。
3.2 优化结果与分析
3.2.1 多酚类物质的去除
从图 3-1a 和图 3-1b 可以看出,取自同一材料同一部位的样品,研磨后未加入 β-巯基乙醇的 CTAB 溶液在水浴 20min 时明显变褐色,水浴 40min 结束时褐化更明显,而加入β-巯基乙醇的溶液还保持着绿色,说明 β-巯基乙醇能够在卡特兰 DNA 提取过程中起到防止褐化的作用。从图 3-1c 可以看出在 DNA 沉淀后用酒精清洗时,加入 β-巯基乙醇的实验组 DNA 为白色,而未加入 β-巯基乙醇的为浑浊的黄色,且用 70%酒精清洗 3 次后黄色仍为褪去。(见图 3-1)观察 DNA 的电泳结果,M 为 Marker DL2000,YY1,YY2,YY3 为对照组,即未加入β-巯基乙醇的实验组,YB1,YB2,YB3 为加入 β-巯基乙醇的实验组。从电泳结果可以看出,未加入 β-巯基乙醇依旧能够提取出来 DNA。(见图 3-2)在 ODA260/OD280 值上可以看出,未加入 β-巯基乙醇组的 DNAOD260/OD280 在 1.6左右,污染严重,加入 β-巯基乙醇组的 DNAOD260/OD280 值明显上升,接近 2.0。观察DNA 含量发现,未加入 β-巯基乙醇组的样品提取出来 DNA 含量最高为 400.1ng/?l,平均值为327.5±22.45 ng/?l,而加入β-巯基乙醇组的样品提取出来DNA最少为467.8 ng/?l,平均值为 489.8±20.11ng/?l,表明加入 β-巯基乙醇能够明显提高 DNA 的提取量。即加入β-巯基乙醇不仅能够提高 DNA 质量,减少多酚杂质污染,还能够提高 DNA 含量。(见表3-1)

3.2.2 多糖和盐离子的去除
从图 3-3a 和图 3-3b 可以看出,不同的沉淀方法对 DNA 影响效果较大。1 号加入无水乙醇后能够看到少许絮状沉淀, 2 号加入异丙醇后也出现少许絮状沉淀(见图 3-3a)。
图 3-3b 为在实验开始阶段加入了 PVP,沉淀时用无水乙醇沉淀,絮状沉淀十分明显。3号试管在上清液中加入 NaCl 溶液后未见变化,混匀后加入 2 倍体积无水乙醇迅速出现白色粉末状沉淀,且在冰箱中放置片刻后沉淀越来越多(见图 3-3a),用 70%酒精清洗三次仍为图 3-3c 所示,白色颗粒物虽然有所减少,但是量仍旧很多。说明前人提到的在无水乙醇沉淀步骤时加入 NaCl 溶液的方法并不适用于卡特兰,而无水乙醇沉淀的方法和异戊醇沉淀的效果从表象上相似,沉淀效果一般,实验中加入 PVP 然后用无水乙醇沉淀的DNA 得到的絮状沉淀最多最明显。(见图 3-3)

图中 P1,P2,P3 为加入 PVP 用无水乙醇沉淀的实验组。C1,C2,C3 为用 NaCl 溶液和无水乙醇沉淀的实验组。B1,B2,B3 为加入 PVP 并且用 NaCl 和无水乙醇沉淀的实验组。
T1,T2,T3 为原始的用无水乙醇沉淀的实验组。Y1,Y2,Y3 为用异丙醇沉淀的实验组。M 为Marker DL2000(见图 3-4)。从电泳图可以发现 C1,C2,C3 和 B1,B2,B3 样本条带拖尾明显,且 DNA 条带发生变形,说明杂质污染严重。P1,P2,P3 样本、T1,T2,T3 样本和 Y1,Y2,Y3样本 DNA 条带较清晰,其中 P1,P2,P3 样本 DNA 最清楚,且拖尾最少。
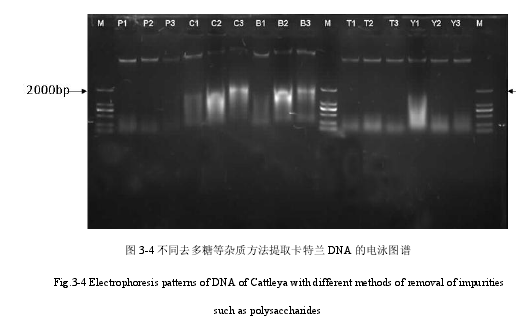
表 3-2 中,P1,P2,P3 为加入 PVP 用无水乙醇沉淀的实验组。T1,T2,T3 为原始的用无水乙醇沉淀的实验组。C1,C2,C3 为用 NaCl 溶液和无水乙醇沉淀的实验组。B1,B2,B3 为加入 PVP 且用 NaCl 和无水乙醇沉淀的实验组。。Y1,Y2,Y3 为用异丙醇沉淀的实验组(见表 3-2)。数据显示加入 PVP 且用无水乙醇沉淀的 DNA OD260/OD230 值在 2.0 附近,并且大部分大于 2.0,效果最好,DNA 的含量最高,平均值为 427.2±18.75ng/?l,略高于仅用无水乙醇沉淀的 DNA,无水乙醇沉淀和异戊醇沉淀得到 DNA 质量相近,OD260/OD230 值在 1.74~1.91 之间,含量差异不显着,DNA 含量均值分别为 392.8±9.95ng/?l 和 329.6±32.90ng/?l。在实验中加入 NaCl 溶液效果较差,C1,C2,C3,B1,B2,B3 的DNA 含量均值分别为 169.4±14.99ng/?l 和 125.0±11.70ng/?l。OD260/OD230 值均小于1.3,说明杂质污染严重,表明在溶液中加入 NaCl 的方法不适合卡特兰 DNA 的提取(见表 3-2)。
3.2.3 蛋白的去除
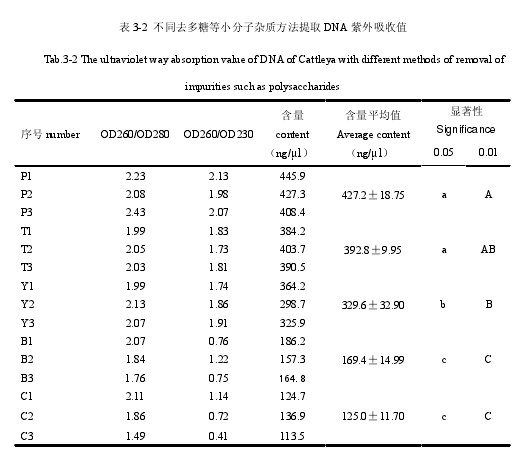
图 3-5a 为用氯仿异戊醇抽提一次得到的 DNA 进行的电泳,图 3-5b 为用氯仿异戊醇抽提两次得到的 DNA 的电泳图。M 为 Marker DL2000。从 3-5a 图可以明显看到胶孔处有亮带,说明 DNA 有蛋白质污染,进行两次抽提后得到电泳图 3-5b 看到胶孔处明显变干净,说明多次抽提对于 DNA 蛋白质的去除作用明显(见图 3-5)。
3.2.4 RNA 的去除
1,2,3,4,5 号泳道分别为试管中加入 0.1?l,0.2?l,0.3?l,0.4?l,0.5?l 的 Rnase,得到的 DNA的电泳图片,从实验中可以看出随着 Rnase 量的增加,RNA 带的亮度减小,说明 RNA含量变少,同时 DNA 的带也逐渐变弱,可能是 Rnase 能够消化部分的 DNA。同时还发现在抽提两次的胶孔处又出现了白色亮带,说明又有蛋白质污染。原因可能是因为 Rnase去蛋白酶属于蛋白质,在加入 Rnase 后虽能有效去除 RNA,但是又引入了新的杂质蛋白质。所以应该在加入 Rnase 之后再进行一次抽提,以除去 DNA 中的蛋白质(见图 3-6)。
3.3 小结
本部分试验采取分部优化的方式,试验结果发现在 CTAB 溶液中加入β-巯基乙醇能够有效防止 DNA 褐化,而且能够防止 DNA 降解,提高 DNA 的提取量。对于卡特兰多糖以及盐类等分子杂质的去除使用加入 CTAB 加 PVP 并且用预冷无水乙醇沉淀的方法最好,优于异丙醇沉淀,而且加入 NaCl 的方法不适合卡特兰。提取卡特兰过程中,用氯仿异戊醇进行两次抽提效果较好,蛋白含量明显减少。再通过加入 RNAse 的方法就能够去除样品中残存的 RNA。随着加入 Rnase 的含量 RNA 杂质减少,但同时 DNA 含量也在减少,而且蛋白质有所增加,所以最后需要再进行一次氯仿异戊醇抽提。所以,实验表明最适合的 Rnase 的量为 0.5?l,在此浓度下可以消除 RNA 同时还能保证 DNA 的含量。
采用上述分部试验得到的结果,将其组合成完整的方法步骤进行试验,即①将用液氮充分研磨后,装在 2ml 离心管中,加入 700?l 含 2%PVP 的 CTAB 溶液,和 14?lβ-巯基乙醇,震荡摇匀,水浴 40min。②加入 700?l 氯仿异戊醇,轻缓混匀,离心 12000 转10min。③吸取 400-500?l 上清液,转入新的离心管中。继续加入等体积氯仿异戊醇,进行二次抽提。④向吸出的上清中加入 0.5?l 的 Rnase,37℃水浴 30min。⑤向上清液中加入等体积氯仿异戊醇,12000 转离心 10min。⑥吸取上清,加入 2 倍体积-20℃预冷的无水乙醇,颠倒混匀,放入-20℃静置 40min,得到沉淀 DNA。⑦用 70%乙醇洗三次然后用无水乙醇洗涤晾干,晾干后溶于 200?lTE 溶液中,保存于 4℃。
按照此试验步骤提取 DNA 得到电泳图见图 3-7,紫外吸收值见表 3-3。从电泳图可以看出,DNA 条带清晰,胶孔清晰,没有条带弥散的现象,也没有 RNA 污染,说明 DNA纯净(见图 3-7)。用此方法提取的 DNA 的 OD260/OD280 在 1.8 到 2.0 之间,OD260/OD230都大于 2,说明 DNA 质量很纯净,不含杂质。含量在最少为 287.3ng/?l 以上,能够符合现在大部分分子技术对 DNA 的需求,达到了 DNA 优化的目的(表 3-3)。