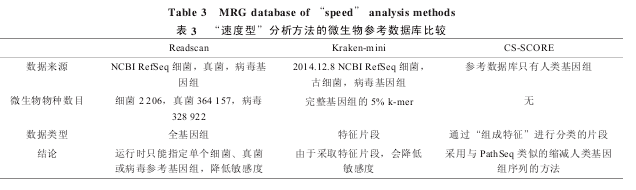
参考微生物数据库是否全面对软件的检测结果影响较大,因此这里对三种方法的数据库进行了综合比较,结果如表3.
“速度型”检测方法针对微生物检测过程中的速度问题进行了优化(环节 B),如 Kraken 的精简数据库法,CS-SCORE 的基于 cs-score 的比对方法等,都在很大程度上加速了比对核心流程。但是速度的增加会不可避免地带来精度的下降,比如降低参考基因组大小会增加检测速度,但是会降低比对过程中正确匹配的reads 数量,从而降低敏感度,增加参考基因组大小能够提高结果正确性,但是由于数据量增加,处理速度也会相应降低。目前最新的检测方法一般提供了两种模式。第一种模式为只将样本序列与 MRG 进行比对的快速模式,其优点在于检测速度快,缺点在于无法检测MRG 中缺失的序列;另一种为逐步清除人类基因序列的方式,优点是能够检测未知微生物序列,缺点是检测速度较低。用户可以根据需求选择适合自己的检测模式,这也是在微生物检测的速度和精度之间进行平衡的最佳方案。
4总结与展望。
基于 NGS 技术的微生物检测数据分析方法具有无需预先培养样本、灵敏度高、能够检测未知的微生物的特点,为疾病防控和生物食品安全提供了新的解决方案。本文对目前常见的 12 种基于 NGS的微生物检测方法进行了简要介绍和比较研究,对各个检测方法的软件流程和数据处理方案分别进行了环节上和优化方向上的分析。在数据分析环节方面,将基于NGS 的微生物检测数据分析方法分为“基础型”,“功能型”,“速度型”,“完整型”等四种类型。在优化方向方面,侧重两个性能指标:速度、精度以及四个应用因素:计算资源、体系结构、能耗和可扩展性方面进行比较分析。通过对现有数据分析方法及其实现的总结评价,希望为生物和计算领域的相关研究提供参考价值。
本实验室也对该领域的研究进行了一定的探索:李定辰等[48]针对从非培养样本中鉴定未知病原微生物的问题,从软件层面对微生物检测流程进行了分析评估的软件包括序列比对软件Bowtie2、BWA、 BLAST + 、 MUMer[49];基因组拼接软件Velvet[50]、 SOAPdenovo[51]; 后 续 处 理 软 件BEDTools[52]、 MEGAN4[53]、 MAUVE[54]、 IGV[55]、Circos[56]等。分别从常规病原体检测、高突变率下的病原体检测、不同测序深度以及读长下的病原体检测、数据量不充分时的病原体检测和混合样品中的病原体检测几个方面,对以上软件做了系统评估, 其研究和评估结果将有助于指导以后临床病原微生物鉴定分析工作。叶福强等[57]针对胆总管结石的微生物组研究问题,使用宏基因组测序手段,对15 位中国胆总管结石患者的胆汁样本进行全宏基因组鸟枪法测序和 16 S 核糖体扩增子测序分析,其研究发现了13 个之前未报道的高基因组覆盖度的胆道细菌。还鉴别出与胆石形成汁耐受相关的基因,是微生物检测技术在鉴别新型微生物上的实际应用。王恒等[58]开发了“天河二号”上的基于 IntelMIC的高通量 DNA 序列比对并行软件,其中DNA 序列比对软件 MICA[57]结合天河二号超级计算机软硬件架构设计,能够充分发挥MIC 的并行潜力,具有接近线性加速比的扩展性能。李定辰的工作属于从细粒度上对基于NGS 的微生物检测流程做出分析,如果能够将粗粒度(检测方法)和细粒度(软件)分析相结合,则能够使人更清晰地分析和总结微生物检测的过程。叶福强的工作属于传统微生物检测方法的进一步发展,如果能够将16 S 分析和基于NGS 的微生物检测相结合,则能够使检测更加准确。王恒的工作能够推动基于 NGS 的微生物检测方法向速度更优方向发展。结合本实验室其他研究,则能够对相关领域的研究作出促进。另外本人所在课题组建立并发布一套用于综合评估基于NGS 微生物检测的计算分析方法的性能评测数据和工具集,包含不同突变率的测试数据、不同规模的测试数据以及与真实数据接近的模拟数据,相关学术成果已投稿PDP 2017 国际会议。
未来基于NGS 的微生物检测方法的发展方向可以在速度和精度上做重点优化,除此之外,现有计算分析方法在计算资源、能耗、体系结构等方面也都存在着优化空间。计算资源方面,通过精简参考基因集等方法能够有效降低运行内存;也可以通过专用硬件加速卡来解决,缩减微生物检测数据分析方法的处理时间,更好地面对生物威胁。在体系结构方面,可通过上传数据至云计算服务器,可以使检测方法有更高的操作系统适配性等。如GPU、ARM低能耗处理器以及FPGA都能够从不同层次上对检测方法进行加速或其他方面的优化。
除了计算技术方面的优化,生物技术的创新也能给微生物检测方法带来革新。未来生物技术的发展使测序技术向着高通量、低成本、长读取长度的方向发展,目前已接近实用的第三代测序技术具有超长读长的特点,一旦应用将会极大地改善微生物检测方法的流程。对微生物检测数据分析方法提出了新的要求,这就需要新的数据分析方法能够适应测序技术,在速度和精度上达到更高的标准,为微生物检测领域提供更快速、更准确的微生物检测方法。
参 考 文 献:
[1] Steingart K R, Henry M, Ng V, et al. Fluorescence versusconventional sputum smear microscopy for tuberculosis: asystematic review. The Lancet Infectious Diseases, 2006, 6 (9):570-581.
[2] Lemieux B, Aharoni A, Schena M. Overview of DNA chiptechnology. Molecular Breeding, 1998, 4(4): 277-289.
[3] Belgrader P, Benett W, Hadley D, et al. Rapid pathogen detectionusing a microchip PCR array instrument. Clinical Chemistry, 1998,44(10): 2191-2194.
[4] Call D R. Challenges and opportunities for pathogen detectionusing DNA microarrays. Critical Reviews in Microbiology, 2005,31(2): 91-99.
[5] Lazcka O, Del Campo F J, Munoz F X. Pathogen detection: aperspective of traditional methods and biosensors. Biosensors andBioelectronics, 2007, 22(7): 1205-1217.
[6] Schuster S C. Next-generation sequencing transforms today'sbiology. Nature, 2007, 200(8): 16-18.
[7] Barzon L, Lavezzo E, Costanzi G, et al. Next-generation sequencingtechnologies in diagnostic virology. Journal of Clinical Virology,2013, 58(2): 346-350.
[8] Reis-Filho J S. Next-generation sequencing. Breast CancerResearch, 2009, 11(3): 1-8.
[9] Metzker M L. Sequencing technologies-the next generation.Nature Reviews Genetics, 2010, 11(1): 31-46.
[10] Mandal P, Biswas A, Choi K, et al. Methods for rapid detection offoodborne pathogens: an overview. American Journal of FoodTechnology, 2011, 6(2): 87-102.
[11] Li R, Zhu H, Ruan J, et al. De novo assembly of human genomeswith massively parallel short read sequencing. Genome Research,2010, 20(2): 265-272.
[12] Li H, Homer N. A survey of sequence alignment algorithms fornext-generation sequencing. Briefings in Bioinformatics, 2010,11(5): 473-483.
[13] Mardis E R. Next-generation DNA sequencing methods. Annu RevGenomics Hum Genet, 2008, 9(3):87-402.
[14] Wang D G, Fan J B, Siao C J, et al. Large-scale identification,mapping, and genotyping of single-nucleotide polymorphisms inthe human genome. Science, 1998, 280(5366): 1077-1082.
[15] Kostic A D, Ojesina A I, Pedamallu C S, et al. PathSeq: software toidentify or discover microbes by deep sequencing of human tissue.Nature Biotechnology, 2011, 29(5): 393-396.
[16] Bhaduri A, Qu K, Lee C S, et al. Rapid identification of non-humansequences in high-throughput sequencing datasets. Bioinformatics,2012, 28(8): 1174-1175.
[17] Borozan I, Wilson S, Blanchette P, et al. CaPSID: A bioinformaticsplatform for computational pathogen sequence identification inhuman genomes and transcriptomes. BMC Bioinformatics, 2012,13(1): 157.
[18] Chen Y, Yao H, Thompson E J, et al. VirusSeq: software to identifyviruses and their integration sites using next-generation sequencingof human cancer tissue. Bioinformatics, 2013, 29(2): 266-267.
[19] Wang Q, Jia P, Zhao Z. VirusFinder: software for efficient andaccurate detection of viruses and their integration sites in hostgenomes through next generation sequencing data. PloS One, 2013,8(5): e64465.
[20] Naeem R, Rashid M, Pain A. READSCAN: a fast and scalablepathogen discovery program with accurate genome relativeabundance estimation. Bioinformatics, 2013, 29(3): 391-392.
[21] Wood D E, Salzberg S L. Kraken: ultrafast metagenomic sequenceclassification using exact alignments. Genome Biol, 2014, 15 (3):R46.
[22] Naccache S N, Federman S, Veeraraghavan N, et al. Acloud-compatible bioinformatics pipeline for ultrarapid pathogenidentification from next-generation sequencing of clinical samples.Genome Research, 2014, 24(7): 1180-1192.
[23] Scheuch M, H觟per D, Beer M. RIEMS: a software pipeline forsensitive and comprehensive taxonomic classification of reads frommetagenomics datasets. BMC Bioinformatics, 2015, 16(1): 1.
[24] Kilianski A, Carcel P, Yao S, et al. Pathosphere. org: pathogendetection and characterization through a web-based, open sourceinformatics platform. BMC Bioinformatics, 2015, 16(1): 1.
[25] Haque M M, Bose T, Dutta A, et al. CS-SCORE: Rapididentification and removal of human genome contaminants frommetagenomic datasets. Genomics, 2015, 106(2): 116-121.
[26] Wang Q, Jia P, Zhao Z. VERSE: a novel approach to detect virusintegration in host genomes through reference genomecustomization. Genome Medicine, 2015, 7(1): 1-9.
[27] Li Y, Wang H, Nie K, et al. VIP: an integrated pipeline formetagenomics of virus identification and discovery. ScientificReports, 2016, 6: 23374· 67·。
[28] Li H, Durbin R. Fast and accurate short read alignment withBurrows-Wheeler transform. Bioinformatics, 2009, 25 (14): 1754-1760.
[29] Chen Y, Ye W, Zhang Y, et al. High speed BLASTN: anaccelerated MegaBLAST search tool. Nucleic Acids Research,2015, gkv784.
[30] Altschul S F, Gish W, Miller W, et al. Basic local alignment searchtool. Journal of Molecular Biology, 1990, 215(3): 403-410.
[31] Langmead B, Trapnell C, Pop M, et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome.Genome Biol, 2009, 10(3): R25.
[32] Kent W J. BLAT-the BLAST-like alignment tool. GenomeResearch, 2002, 12(4): 656-664.
[33] Lee W-P, Stromberg M P, Ward A, et al. MOSAIK: a hash-basedalgorithm for accurate next-generation sequencing short-readmapping. PloS One, 2014, 9(3): e90581.
[34] Camacho C, Coulouris G, Avagyan V, et al. BLAST+: architectureand applications. BMC Bioinformatics, 2009, 10(1): 1-9[35] Ponstingl H, Ning Z. SMALT-a new mapper for DNA sequencingreads. F1000 Posters, 2010, 1:313.
[36] Zaharia M, Bolosky W J, Curtis K, et al. Faster and more accuratesequence alignment with SNAP. arXiv preprint arXiv:11115572,2011.
[37] Ye Y, Choi J H, Tang H. RAPSearch: a fast protein similaritysearch tool for short reads. BMC Bioinformatics, 2011, 12(1): 1.
[38] Margulies M, Egholm M, Altman W E, et al. Genome sequencingin microfabricated high-density picolitre reactors. Nature, 2005,437(7057): 376-380.
[39] Langmead B, Salzberg S L. Fast gapped-read alignment withBowtie 2. Nature Methods, 2012, 9(4): 357-359.
[40] Li H, Durbin R. Fast and accurate short read alignment withBurrows-Wheeler transform. Bioinformatics, 2009, 25 (14): 1754-1760.
[41] Bhatt A S, Manzo V E, Pedamallu C S, et al. Brief report: in searchof a candidate pathogen for giant cell arteritis: sequencing-basedcharacterization of the giant cell arteritis microbiome. Arthritis &Rheumatology, 2014, 66(7): 1939-1944.
[42] Hercus C. Novoalign. Selangor: Novocraft Technologies, 2012.
[43] Zeitouni B, Boeva V, Janoueix-Lerosey I, et al. SVDetect: a tool toidentify genomic structural variations from paired-end andmate-pair sequencing data. Bioinformatics, 2010, 26 (15): 1895-1896.
[44] Simpson J T, Wong K, Jackman S D, et al. ABySS: a parallelassembler for short read sequence data. Genome Research, 2009,19(6): 1117-1123.
[45] Treangen T J, Sommer D D, Angly F E, et al. Next generationsequence assembly with AMOS. Current Protocols inBioinformatics, 2011, 11(S33): 11.18. 1-11.18. 18.
[46] Morgulis A, Gertz E M, Sch觌ffer A A, et al. A fast and symmetricDUST implementation to mask low-complexity DNA sequences.Journal of Computational Biology, 2006, 13(5): 1028-1040.
[47] Jacob A, Lancaster J, Buhler J, et al. Mercury BLASTP:Accelerating protein sequence alignment. ACM Transactions onReconfigurable Technology and Systems (TRETS), 2008, 1(2): 9.
[48] 李定辰。 基于高通量测序平台的未知病原微生物检测系统[D].北京: 中国人民解放军军事医学科学院, 2016Li D C. Unknown Pathogen Detection System Based onHigh-throughput Sequencing Platform [D]. Beijing: Academy ofMilitary Medical Sciences, 2016.
[49] Kurtz S, Phillippy A, Delcher A L, et al. Versatile and opensoftware for comparing large genomes. Genome Biology, 2004,5(2): R12.
[50] Zerbino D R, Birney E. Velvet: algorithms for de novo short readassembly using de Bruijn graphs. Genome Research, 2008, 18 (5):821-829.
[51] Li R, Li Y, Kristiansen K, et al. SOAP: short oligonucleotidealignment program. Bioinformatics, 2008, 24(5): 713-714.
[52] Quinlan A R, Hall I M. BEDTools: a flexible suite of utilities forcomparing genomic features. Bioinformatics, 2010, 26(6): 841-842.
[53] Huson D H, Mitra S, Ruscheweyh H J, et al. Integrative analysis ofenvironmental sequences using MEGAN4. Genome Research,2011, 21(9): 1552-1560.
[54] Darling A C, Mau B, Blattner F R, et al. Mauve: multiple alignmentof conserved genomic sequence with rearrangements. GenomeResearch, 2004, 14(7): 1394-1403.
[55] Thorvaldsdóttir H, Robinson J T, Mesirov J P. Integrative GenomicsViewer (IGV): high-performance genomics data visualization andexploration. Briefings in Bioinformatics, 2013, 14(2): 178-192.
[56] Krzywinski M, Schein J, Birol I, et al. Circos: an informationaesthetic for comparative genomics. Genome Research, 2009,19(9): 1639-1645.
[57]叶福强。胆总管结石患者胆道和阿尔茨海默症小鼠肠道的宏基因组学研究[D].北京:中国人民解放军军事医学科学院, 2016Ye F Q. Metagenomic Studies on The Biliary Microbiota ofPatients With Choledocholithiasis and The Gut Microbiota of MiceWith Alzheimer's Disease [D]. Beijing: Academy of MilitaryMedical Sciences, 2016.
[58] Wang H, Chan S-H, Cheung J, et al. MICA: A fast short-readaligner that takes full advantage of Intel Many Integrated CoreArchitecture (MIC)。 arXiv preprint arXiv, 2014: 14024876.