基于NGS的微生物检测数据分析技术探究(3)
来源:生物化学与生物物理进展 作者:周子寒;彭绍亮;伯晓晨
发布于:2017-06-15 共15890字
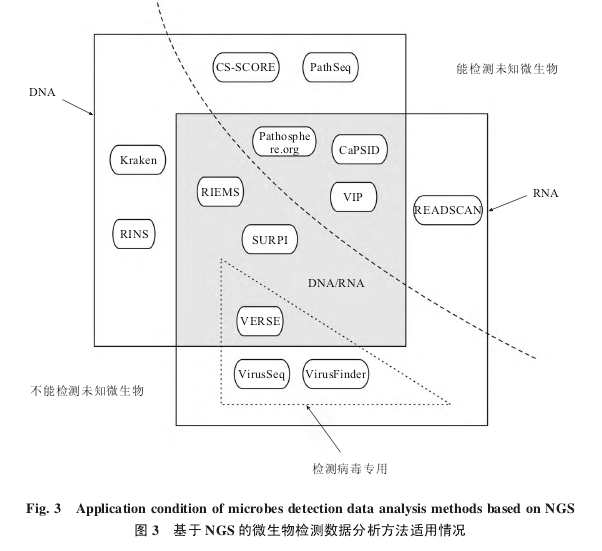
a.在计算速度方面,以 CS-SCORE 的 cs-score值计算法、Kaken 的精确 k-mer 匹配法、RINS 的基于先验知识的加速法为代表的比对方法优化(环节B),对核心比对流程进行了算法上的改进,使其运行速度大大增加。b.在计算精度上,以PathSeq为代表的多次循环比对法,在最大限度上规避了微生物参考基因组MRG 不完善导致的精度下降。c.在计算资源方面,Kraken 的精简数据库法和 CS-SCORE 的基于 cs-score 值 计算法使运行内存大大降低。d.体系结构方面,目前基于 NGS的数据处理方法都是基 于 Linux 开 发 , 但 是Pathosphere.org工作流程为上传数据到云服务器,在服务器上完成计算产生结果报告,对于运行平台没有要求。e.在可扩展性方面,以 VirusSeq 和Kraken 为代表的方法都提供了多线程处理选项,能够有效增加运行效率。f.在功能方面,以Virusseq、CaPSID 为代表的病毒结合位点分析以及SNP 分析等都属于对于后续功能的完善。尽管最近几年都有新的检测方法被提出,但是早期的检测方法由于其他方面的优势,在不同的应用场景下,可与新的方法互补使用。例如在高精度的病毒转录组数据检测中,VirusFinder 的效果较 Kraken 更好。这里总结基于NGS 的微生物检测流水线的适用情况,如图 3 所示。
在上述基于NGS 的微生物检测数据分析方法中,一类方法针对检测未知微生物进行序列比对精度上的优化,其处理方式为从样本文件中逐步清除人类基因序列,最后剩余包含已知和未知生物基因序列,典型方法包括 Pathseq、CS-SCORE 等,另一部分则侧重快速检测已知微生物,其处理方式为直接与微生物基因组进行比对,但不足之处在于,受限于所选取的参考基因组,无法检出未知或罕见的微生物种类,典型算法包括RINS、Kraken 等。在输入数据方面,一些方法只能处理DNA 序列文件,如Kraken,有的方法只能处理 RNA 序列文件,如READSCAN,大部分方法两者都能处理,包括VERSE、SURPI 等。在检测应用领域方面,VERSE、VirusSeq、VirusFinder作为专门检测病毒基因序列的方法,采用的参考数据库只包含病毒基因序列。鉴于病毒参考基因组远小于细菌参考基因组,此类软件分析更为快速便捷。
下文我们将对不同类型的检测方法做进一步的介绍:
a.“基础型”检测方法“基础型”检测方法。
采用的数据处理策略为A-C-D-E,包含 PathSeq 和 RINS.属于基于 NGS 的微生物检测方法刚起步的阶段。其中 PathSeq 针对微生物检测的精度问题进行了优化,RINS 针对微生物检测的速度问题进行了优化。
PathSeq是 2011 年麻省理工学院和哈佛大学联合研究所的 Kostic[15]提出的基于 Amazon 云平 台[41]的微生物检测数据分析方法。PathSeq 提出了多次过滤法,用以提高微生物检测的精度:在进行将输入样本宏基因组数据与参考基因组做序列比对(环节C)时,采用MAQ、MegaBlast、BlastN 对其中包含的人类基因序列进行多次循环过滤,充分去除人类基因序列。PathSeq 的缺点为由于多次进行序列比对,其运行速度较慢。PathSeq 适用于对检测速度要求不高,精度要求很高的案例,譬如查找大规模疫情中的新型细菌病毒。Bhatt 等[42]在 2014 年将该数据分析方法用于巨细胞动脉炎(giant cellarteritis, GCA) 病原体检测,样本来自于 17 名GCA 患者,采用 PathSeq 将其中的人类 DNA 序列去除,然后对剩余序列进行了聚类分析。该实验中PathSeq 对人类 DNA 序列的清除率为 100%,起到了良好的效果。
RINS(rapid identification of nonhuman sequences)是2012 年 Bhaduri[16]提出的基于先验知识的微生物检测数据分析方法。RINS 提出了基于先验知识的加速法,用以提高微生物检测的速度。其处理流程与一般微生物检测数据分析方法不同,首先针对微生物的物理化学性质(形态学观测等方法),对其种属进行假设,根据假设结果选择部分微生物参考基因组MRG 与样本数据进行序列比对,确认其中是否包含已知微生物序列。由于选择的微生物参考基因组远远小于人类参考基因组HRG,先验知识法可以达到减小运算量,加快运行速度的效果。其比对过程(环节 C)采用的软件为 BLAST.但如果参考微生物基因组选择错误,则需要重新选择参考基因组,造成计算冗余,增加运算时间。RINS 数据分析方法适用于快速检测常见症状的、潜伏期短的突发 疫 情 中 的 微 生 物 .Bhaduri 等 利 用 RINS 对CA-HPV-10 前列腺癌细胞序列数据 130 万个长度为100 bp 的 reads 进行检测用时 2 h,而 PathSeq在处理相似数据量的问题用时约为13 h,说明RINS 在检测速度上的提升效果显着。