摘 要: 材料基因工程是近年来材料领域兴起的前沿技术,其基本理念是融合材料高通量计算、高通量实验和数据技术加速新材料的设计和研发.分子筛作为一种重要的化工材料,因其良好的热稳定性、较高的比表面积、独特的孔道结构及可调变的元素组成和酸性,在气体吸附、分离、异相催化和离子交换等工业领域应用广泛.近年来,融合高通量计算、高通量实验和数据库技术的材料基因工程技术正逐步应用于分子筛研发等领域:高通量计算能够从理论上预测并筛选出具有优异性能的分子筛合成目标、高通量实验显着提升了分子筛材料合成与表征的效率、数据库技术则为未来挖掘分子筛材料的合成规律与构效关系奠定了数据基础.本文主要从这3个方面阐述材料基因工程技术在分子筛材料研发领域的应用及进展,总结以功能为导向、定向设计和构筑分子筛材料所面临的机遇与挑战,并对材料基因工程技术在分子筛领域的前景进行了展望.
关键词: 分子筛; 高通量计算; 高通量实验; 数据库; 材料基因工程;
Abstract: Materials genome engineering integrates high-throughput computation,high-throughput experimentation and data technology to accelerate the design and development of new materials. As an important family of microporous materials,zeolites have been widely used in gas adsorption,separation,heterogeneous catalysis and ion exchange because of their excellent thermal stabilities,high specific surface areas,unique welldefined porous structures,and tunable framework compositions and acidities. To facilitate the development of novel zeolitic materials,materials genome engineering technology has recently been applied in many different areas in zeolite research. In this review,we will discuss about the recent progress in high-throughput computation,high-throughput experimentation and database of zeolites. Our perspectives on the future development of materials genome engineering of zeolites will be provided in the last section.
Keyword: Zeolites; High-throughput computation; High-throughput experimentation; Database; Materials genome engineering;

传统意义上的沸石分子筛是指以硅氧四面体和铝氧四面体为基本结构单元、通过桥氧共顶点相互连接构成的一类具有笼形或孔道结构的硅铝酸盐晶体.自1948年首次人工合成丝光沸石[1],沸石分子筛材料的合成与应用得到迅速发展[2,3,4,5,6,7,8].分子筛多孔晶体材料因其特殊的孔道结构,被广泛用于吸附分离、催化反应、离子交换等领域,在石油化工、精细化工及功能材料中有重要应用[4,9].然而,目前仅有十余种分子筛结构类型得到大规模工业应用,因此根据性能需求定向合成具有特定孔道结构的新型分子筛材料对拓展其应用具有重要意义.由于影响分子筛合成与性能的因素较多,分子筛的催化反应机理、成核与晶化机制、结构与性能间的构效关系尚未清晰,因此,分子筛材料的定向设计与合成仍充满挑战.
与传统化学合成不同,分子工程学是以功能为导向,对结构进行设计与剪裁,并在计算机辅助下实现对特定功能和结构材料的定向合成、制备和组装.吉林大学徐如人和于吉红课题组[10,11,12,13,14,15,16,17,18,19,20,21,22]自2000年起就致力于分子筛材料的分子工程学研究,提出从功能需求出发,在计算机技术的辅助下,设计新颖的多孔晶体结构、最终实现其定向合成的分子筛研发思路[23](图1),这种理念与后来美国政府提出的材料基因组计划相似.近期,我国也提出了材料基因工程计划,旨在融合高通量计算、高通量实验和数据库三大技术,探索“理论预测-实验验证-应用示范”这一新材料研发新模式,大幅缩短新材料的研发周期,降低研发成本.除了在生物制药、有机合成、催化剂合成等领域的广泛应用[24],材料基因工程技术近年来在分子筛研发领域也取得了长足发展[25,26,27,28,29,30,31,32,33,34,35,36,37,38,39,40,41,42].
本文重点介绍近年来高通量计算、高通量实验和数据库三大技术在分子筛材料研发领域的应用与进展,聚焦三者在分子筛研发领域所面临的挑战,并对材料基因工程技术在分子筛材料领域的前景进行了展望.
Fig.1 Rational synthesis of zeolitic materials with desired functionalities and structures[23]
SDA:structure directing agent.Copyright 2010,American Chemical Society.
![Fig.1 Rational synthesis of zeolitic materials with desired functionalities and structures[23]](http://www.xueshut.com/uploads/allimg/210304/36-210304094559515.jpg)
1 、高通量计算在分子筛领域的应用
高通量计算是材料基因工程的基础,是利用并行、自动化的流程管理,系统计算材料的结构、组成、性能等属性,并对材料进行大规模理论预测与筛选,从海量备选材料中确定合成目标,进而指导新材料实验合成的理论方法.与传统的高性能计算相比,高通量计算通过并行、智能的任务流程管理,实现对复杂材料体系的大规模无监督计算,显着提高了对材料结构与性质空间搜索的效率.在分子筛领域,高通量计算主要用于分子筛材料的结构预测、性质筛选、模板剂预测等方面.
1.1、 结构预测
分子筛材料的结构预测是利用计算机方法搭建理论上合理的假想分子筛骨架结构模型,为新型分子筛材料的定向合成提供靶向目标.目前国际上主流的结构预测方法包括模拟退火法[43,44,45]、原子禁区法[46,47]及对称约束键连搜索法[48]等,已预测出数百万种假想分子筛骨架结构[49,50,51].然而,由于分子筛的骨架结构极其复杂,实际可能存在的分子筛骨架结构类型远远超出现有理论方法的预测能力,亟需开发更为高效的分子筛结构枚举方法,以实现对假想分子筛骨架的高效预测.
面对这一挑战,Li等[52]借鉴生物基因组思想提出了无机晶体结构基因概念,成功枚举出84292种假想ABC-6分子筛结构(图2).他们根据ABC-6分子筛的6元环堆积特点,设计出用计算机方法将三维晶体结构表达为一维的“基因”编码,通过枚举可能存在的基因编码,实现对ABC-6分子筛结构类型的高通量枚举.通过分析这些结构的编码特征,进一步预测出1127种最有可能在实验中被合成的结构类型,其中部分预测结构已在后来的合成实验中得到证实[53,54].这种“基因”编码方法为分子筛材料的高通量结构预测提供了一种全新的研究思路.Li等[55]又从枚举出的ABC-6结构出发,构建了相应的磷酸铝分子筛结构模型,通过引入基于距离的简单数学模型,实现对八万余种假想磷酸铝分子筛中杂原子取代必要性的高通量预测,预测结果具有较高的准确度.与传统用于预测杂原子取代分子筛的计算方法相比,这种基于距离的新方法不需要构建复杂的杂原子取代模型,仅需对纯磷酸铝结构模型进行评估,大幅降低了高通量计算的复杂度,为假想结构的实验合成提供了重要的理论依据.
Fig.2 Two ABC-6 zeolite topologies predicted by a genomic approach realized in experiment[52]
Copyright 2015,Springer Nature.
![Fig.2 Two ABC-6 zeolite topologies predicted by a genomic approach realized in experiment[52]](http://www.xueshut.com/uploads/allimg/210304/36-210304094334F6.jpg)
1.2 、性质筛选
高通量计算预测出的海量假想分子筛结构模型需进一步计算其物理化学性质,以筛选出具有优异性能的假想结构模型作为实验合成目标.高通量计算方法已被广泛用于计算分子筛的物理化学性质,如吸附性能、分离性能及催化性能等[56,57,58].为实现对海量结构的快速评价,传统方法通常采用计算量和精度均较低的力场计算方法,往往难以得到与实验相符的满意结果.Gaillac等[59]为分子筛性能的高通量精确预测提供了一种新思路.他们从590811种假想分子筛结构出发,采用传统分子力学方法进行了力学性质计算.与高精度密度泛函(DFT)计算相比,传统分子力学计算的精度较低,尤其对于各项异性的力学性质(如泊松比等),分子力学计算的偏差较大.他们利用梯度提升回归(GBR)的机器学习方法,利用已有的数百种分子筛材料的DFT数据,训练出预测分子筛材料机械性能的数学模型,预测结果的准确性远远高于传统的分子力学方法.同时,机器学习模型揭示出分子筛结构属性(如孔体积、Si—O键长标准差、Si—O—Si键角标准差等)与其机械性能的关联性,展现了结合DFT计算的机器学习方法在分子筛高通量性质预测中的潜力.Gu等[60]通过以DFT数据为基础的特征学习方法,实现对与假想分子筛氮气吸附性能的高通量筛选.他们采用梯度提升、k-最近邻、多重线性回归等机器学习方法,从有限的DFT计算数据出发,提取出3个最具代表性的结构描述符(包括有效孔体积、最大容纳球直径和局部几何变形),预测了200181种假想分子筛骨架的氮分子结合能,并从中筛选出24441种有利于氮气吸附的分子筛骨架(图3).这些机器学习方法的引入大幅降低了分子筛性质预测的计算量,有效提升了高通量性质筛选的效率.
Fig.3 Binding energies(EbPL,1 kcal=4.19 k J) of 200181 hypothetical zeolites(A) and 100 known zeolites(B)against diameters(1?=0.1 nm) of the largest included spheres(PLD)[60]
Copyright 2020,American Chemical Society.
![Fig.3 Binding energies(EbPL,1 kcal=4.19 k J) of 200181 hypothetical zeolites(A) and 100 known zeolites(B)against diameters(1?=0.1 nm) of the largest included spheres(PLD)[60]](http://www.xueshut.com/uploads/allimg/210304/36-210304094255Q9.jpg)
1.3 、有机结构导向剂预测
有机结构导向剂(OSDA)是实验合成特定结构分子筛材料的重要因素[61].利用高通量计算方法,预测能够合成目标结构的OSDA,对实现功能导向分子筛的定向合成具有关键作用.
传统的OSDA预测方法需要人为指定少量备选有机胺分子,通过计算备选分子与目标骨架的相互作用能来评价备选分子对目标分子筛骨架的结构导向能力[62,63,64].如Shi等[65]利用分子动力学方法研究了21种用于合成STW分子筛的咪唑类OSDA分子,发现与STW骨架具有较强相互作用的咪唑分子具有氮原子带负电及分子偶极矩较小等特点.基于此,他们设计了7种新型咪唑类OSDA分子,均展现出较强的与STW的结合作用.
为突破传统依赖人为选择备选OSDA的半经验方法进而实现对海量OSDA分子的高通量预测,Deem等[66]开发了一种从头计算方法实现了对海量OSDA模型的高通量自动搭建和评价.他们从有机试剂库出发,根据有机分子在分子筛合成条件下的稳定性、刚性、体积、几何相容性及与目标分子筛的相互作用等对OSDA分子进行评价,并根据已知的化学反应库自动构建新的OSDA分子,实现对现有OSDA数据库的快速扩展.他们以AEI,ITE和STF 3种拓扑结构为例,从12838种有机化合物中为每种分子筛结构筛选出最优的OSDA分子.最近,他们又采用这种方法实现了BEA,STW等分子筛OSDA分子的高通量预测[67].与传统依赖人为筛选的半经验预测方法相比,这些自动化的高通量计算方法大大提升了OSDA分子的预测效率,为假想分子筛骨架的合成提供了重要的理论指导.
2、 高通量实验在分子筛领域的应用
高通量实验是从组合化学概念上发展起来的实验制备与表征技术,是通过设计特定的实验装置与技术,在有限时间内平行合成或表征传统方法难以获得的海量样品.分子筛材料的反应条件复杂,高通量实验通过平行调变多种影响分子筛合成的实验因素,可以更高效地筛选分子筛合成的最佳反应条件,提高发现新型分子筛材料的效率,缩短分子筛材料的研发周期.
2.1、 高通量合成
1998年,Akporiaye等[68]设计了10×10的100孔组合釜研究分子筛合成的相图,从而筛选反应条件,首次将高通量合成方法应用于分子筛领域.自此,多种分子筛材料通过高通量实验方法被研发出来,如在64孔组合釜下合成出的二维层状磷酸铝[69]、用21孔反应器合成的ZSM-5分子筛膜[70]、用15孔反应器合成出的锗酸盐分子筛ITQ-30[71]等.
在锗酸盐分子筛合成体系中,Jiang等[72]选择7种OSDA分子分别设计了33×5个高通量实验(图4),探索了多种参数对合成产物的影响.他们在945个反应中获得了395组分子筛晶体产物,包含了Beta,ITQ-7,ITQ-17,ITQ-15,ITQ-21,ITQ-37,ITQ-43和ITQ-44等共8种分子筛晶体类型,其中ITQ-15,ITQ-37,ITQ-44和ITQ-43有超大孔硅锗分子筛结构.这种对分子筛合成条件的系统平行调变体现了高通量实验的优势,高效探索了OSDA及多种合成参数对产物结构的影响,展现了高通量实验在筛选合成条件及研发新型分子筛材料的巨大潜力.
2.2 、高通量表征
高通量表征是基于特定的表征设备或方法,在有限时间内对大量样品进行快速、平行、自动化的表征,大幅度提高了材料表征的效率.其中,X射线衍射(XRD)技术已被用于大量分子筛材料的快速高通量表征[73,74].如Klein等[75]将单晶Si片引入100孔反应装置,首次将高通量实验与XRD组合装置结合应用于分子筛研究,对25000种分子筛晶体样品进行了高通量XRD表征.除XRD外,显微X射线荧光(MXRF)[76]、傅里叶转换红外光谱(FTIR)[77]、定量多流质谱仪筛选(MSMSS)[78]等也被应用于分子筛材料的高通量表征中.Cichocka等[79]开发了连续旋转电子衍射(c RED)方法,实现了对分子筛晶体样品的高通量数据收集.他们为电子显微镜设计了特定的计算机程序,利用深层卷积神经网络技术,根据衍射数据自动识别需要表征分子筛晶体.这种高通量方法的开发大幅简化了传统c RED数据收集过程中所需的手工操作,降低了人为错误的风险,显着提高了分子筛材料的表征效率.
Fig.4 Crystallization field diagram for all 7 OSDAs at n(H2O)/n(Si+Ge)=3[72]
Copyright 2011,American Chemical Society.
![Fig.4 Crystallization field diagram for all 7 OSDAs at n(H2O)/n(Si+Ge)=3[72]](http://www.xueshut.com/uploads/allimg/210304/36-210304094221127.jpg)
3 、数据库技术在分子筛领域的应用
高通量计算与高通量实验产生了大量的理论与实验数据,这些数据形成了材料研发数据库,不仅为研究人员提供了数据共享、查询与检索的平台,更重要的是为后续的数据挖掘研究提供了丰富的源数据.
3.1 、数据库构建
分子筛数据库包含结构数据库和合成数据库两大类.结构数据库收录已知或假想分子筛材料的各种结构数据信息,为合成实验、产物表征及理论计算提供了基础数据.其中,国际分子筛协会结构委员会建立的分子筛结构数据库收录了迄今为止发现的250余种分子筛骨架类型,提供了包括拓扑结构、次级结构单元、孔道性质、孔道体积、骨架密度以及参考文献等信息的浏览与检索,是分子筛领域最重要的结构数据库之一[80].作为分子筛材料的重要分支,开放骨架磷酸铝因其独特的物理化学性质一直备受关注.Zheng等[81,82]从公开文献中收集了312个开放骨架磷酸铝晶体结构,构建了磷酸铝结构数据库.同时,高通量计算产生了大量假想分子筛的结构与性质数据,组成了分子筛结构的理论数据库.目前国际上主流的理论数据库包括Deem等[83]基于平行回火原理构建的数据库和Treacy等[48]基于对称限制键连搜索法构建的数据库.在此基础上,Li等[84]基于分子筛材料基因编码和平行回火原理等方法建立了分子筛假想结构数据库,收录了数十万种理论可行的假想分子筛结构数据.与其它假想结构数据相比,该数据库包含更多理论可行的分子筛结构数据.
与结构数据库相比,分子筛合成数据库的发展比较缓慢.自上世纪90年代开始,吉林大学徐如人和于吉红课题组致力于建立分子筛材料的合成数据库.2009年,他们发布了分子筛领域首个合成数据库—ZEOBANK[85,86],该数据库收录了多种磷酸铝分子筛的合成信息,包括合成配比、反应原料、晶化温度、晶化时间、有机模板剂、产物结构信息、孔道维数、窗口环数、骨架密度等.为更高效地收集分子筛材料的合成数据,Jensen等[87]开发了一种可以查找、提取和格式化分子筛合成数据的自动数据提取方法,从7万份论文内的合成表中自动提取与分子筛合成相关的凝胶成分数据,并确定了合成变量和合成产物之间的对应趋势.这些数据有助于加深人们对分子筛材料结构化学的认识,为探索合成与结构之间的关联提供了重要的数据基础.
3.2 、数据挖掘技术
数据挖掘是从大量数据中提取隐含信息的计算机方法.目前分子筛领域的数据挖掘方法主要用于预测特定分子筛的合成条件与构效关系,具体方法包括决策树、神经网络、支持向量机、聚类算法及主成分分析等.
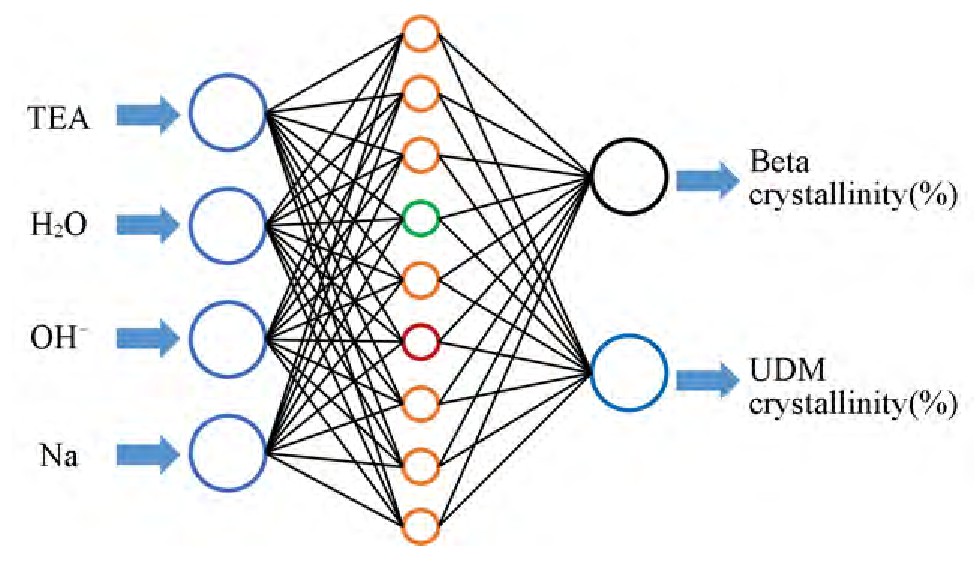
在合成条件预测方面,2003年,Liu等[88]采用决策树等数据挖掘方法成功预测了Al PO-5分子筛的合成条件.随后,Moliner等[89]使用人工神经网络研究了Beta分子筛和UDM分子筛的合成条件.通过构建模拟人脑的神经网络模型(图5),他们将不同反应凝胶组成比映射为Beta分子筛和UDM分子筛的结晶度,进一步找到合成Beta分子筛的最优凝胶组成.Li等[90]利用支持向量机研究了含(6,12)环的磷酸铝分子筛的合成因素.他们将磷酸铝分子筛合成反应数据库中的数据进行整理,计算了2047种不同的合成参数组合,得到了能够准确分类含(6,12)环磷酸铝分子筛的合成参数组合.这些数据挖掘方法能较准确地预测不同反应组成所得到的产物,避免了反复试验所消耗的原料与时间成本,提高了分子筛材料实验合成的效率.Daeyaert等[91]结合从头设计,从4781种备选OSDA分子出发,构建了人工神经网络模型,从中筛选出469种用于导向Beta分子筛的新型OSDA,其与Beta分子筛骨架的结合作用优于现有Beta分子筛的OSDA分子.这些研究展现出人工神经网络代替需要高昂计算量的传统计算化学方法的潜力,为指导分子筛材料的定向合成、理解分子筛材料的构效关系提供了新的研究思路.
Fig.5 Artificial neural network
4、 总结与展望
材料基因工程技术对定向设计具有特定功能的分子筛材料具有重要意义.借助高通量计算、高通量实验与数据库技术的深度融合,有望解决分子筛领域复杂的反应机制、晶化机理、构效关系等问题.未来,材料基因工程技术在以下领域的发展值得期待:
高通量计算.由于分子筛材料的骨架结构复杂,结构预测与性质筛选的计算量巨大,因此需要开发更为廉价易行的计算方法和模型.此外,由于理论模型与实验条件的差异,高通量计算对实验的指导作用仍十分有限[92,93].在理论计算与实验中建立有效的交互机制,用实验数据修正计算模型,将有助于提高理论计算的精度,缩小理论计算与实验数据的差距,从而增强理论计算对实验的指导作用.
高通量实验.分子筛合成过程中同时涉及固态和液态试剂、变化的温度与压力,由于装置和实验条件的限制,目前分子筛材料的高通量实验合成仍充满挑战.此外,合成实验中数据的自动记录及一体化表征仍未实现,在未来需要设计更为高效、自动化的多通道合成与反应装置,实现合成及表征的自动化记录与调度,从本质上提升分子筛材料合成的实验效率.
数据库.尽管分子筛领域已建立比较完善的结构数据库,分子筛实验合成数据库与性质数据库的建设仍处在起步阶段.高通量实验装置与技术的改进将有助于提高实验数据的自动记录与管理.同时,以自然语言识别为代表的机器学习技术有望用于提取海量文献中记录的实验数据,快速构建分子筛材料的实验数据库.在这个方面,有监督学习与无监督学习的相互结合对开发分子筛领域的自然语言识别模型至关重要.
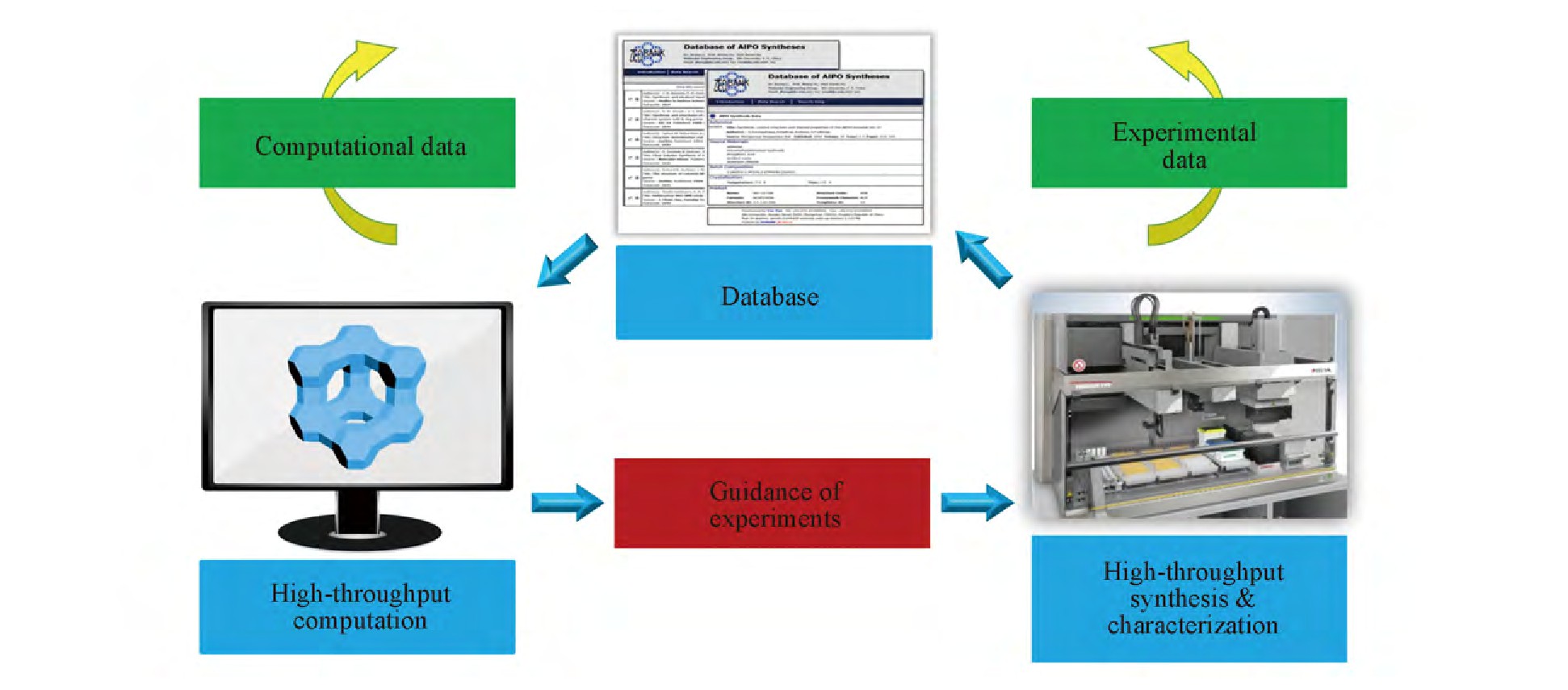
总之,高通量计算、高通量实验与数据库三大技术在分子筛材料领域已取得长足进步,但三者之间的联系仍需加强.未来,需强化高通量计算对实验的指导作用,同时反馈实验数据为计算模型进行修正.此外,需构建并完善自动化的数据库平台,收集整合高通量计算与实验产生的数据,进一步通过数据挖掘指导分子筛材料的计算与实验设计(图6).这些技术的实现将极大促进新型分子筛材料的研发,拓展其在传统化工与新兴交叉领域中的应用[94].
Fig.6 Materials genome engineering of zeolites
参考文献
[1] Barrer R.,J. Chem. Soc.,1948,2,127—132
[2] Yu J. H.,Xu R. R.,J. Mater. Chem.,2008,18(34),4021—4030
[3] Yu J. H.,Xiao F. S.,Corma A.,Chin. J. Catal.,2015,36(6),787—788
[4] Yi T.,Li J. W.,Zhang Y. B.,Yang X. G.,Chem. Res. Chinese Universities,2018,34(4),661—664
[5] Weckhuysen B.,Yu J. H.,Chem. Soc. Rev.,2015,44(20),7022—7024
[6] Song X. W.,Li Y.,Gan L.,Wang Z. P.,Yu J. H.,Xu R. R.,Angew. Chem. Int. Ed.,2009,48(2),314—317
[7] Che Y. P.,Wang Y.,You T. T.,Chang H. Q.,Yin P. G.,Zhai J.,Chem. Res. Chinese Universities,2018,34(5),772—780
[8] Bai R. S.,Song Y.,Li Y.,Yu J. H.,Trends Chem.,2019,1(6),601—611
[9] Xu R. R.,Pang W. Q.,Yu J. H.,Huo Q. S.,Chen J. S.,Chemistry of Zeolites and Related Porous Materials,Science Press,Beijing,2004,13—24(徐如人,庞文琴,于吉红,霍启升,陈接胜.分子筛与多孔材料化学,北京:科学出版社,2004,13—24)
[10] Wang K. X.,Li J. Y.,Yu J. H.,Xu R. R.,Acta Chim. Sin.,2000,58(12),1626—1630
[11] Yu J. H.,Xu R. R.,Chem. Soc. Rev.,2006,35(7),593—604
[12] Yu J. H.,Xu R. R.,Stud. Surf. Sci. Catal.,2004,154(A),1—13
[13] Yan W. F.,Yu J. H.,Shi Z.,Miao P.,Wang K. X.,Wang Y.,Xu R. R.,Micropor. Mesopor. Mater.,2001,50(2/3),151—158
[14] Song Y.,Yu J. H.,Li Y.,Zhang Y.,Zhang M.,Xu R. R.,Stud. Surf. Sci. Catal.,2004,154(A),1028—1034
[15] Song Y.,Yu J. H.,Li Y.,Zhang M.,Xu R. R.,Eur. J. Inorg. Chem.,2004,(18),3718—3723
[16] Song Y.,Yu J. H.,Li G. H.,Li Y.,Wang Y.,Xu R. R.,Chem. Commun.,2002,2(16),1720—1721
[17] Lu J. R.,Li Y.,Yu J. H.,Lu Y.,Acta Phys. Chim. Sin.,2013,29(8),1661—1665
[18] Li J. Y.,Yu J. H.,Xu R. R.,Chin. J. Inorg. Chem.,2004,20(1),1—16
[19] Li J. Y.,Yu J. H.,Sun J. G.,Dong X. C,Li Y.,Wang Z. P.,Wang S. X.,Xu R. R.,Stud. Surf. Sci. Catal.,2007,170,168—176
[20] Huo W. F.,Li Y.,Lu J. R.,Yu J. H.,Xu R. R.,Li J.,Acta Phys. Chim. Sin.,2012,28(3),536—540
[21] Huo W. F.,Gao N.,Yan Y.,Li J. Y.,Yu J. H.,Xu R. R.,Acta Phys. Chim. Sin.,2011,27(9),2111—2117
[22] Yu J. H.,Xu R. R.,Acc. Chem. Res.,2003,36(7),481—490
[23] Yu J. H.,Xu R. R.,Acc. Chem. Res.,2010,43(9),1195—1204
[24] De Pablo J. J.,Jones B.,Lind C. L.,Ozolins V.,Ramirez A. P.,Curr. Opin. Solid State Mater. Sci.,2014,18(2),99—117
[25] Cheng T.,Xu J.,Li X.,Li Y.,Zhang B.,Yan W. F.,Yu J. H.,Sun H.,Deng F.,Xu R. R.,Micropor. Mesopor. Mater.,2012,152,190—207
[26] Li J. Y.,Corma A.,Yu J. H.,Chem. Soc. Rev.,2015,44(20),7112—7127
[27] Li J. Y.,Wang D.,Wang Y.,Du H. B.,Deng L.,Hou X. L.,Yu J. H.,Chin. Sci. Bull.,2015,60(21),1987—1991(李激扬,王栋,王远杜红宾,邓亮,侯雪龙,于吉红.科学通报,2015,60(21),1987—1991)
[28] Li Y.,Cao H. X.,Yu J. H.,ACS Nano,2018,12(5),4096—4104
[29] Li Y.,Yu J. H.,Chem. Rev.,2014,114(14),7268—7316
[30] Li Y.,Yu J. H.,Chem. Sci.,2016,7(6),3472—3481
[31] Li Y.,Yu J. H.,Xu R. R.,Angew. Chem. Int. Ed.,2013,52(6),1673—1677
[32] Lu H. Y.,Xu J.,Gao P.,Yan W. F.,Deng F.,Xu R. R.,Micropor. Mesopor. Mater.,2015,208,105—112
[33] Stock N.,Micropor. Mesopor. Mater.,2010,129(3),287—295
[34] Tang Y. Q.,Xu R. R.,Guo G. L.,Yu J. H.,Wang Y.,Lai L. H.,Wu K.,Chem. Bull.,2011,74(11),970—982(唐有祺,徐如人,郭国霖,于吉红,王远,来鲁华,吴凯.化学通报,2011,74(11),970—982)
[35] Tong X. Q.,Xu J.,Li X.,Li Y.,Yan W. F.,Yu J. H.,Deng F.,Sun H.,Xu R. R.,Micropor. Mesopor. Mater.,2013,176,112—122
[36] Tong X. Q.,Xu J.,Wang C.,Lu H. Y.,Huang P.,Yan W. F.,Yu J. H.,Deng F.,Xu R. R.,Micropor. Mesopor. Mater.,2012,155,153—166
[37] Tong X. Q.,Xu J.,Xin L.,Huang P.,Lu H. Y.,Wang C.,Yan W. F.,Yu J. H.,Deng F.,Sun H.,Xu R. R.,Micropor. Mesopor.Mater.,2012,164,56—66
[38] Wang Z. P.,Yu J. H.,Xu R. R.,Chem. Soc. Rev.,2012,41(5),1729—1741
[39] Yan W. F.,Song X. W.,Xu R. R.,Micropor. Mesopor. Mater.,2009,123(1—3),50—62
[40] Yan W. F.,Xin L.,Olman V.,Yu J. H.,Wang Y.,Xu Y.,Xu R. R.,Micropor. Mesopor. Mater.,2010,131(1—3),148—161
[41] Yu J. H.,Qiu S. L.,Li J. Y.,Xu R. R.,Chem. Bull.,2011,74(11),983—988
[42] Zhang B.,Xu J.,Fan F. T.,Guo Q.,Tong X. Q.,Yan W. F.,Yu J. H.,Deng F.,Li C.,Xu R. R.,Micropor. Mesopor. Mater.,2012,147(1),212—221
[43] Falcioni M.,Deem M.,Phys. Rev. E,2000,61(5),5948—5952
[44] Chen L. G.,Deem M.,J. Chem. Inf. Comput. Sci.,2001,41(4),950—957
[45] Chen L. G.,Deem M.,Molec. Phys.,2002,100(13),2175—2181
[46] Li Y.,Yu J. H.,Liu D. H.,Yan W. F.,Xu R. R.,Xu Y.,Chem. Mater.,2003,15(14),2780—2785
[47] Li Y.,Yu J. H.,Wang Z. P.,Zhang J. N.,Guo M.,Xu R. R.,Chem. Mater.,2005,17(17),4399—4405
[48] Treacy M.,Rivin I.,Balkovsky E.,Randall K.,Foster M.,Micropor. Mesopor. Mater.,2004,74(1—3),121—132
[49] Li Y.,Yu J. H.,Xu R. R.,Baerlocher C.,McCusker L.,Angew. Chem. Int. Ed.,2008,47(23),4401—4405
[50] Zhou D.,Xu J.,Yu J. H.,Chen L.,Deng F.,Xu R. R.,J. Phys.Chem. B,2006,110(5),2131—213
[51] Lu J. R.,Shi C.,Li Y.,Yu J. H.,Chin. Chem. Lett.,2017,28(7),1365—1368
[52] Li Y.,Li X.,Liu J. C.,Duan F. Z.,Yu J. H.,Nat. Commun.,2015,6,8328
[53] Turrina A.,Garcia R.,Cox P.,Casci J.,Wright P.,Chem. Mater.,2016,28(14),4998—5012
[54] Turrina A.,Garcia R.,Watts A.,Greer H.,Bradley J.,Zhou W.,Cox P.,Shannon M.,Mayoral A.,Casci J.,Wright P.,Chem.Mater.,2017,29(5),2180—2190
[55] Li L.,Slater B.,Yan Y.,Wang C. M.,Li Y.,Yu J. H.,J. Phys. Chem. Lett.,2019,10(6),1411—1415
[56] Deka R.,Vetrivel R.,Comb. Chem. High Throughput Screening,2003,6(1),1—9
[57] Lee Y.,Barthel S.,Dlotko P.,Moosavi S.,Hess K.,Smit B.,J. Chem. Theory Comput.,2018,14(8),4427—4437
[58] Yan Z. G.,Tang S.,Zhou X. M.,Yang L.,Xiao X. Q.,Chen H. Y.,Qin Y. H.,Sun W.,Chin. J. Chem. Eng.,2019,27(1),174—181
[59] Gaillac R.,Chibani S.,Coudert F.,Chem. Mater.,2020,32(6),2653—2663
[60] Gu Y. M.,Liu Z. T.,Yu C. Z.,Gu X.,Xu L. L.,Gao Y.,Ma J.,J. Phys. Chem. C,2020,124(17),9314—9328
[61] Moliner M.,Román-Leshkov Y.,Corma A.,Acc. Chem. Res.,2019,52(10),2971—2980
[62] Song Y.,Li J. Y.,Yu J. H.,Wang K. X.,Xu R. R.,Top. Catal.,2005,35(1/2),3—8
[63] Li J. Y.,Yu J. H.,Xu R. R.,Micropor. Mesopor. Mater.,2007,101(3),406—412
[64] Li J. Y.,Li L.,Liang J.,Chen P.,Yu J. H.,Xu Y.,Xu R. R.,Cryst. Growth Des.,2008,8(7),2318—2323
[65] Shi C.,Li L.,Yang L.,Li Y.,Chin. Chem. Lett.,2020,31(7),1951—1955
[66] Pophale R.,Daeyaert F.,Deem M.,J. Mater. Chem. A,2013,1(23),6750—6760
[67] Daeyaert F.,Deem M.,RSC Adv.,2019,9(71),41934—41942
[68] Akporiaye D.,Dahl I.,Karlsson A.,Wendelbo R.,Angew. Chem. Int. Ed.,1998,37(5),609—611
[69] Song Y.,Yu J. H.,Li Y.,Li G. H.,Xu R. R.,Angew. Chem. Int. Ed.,2004,43(18),2399—2402
[70] Lai R.,Kang B. S.,Gavalas G.,Angew. Chem. Int. Ed.,2001,40(2),408—411
[71] Corma A.,Diaz-Cabanas M.,Moliner M.,Martinez C.,J. Catal.,2006,241(2),312—318
[72] Jiang J. X.,Xu Y.,Cheng P.,Sun Q. M.,Yu J. H.,Corma A.,Xu R. R.,Chem. Mater.,2011,23(21),4709—4715
[73] Choi K.,Gardner D.,Hilbrandt N.,Bein T.,Angew. Chem. Int. Ed.,1999,38(19),2891—2894
[74] Caremans T.,Kirschhock C.,Verlooy P.,Paul J.,Jacobs P.,Martens J.,Micropor. Mesopor. Mater.,2006,90(1—3),62—68
[75] Klein J.,Lehmann C.,Schmidt H.,Maier W.,Angew. Chem. Int. Ed.,1998,37(24),3369—3372
[76] Havrilla G.,Miller T.,Rev. Sci. Instrum.,2005,76(062201),1—6
[77] Kubanek P.,Schmidt H.,Spliethoff B.,Scuth F.,Micropor. Mesopor. Mater.,2005,77(1),89—96
[78] Wang H.,Liu Z. M.,Shen J. H.,J. Comb. Chem.,2003,5(6),802—808
[79] Cichocka M.,Angstrom J.,Wang B.,Zou X.,Smeets S.,J. Appl. Crystallogr.,2018,51,1652—1661
[80] Baerlocher C.,McCusker L. B.,2001,http://www.iza-structure.org/databases/
[81] Zheng C. T.,Li Y.,Yu J. H.,2019,https://figshare.com/articles/dataset/AlPO_Structure_Database/7822574
[82] Zheng C. T.,Li Y.,Yu J. H.,Sci. Data,2020,7(107),1—8
[83] Earl D.,Deem M.,Ind. Eng. Chem. Res.,2006,45(16),5449—5454
[84] Li Y.,Yu J. H.,2019,https://figshare.com/articles/dataset/Enumerated_ABC-6_zeolites/7808477
[85] Yan Y.,Li J. Y.,Qi M.,Zhang X.,Yu J. H.,Xu R. R.,Sci. China Ser. B-Chem.,2009,52(11),1734—1738
[86] Li J. Y.,Yu J. H.,Xu R. R.,2011,http://zeobank.jlu.edu.cn/
[87] Jensen Z.,Kim E.,Kwon S.,Gani T.,Román-Leshkov Y.,Moliner M.,Corma A.,Olivetti E.,ACS Cent. Sci.,2019,5(5),892—899
[88] Liu X. D.,Xu Y. H.,Yu J. H.,Li Y.,Zeng W.,Chen C.,Li J. Y.,Pang W. Q.,Xu R. R.,Xu Y.,Chem. J. Chinese Universities,2003,24(6),949—952(刘晓东,徐翊华,于吉红,李乙,曾伟,陈超,李激扬,庞文琴,徐如人,XU Ying.高等学校化学学报,2003,24(6),949—952)
[89] Moliner M.,Serra J.,Corma A.,Argente E.,Valero S.,Botti V.,Micropor. Mesopor. Mater.,2005,78(1),73—81
[90] Li J. Y.,Qi M.,Kong J.,Wang J. Z.,Yan Y.,Huo W. F.,Yu J. H.,Xu R. R.,Xu Y.,Microporous Mesoporous Mater.,2010,129(1/2),251—255
[91] Daeyaert F.,Ye F. D.,Deem M. W.,Proc. Natl. Acad. Sci. U. S. A.,2019,116(9),3413—3418
[92] Schmidt J.,Deem M.,Lew C.,Davis T.,Top. Catal.,2015,58(7—9),410—41
[93] Davis T.,Liu A.,Lew C.,Xie D.,Benin A.,Elomari S.,Zones S.,Deem M.,Chem. Mater.,2016,28(3),708—711
[94] Li Y.,Li L.,Yu J. H.,Chem.,2017,3(6),928—949





