摘 要: 组蛋白是染色质的重要组成部分,其含量的高低显着影响着染色质高级结构的形成、基因表达、DNA复制等重要生命活动。蛋白质降解是组蛋白含量调控的重要机制,大量研究表明,组蛋白的降解与其氨基酸残基的修饰方式有着密不可分的联系。此外,组蛋白的降解与其泛素化、乙酰化等共价修饰密切相关。该文以组蛋白氨基酸残基翻译后修饰(PTM)为关注点,重点介绍组蛋白降解与氨基酸残基修饰之间的相互关系,并对该领域的研究进展进行综述。
关键词 : 组蛋白降解;泛素化;乙酰化;组蛋白修饰;翻译后修饰;
Abstract: Histone is an important component of chromatin,and its content significantly affects the formation of higher structure,gene expression,DNA replication and other important life activities of chromatin.Protein degradation is an important mechanism for the regulation of histone content.Numerous studies have shown that the degradation of histone is closely related to the modification of its amino acid residues.Besides,histone degradation is closely correlated with its covalent modifications such as ubiquitination and acetylation.This review focuses on PTM (post-translational modification) of histone amino acid residues,introduces the relationship between histone degradation and modification of amino acid residues,and reviews the recent advances in this field.
Keyword: histone degradation; ubiquitination; acetylation; histone modifications; post-translational modification;
染色质结构调控是生命科学研究的一个基本问题。染色质是由核小体串联而成的聚合物,每个核小体都由1个核心组蛋白八聚体、1个连接组蛋白H1以及1条包含大约147个碱基对的DNA组成。核心组蛋白则由H2A和H2B的2个异源二聚体、H3和H4的四聚体组成[1],组蛋白代谢在体内平衡中起着重要作用。组蛋白在很大程度上是稳定的,但在特定生理条件下,组蛋白的降解至关重要,影响着细胞的诸多生命活动。最新的研究发现,几乎所有的组蛋白都可以被共价修饰,组蛋白的尾巴从核小体伸出,可以发生多种不同的翻译后修饰,如:泛素化、磷酸化、甲基化、乙酰化、SUMO化、ADP核糖基化、丙酰化、丁酰化、巴豆酰化、2-羟基异丁酰化、丙二酰化、琥珀酰化、脂肪化、单氨化和糖基化等[2,3,4]。这些尾巴从染色质聚合物表面突出,占单个组蛋白质量的25%~30%,且对蛋白酶敏感。但它们也为组蛋白与其他蛋白质的相互作用提供了一个暴露的表面[5,6,7]。这些翻译后修饰对组蛋白含量、染色质结构、基因表达、DNA复制以及DNA损伤反应等重要事件都有非常大的影响[8]。但现阶段的研究仍然还存在一些问题:某些蛋白质残基修饰的位点、类型尚未确定;某些蛋白质残基修饰的具体作用仍不够清晰;各种蛋白质残基修饰之间串扰的具体作用及机制未明等。本文将对已经通过研究发现的蛋白质残基的翻译后修饰与组蛋白降解之间的关系做一个全面系统的阐述。
1、 多泛素化依赖的蛋白酶体降解途径
泛素是哺乳动物细胞中两种主要蛋白质降解途径(蛋白酶体途径和自噬–溶酶体途径)的底物识别靶点。最初溶酶体被认为是细胞蛋白质降解的主要场所。然而,后来的研究表明了大多数细胞蛋白质的半衰期都与溶酶体关系不甚密切,并发现了泛素–蛋白酶体这一重要的蛋白质降解的途径,即底物蛋白经泛素化标记后被称为蛋白酶体的26S蛋白水解复合物识别并降解[9]。泛素化是最广泛的蛋白质翻译后修饰之一,泛素–蛋白酶体途径介导的蛋白质降解主要由依赖ATP的E1泛素激活酶、E2泛素结合酶和E3泛素蛋白连接酶等一系列酶依次催化完成[10,11],这些酶将目标蛋白进行多泛素化或单泛素化标记,进而影响蛋白质的降解或生物学功能。泛素主要通过其内部的赖氨酸残基(如K6、K11、K27、K29、K33、K48和K63等)连接形成不同形式的多聚泛素链[12,13]。研究表明,K48或K11连接的多泛素化通常会导致底物被26S蛋白酶体降解,而单泛素化或K63连接多泛素化往往会导致底物的各种非蛋白水解结果,比如影响底物蛋白的生物学功能等[11]。

1.1、 RNF8介导的组蛋白H3的多泛素化修饰与蛋白质降解
RNF8(ring finger protein 8)是一种环指类E3泛素蛋白连接酶。研究发现,组蛋白H3的降解可由表皮生长因子受体(epidermal growth factor receptor,EGFR)诱导,进而导致RNF8的叉头相关(forkhead-associated,FHA)结构域与被PKM2(pyruvate kinase M2)磷酸化的组蛋白H3结合,且PKM2对H3的磷酸化主要发生在第11位苏氨酸(T11)上。RNF8与T11磷酸化的H3的结合,可使组蛋白H3在第四位赖氨酸(K4)发生多泛素化修饰。H3的这一多泛素化修饰则导致组蛋白从染色质上脱离以及核小体的解体,并进一步导致H3通过26S蛋白酶体途径被降解[14](图1)。
有研究表明,在胶质母细胞瘤(glioblastoma,GBM)中,用表皮生长因子(epidermal growth factor,EGF)处理细胞,然后通过低盐(low salt,LS)处理可将染色质分成2个部分:富含转录活性染色质片段的LS可溶部分和转录活性较低的LS不溶部分[14]。通过对2个组分进行定量分析,发现EGF处理细胞后,LS可溶性染色质组分中的组蛋白H3的蛋白水平显着降低,而LS不溶性染色质组分中的H3的蛋白水平基本不变,并且EGF诱导的组蛋白H3蛋白水平的下调可被EGFR抑制剂AG1478预处理所阻断。此外,蛋白酶体抑制剂MG132也可阻断EGF诱导的组蛋白H3蛋白水平的下调[14]。这些结果表明,EGF处理可能导致了转录活性染色质区域H3的多泛素化和蛋白酶体依赖的蛋白质降解。为了确定催化组蛋白H3发生泛素化修饰的E3泛素蛋白连接酶,研究人员通过过表达的方式,筛选了核定位的E3泛素蛋白连接酶[14]。结果发现,只有RNF8的过表达可增强组蛋白H3的蛋白质降解,这说明RNF8在其中很关键。免疫沉淀实验进一步表明,EGF处理诱导了组蛋白H3和RNF8之间的相互作用。RNF8优先与苏氨酸磷酸化的蛋白质结合,而组蛋白H3-T11的磷酸化在EGF促进的基因转录中起着关键作用[15,16]。在EGF的刺激下,上调的PKM2磷酸化组蛋白H3的T11位点,进而导致RNF8与H3的相互作用增强和H3的多泛素化修饰和蛋白质降解,最终促进EGF诱导的MYC和CCND1(CyclinD1)等基因的表达。此外,RNF8介导的核小体解离也促进了RNA聚合酶II与MYC和CCND1启动子区域的结合,从而激活c-Myc和CCND1等基因的表达,促进细胞的增殖和肿瘤发生。这说明,RNF8除了介导组蛋白的泛素化降解外,还影响着细胞其他的生命活动。
图1 RNF8介导的组蛋白H3的多泛素化修饰与蛋白质降解
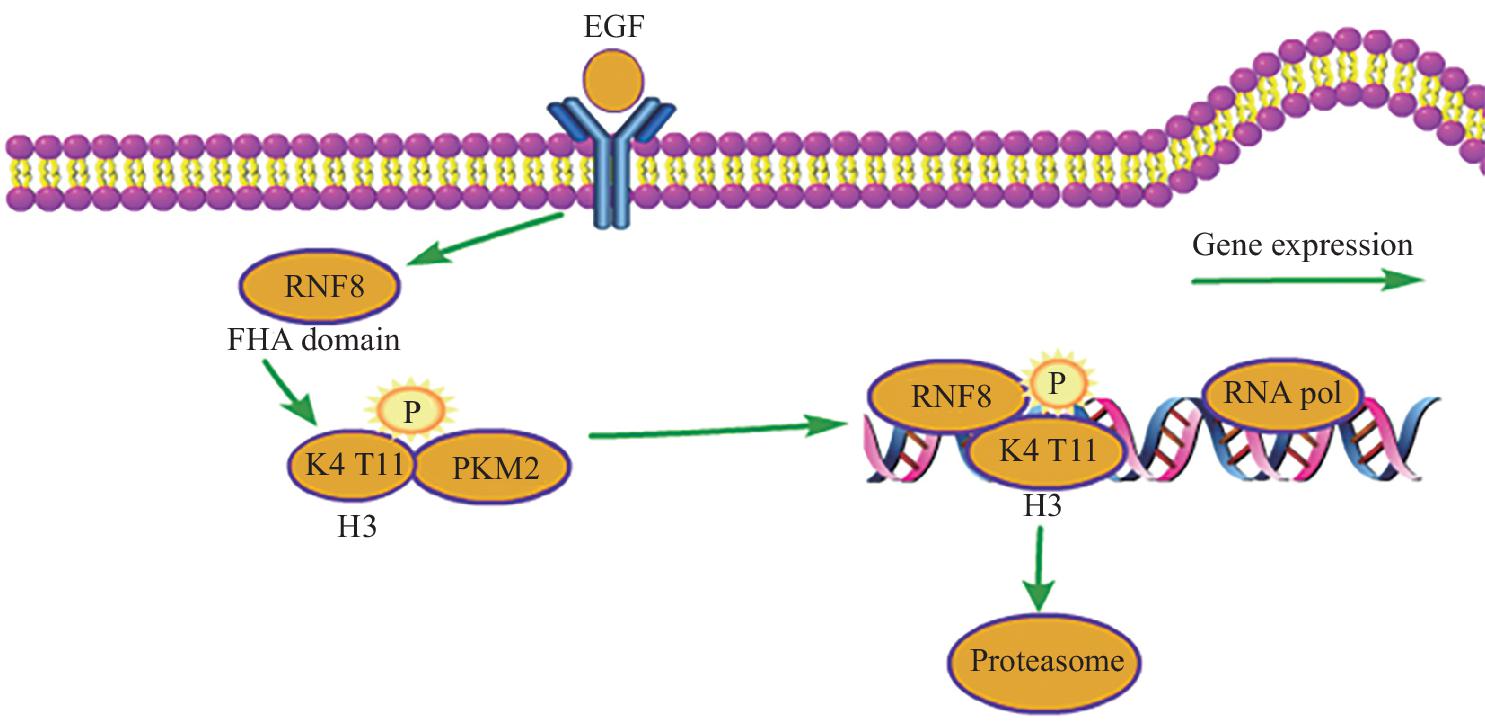
Fig.1 RNF8-mediated polyubiquitination and protein degradation of histone H3
1.2 、组蛋白H2B的蛋白质降解机制
图2 H2B的26S蛋白酶体降解途径
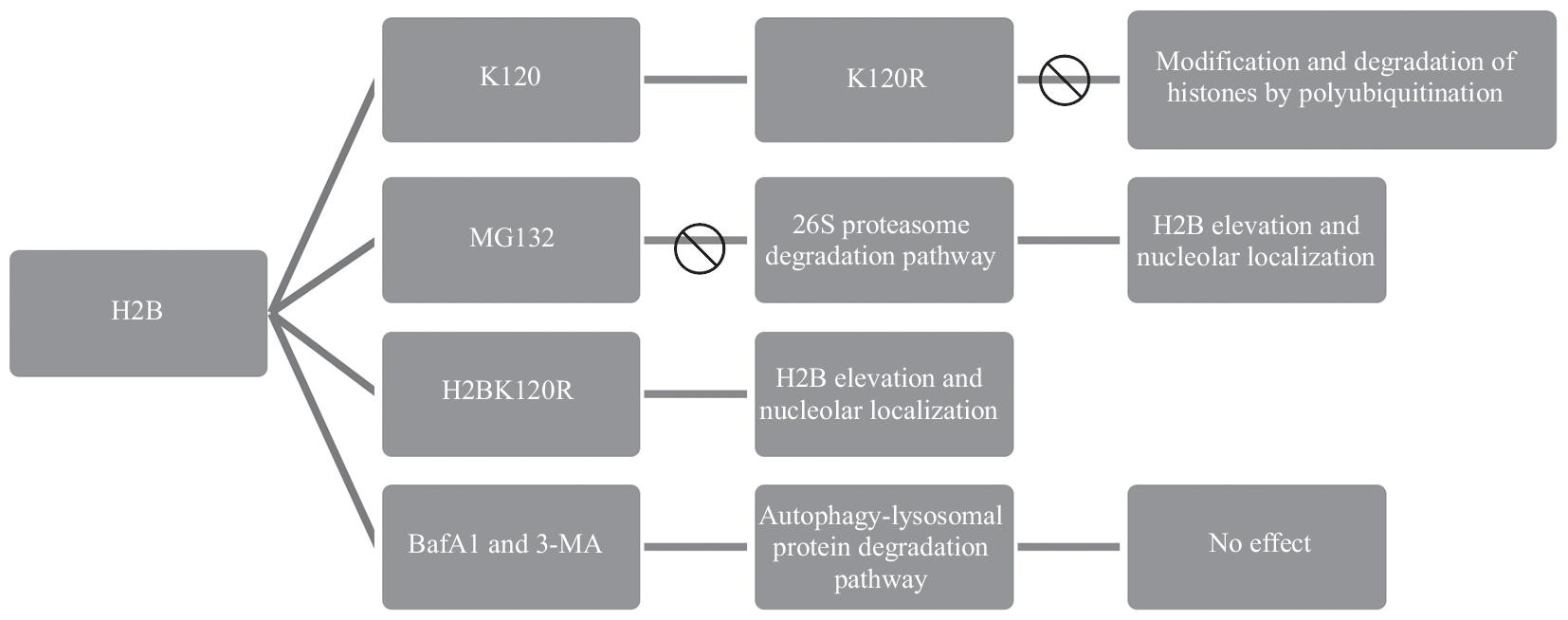
Fig.2 The 26S proteasome degradation pathway of H2B
组蛋白H2B主要通过其第120位的赖氨酸(K120)发生的K48连接,来介导多泛素化修饰,而多泛素化的H2B可以通过26S蛋白酶体途径被降解。研究表明,组蛋白H2B的K120位点突变为精氨酸(K120R)后,显着阻断了H2B的K48连接的多泛素化修饰和蛋白质降解,并导致了H2B发生显着的核仁定位[17]。这一结果表明,H2B的第120位赖氨酸残基是H2B蛋白降解所必需的。用蛋白酶体抑制剂MG132阻断26S蛋白酶体介导的蛋白降解途径,会导致野生型H2B蛋白的显着积累和野生型H2B显着的核仁定位。MG132处理对野生型H2B所产生的影响,与H2BK120R突变体的蛋白质含量和核仁定位变化极为相似。而用自噬抑制剂BafA1(bafilomycin A1)和3-MA阻断自噬–溶酶体介导的蛋白质降解途径则对H2B蛋白水平没有明显影响。这些结果表明,H2B的降解主要是通过26S蛋白酶体途径来完成的(图2)[17]。
除此之外,核仁在H2B降解中也非常重要。研究发现,核仁除了在核糖体发生和蛋白质翻译中的经典作用外,还是H2B蛋白质降解的发生场所[17]。用核仁的标志蛋白核仁素(nucleolin,NCL)对野生型H2B和H2BK120R突变体进行双重染色,发现H2BK120R突变蛋白可与NCL共定位,而野生型H2B在细胞核中则呈弥散样均匀分布,并在核仁位置处呈现出空泡样分布特点。这一结果表明,突变的H2BK120R蛋白可能在核仁中积累。Western blot实验也进一步证实了这一结果,将核仁组分和其他细胞组分分别提取出来,Western blot实验显示,H2BK120R突变蛋白主要分布在核仁组分中,并且MG132处理可显着增加野生型组蛋白H2B在核仁中的含量。H2BFWT的K120同源位点(R164)存在1个天然的K-R突变。研究人员发现,与H2BK120R和MG132处理的野生型H2B类似,H2BFWT存在显着的核仁定位,而将H2BFWT的R164位点反向突变为赖氨酸(R164K)后,H2BFWTR164K突变体的核仁分布则大大减少[17,18,19]。这一结果进一步表明,H2B的K120同源位点在调控H2B的核仁积累中发挥着关键作用,H2B的多泛素化修饰及其蛋白质降解主要发生在核仁内,核仁也是H2B发生降解的主要场所(图3)。
图3 H2B的降解发生在核仁中
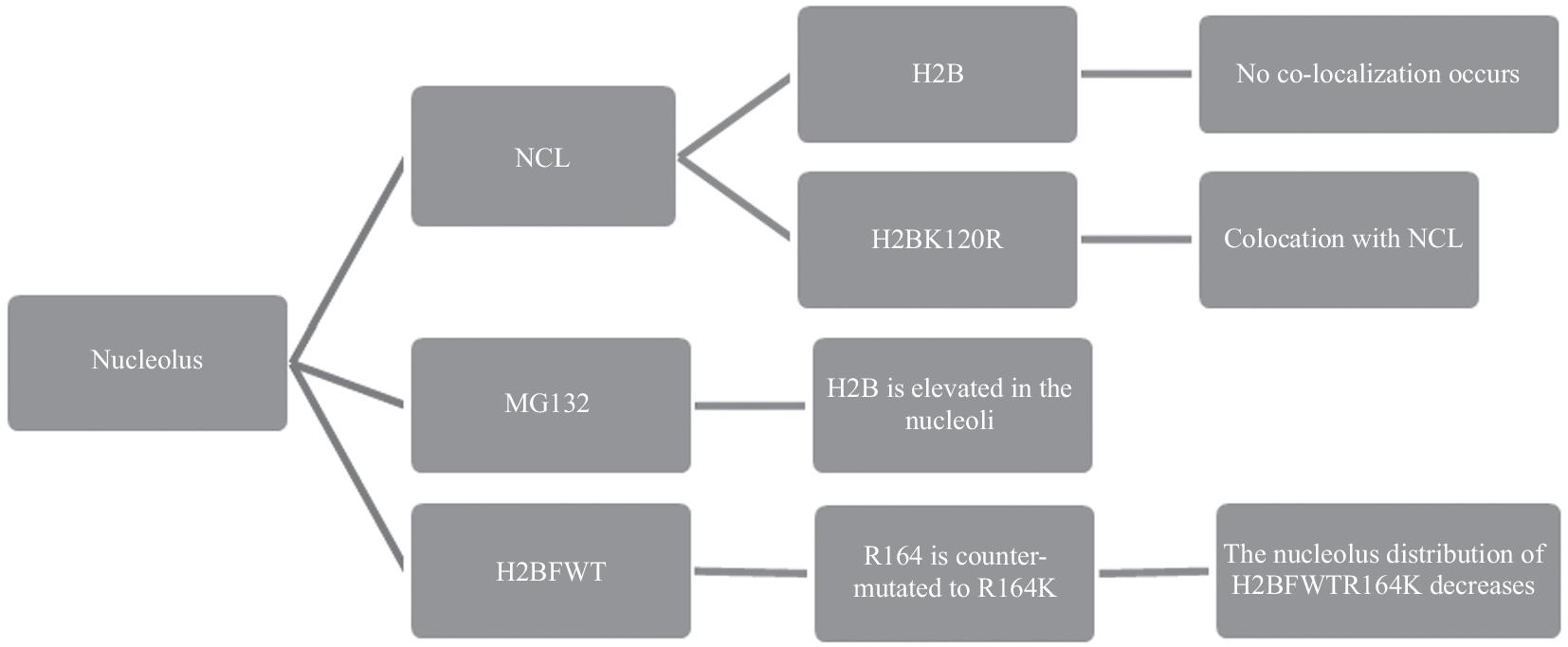
Fig.3 The degradation of H2B occurs in the nucleolus
2 、乙酰化依赖的非ATP蛋白酶体降解途径
组蛋白乙酰化广泛参与多种细胞过程,在染色质重塑、DNA损伤修复和基因表达等表观遗传调控中都起着关键作用。研究发现,组蛋白的乙酰化还在调控组蛋白降解中发挥关键的调控作用,它可通过阻止或促进底物蛋白的多泛素化修饰,从而影响底物蛋白的溶酶体降解或蛋白酶体的降解[20,21,22]。
PA200是一种存在于所有哺乳动物中的蛋白酶体调节组分的激活剂,PA200在睾丸细胞中呈现高表达。Blm10是PA200在酵母中的直系同源物。PA200/Blm10在细胞核中与乙酰化的核心组蛋白共定位,并通过溴结构域样区域(BRD-like,BRDL)结合乙酰化的核心组蛋白,从而促进核心组蛋白发生乙酰化依赖性的蛋白质降解[23,24]。研究发现,在哺乳动物睾丸中精子形成阶段的减数分裂后期,细胞的组蛋白主要被一种过渡蛋白瞬时取代,随后又被精蛋白取代[25,26]。有研究表明,组蛋白乙酰化发生在细长精子细胞中的组蛋白去除之前,并且发生在DNA双链断裂(double strand break,DSB)区域附近[27]。尽管与PA200/Blm10的BRDL结构域结合的乙酰化组蛋白的性质还需进一步鉴定,但免疫沉淀实验发现,与PA200/Blm10的BRDL结构域结合的组蛋白H2B存在乙酰化修饰,表明H2B的乙酰化可能是结合PA200/Blm10的BRDL结构域所必需的。此外,研究还发现,组蛋白H4第16位赖氨酸(K16)的乙酰化也参与了组蛋白的降解[28]。在赖氨酸残基乙酰化过程中,组蛋白去乙酰化酶及其相互作用因子可使转录失调,引起线粒体功能障碍、细胞凋亡、炎症反应和细胞周期停滞等,从而破坏脑细胞环境稳态,引起神经细胞死亡[29]。
在单倍体精子细胞的伸长过程中,PA200通过其BRDL结构域识别乙酰化或其他未知的翻译后修饰的核心组蛋白,将组蛋白从核小体中解离,并导致核心组蛋白裂解成小肽而降解。同时,过渡蛋白被招募到染色质中,并最终被精蛋白所取代。而在体细胞DNA修复过程中,研究发现,DNA双链断裂会触发核心组蛋白发生乙酰化或其他未确定的翻译后修饰,导致核心组蛋白从染色质上释放,从而使DNA修复蛋白能够有效修复受损DNA。同时,乙酰化的核心组蛋白至少有一部分被含有PA200/Blm10的蛋白酶体降解。损伤的DNA修复后,新合成的核心组蛋白与DNA结合并形成核小体[28]。
在酿酒酵母中,PA200的同源蛋白Blm10同样可以诱导DNA损伤修复过程中的组蛋白降解。在二倍体酵母中,MMS和HDAC抑制剂丙戊酸(valproic acid,VPA)联合处理显着降低了核心组蛋白H2B的蛋白水平;而用蛋白酶体抑制剂MG132处理或删除Blm10可显着阻断H2B蛋白水平的这种减少效应[30]。因此,这一结果提示,DNA双链断裂可能促进了核心组蛋白的乙酰化相关蛋白酶体降解。在小鼠体内,PA200的缺失延缓了细长精子细胞核心组蛋白的消失,导致核心组蛋白在伸长的精子细胞中积累。
最新的研究发现,PA200和Blm10的缺失都可以抑制H4或者组蛋白转录偶联变异体H3.3的降解。组蛋白去乙酰化酶抑制剂能使H3的降解速度加快,PA200的乙酰赖氨酸结合区的突变则抑制了G1期细胞中组蛋白的降解。尤其是在细胞老化的过程中,PA200的缺失能造成活性转录标志物(H3K4me3和H3K56ac)在细胞中的显着积累。PA200和Blm10的缺失均会加速细胞衰老,PA200缺陷小鼠表现出一系列衰老的特征,包括免疫功能障碍、焦虑样行为甚至寿命的缩短。PA200促进核心组蛋白与转录相关的降解,在转录和老化过程中维持组蛋白标志物的稳定性起着重要作用[30]。
虽然PA200与乙酰化依赖的蛋白酶体降解通路有密切联系,影响正常的生命活动,但最近有研究发现,缺乏α4S的小鼠疾病表型甚至比缺乏PA200的小鼠还要严重[33]。在睾丸中,蛋白酶体主要特异化为精浆蛋白酶体,除PA200外,精浆蛋白酶体还含有睾丸特异性的20S亚基α4S/PSMA8和免疫蛋白酶体的催化亚基。其中α4S特异性表达于粗线期精母细胞及其衍生的细胞(包括精子细胞及精子),能够促进精浆蛋白酶体的正确形成,从而对男性生育能力产生至关重要的作用。精母细胞标志物SYCP3(synthetic peptide 3)的免疫染色表明,在α4S缺陷的睾丸中,减数分裂过程在精母细胞阶段停止,阻止了精母细胞DNA损伤位点核心组蛋白的降解,导致其在减数分裂中期I停滞,而PA200可以刺激α4S通过26S蛋白酶体通路降解乙酰化的核心组蛋白。典型的26S蛋白酶体除了19S调节颗粒外,还含有3个组成性催化亚基(β1、β2和β5),而免疫蛋白酶体则包含不同的催化亚基(β1i、β2i和β5i)。α4S的缺失显着降低了成人睾丸中PA200和3个组成性催化亚基的蛋白水平,增加了免疫蛋白酶体的催化亚基以及蛋白酶体激活剂PA28α和PA28β的蛋白水平。而免疫蛋白酶体的催化亚基(β1i、β2i和β5i)可以转换为α4S/PSMA8缺陷睾丸中的组成性催化亚基,这两种亚基之间的转换也可能是造成α4S/PSMA8缺陷小鼠严重表型的原因。α4S/PSMA8促进精浆蛋白酶体的正常形成,并且通过促进组蛋白降解,对减数分裂中期I的DNA修复和雄性生育功能等起到重要作用。在α4S缺失的睾丸中,乙酰化组蛋白的降解减少证明含有PA200的蛋白酶体在体细胞DNA损伤修复和精子发生过程中降解乙酰化的核心组蛋白。这项研究表明,在精母细胞减数分裂修复和雄性生育过程中,α4S是去除乙酰化核心组蛋白所必需的。PA200基因的缺失只是延缓了减数分裂后精子细胞伸长过程中核心组蛋白的降解,从而降低了雄性生育力,而组蛋白的最终降解应由20S颗粒中的催化亚基执行[33]。
3、 组蛋白残基修饰的动态交叉调节
组蛋白常被认为是一类结构性蛋白,在将DNA压缩、组装成染色质的过程中发挥关键的结构性作用。如前所述,组蛋白的大量氨基酸残基存在不同类型的翻译后修饰。这些不同类型的组蛋白修饰调节着许多细胞功能,比如:基因转录,DNA复制、修复和重组,以及组蛋白的降解等[30,31,32]。染色质结构是高度动态变化的,染色质结构的动态变化在一定程度上受组蛋白翻译后修饰的影响。组蛋白的泛素化修饰和其他类型的翻译后修饰,包括组蛋白的磷酸化、甲基化和乙酰化等,可以相互交叉调节[34,35]。例如:一个修饰可以顺式影响同一组蛋白分子的其他修饰或反式调控组蛋白分子之间或跨核小体的一个或多个后续修饰的发生。不同组蛋白修饰相互关联,甚至可以形成多种信息性的“密码”,被称为“组蛋白密码”。这些组蛋白修饰往往通过结合下游的效应器蛋白来驱动不同的下游生物学功能[36,37]。
组蛋白修饰之间的相互调节存在多种类型,例如:同一修饰位点的不同组蛋白修饰之间存在竞争性的相互抑制,这一类组蛋白修饰之间的调控被称为原位串扰。关于原位串扰,一个研究得很好的例子是:组蛋白H3的第9位赖氨酸(K9)既可以被甲基化(H3K9me)也可以被乙酰化(H3K9ac),但两者之间存在相互排斥。组蛋白的乙酰化长期以来一直被认为与基因表达的增强密切相关;而组蛋白的甲基化则与基因的转录激活和转录抑制均存在密切关联,组蛋白甲基化对基因表达的不同影响取决于甲基化发生的部位和程度。除芽殖酵母外,大多数真核生物中都存在高水平的H3K9甲基化,而其通常与H3K9乙酰化在基因调控中的功能相反[38,39]。该残基的乙酰化与基因的活跃转录密切相关,而甲基化则通常与基因的转录抑制有关。这些修饰之间的相互调控可能有助于促进细胞处于不同的转录状态,控制基因的精确表达。
组蛋白修饰的交叉调节也可以发生在同一组蛋白不同修饰位点之间(顺式调控)甚至不同组蛋白的不同修饰之间(反式调控)。例如:组蛋白H2B K120位点的单泛素化修饰是组蛋白H3的K4和第79位赖氨酸(K79)位点甲基化的决定因素[40]。此外,在芽殖酵母中,组蛋白H2B K11位点乙酰化而抑制了其邻位第10位丝氨酸(S10)的磷酸化,组蛋白甲基转移酶Ste20只能在H2BK11去乙酰化的状态下才能对邻位的H2BS10进行磷酸化。这表明,H2B的乙酰化和磷酸化之间存在串扰现象。且有许多研究还提示,H2B的乙酰化和磷酸化之间的相互串扰在高等真核生物中可能也是保守的[41,42,43]。在处于对数生长期的酵母细胞中,组蛋白H2BK11的乙酰化由乙酰转移酶Gcn5催化完成,而在濒临死亡的酵母细胞中,组蛋白去乙酰化酶Hos3可以催化H2BK11ac的脱乙酰化[44,45],且H2BK11的去乙酰化是酵母激活凋亡通路所必需的。而当H2BK11突变为谷氨酰胺(Q)时,谷氨酰胺不带电荷,可以模拟乙酰化状态,进而激活凋亡通路。所以,H2BK11Q的突变体细胞在对H2O2诱导的细胞死亡中具有较好的抵抗力。
有些修饰位点之间的间距很近,也许我们可以通过寻找间距很近的修饰位点来预测组蛋白修饰之间的其他交叉调节[46]。例如,组蛋白H3的Arg2、Thr3和Lys4可以分别被甲基化、磷酸化和甲基化,这些修饰之间就很容易存在串扰。同样,H3R26的甲基化、H3K27的甲基化或乙酰化、H3S28的磷酸化之间也可能存在相互影响。此外,H3R8的甲基化和H3T11的磷酸化可以通过H3K9和H3S10的修饰来交叉调节。组蛋白泛素化和其他类型的翻译后修饰,包括磷酸化、甲基化和乙酰化,也可以相互交叉调节,进而影响组蛋白的降解。最新的研究发现,赖氨酸残基乙酰化位点的缺失会导致邻近位点泛素化和SUMO化功能的丧失[29]。
4、 组蛋白降解的生物学意义
组蛋白的降解不仅调节着染色质结构和基因的表达,还可以影响糖酵解和肿瘤发生等重要病理、生理过程。
4.1 、组蛋白降解与糖酵解调控
RNF8介导的组蛋白H3的多泛素化修饰和蛋白质降解能显着提高肿瘤细胞的糖酵解水平。有研究表明,敲除掉RNF8或组蛋白H3.3后,U87/EGFRvIII细胞可对野生型RNF8(WT-rRNF8)、FHA结构域突变的RNF8(rRNF8-FHA-M)、野生型组蛋白rH3.3B、rH3.3B-K4R突变体进行表达[47]。与U87细胞相比,过表达EGFR的U87/EGFRvIII细胞具有更高的葡萄糖消耗水平和乳酸产生水平,提示该细胞的糖酵解水平显着增加。而糖酵解水平的增加可被RNF8的敲除所抑制。进一步研究表明,RNF8主要是通过调节组蛋白H3的稳定性来调节肿瘤细胞的糖酵解水平[14]。组蛋白乙酰转移酶p300可诱导细胞内磷酸烯醇式丙酮酸羧激酶PCK1乙酰化,且乙酰化后的磷酸烯醇式丙酮酸转化为草酸乙酯的反应增强,以此来调节胞质PCK1的活性,而PCK1是糖异生的关键酶[48]。因此,乙酰化修饰与糖酵解关系密切。
4.2、 组蛋白降解与肿瘤发生
MYC是一个公认的癌基因,而CCND1经常被用来对肿瘤的发展状态进行评估。研究发现,PKM2可使MYC和CCND1基因启动子区域的组蛋白H3的T11位点发生磷酸化,进而促进MYC和CCND1基因的转录[14]。而且EGF刺激会导致PKM2催化的H3T11磷酸化在MYC和CCND1启动子区域增加,同时还导致了E3泛素蛋白连接酶RNF8被募集到MYC和CCND1基因的启动子区域。招募过来的RNF8可特异性识别磷酸化的H3T11,并导致H3的多泛素化修饰和蛋白质降解。与EGF类似,血小板衍生生长因子(platelet-derived growth factor,PDGF)也能诱导PKM2和RNF8依赖的组蛋白H3的蛋白质降解以及c-Myc和CCND1的表达。进一步研究发现,过表达FHA结构域突变的RNF8(rRNF8-FHA-M)和第4位赖氨酸突变的H3(rH3.3B-K4R)可以抑制肿瘤细胞的增殖,表明RNF8的酶活性和H3的多泛素化修饰对肿瘤细胞的增殖至关重要[14]。相应地,RNF8的缺失可以抑制肿瘤的发生,过表达野生型RNF8(WT-rRNF8)可以抑制RNF8缺失所导致的这种生物学效应,但过表达FHA结构域突变的RNF8(rRNF8-FHA-M)则无此效应。依赖于RNF8的组蛋白H3降解在人类原代肿瘤细胞GSC11GBM中也被检测到,表明RNF8依赖的组蛋白H3的多泛素化修饰和蛋白质降解与肿瘤发生有着密切联系[14]。
最近又有研究发现,被广泛熟知的PKM2也参与了调控基因转录,进而影响肿瘤的发生。组蛋白去乙酰化酶8(HDAC8)能够结合PKM2的K62残基并使其去乙酰化,K62去乙酰化后促进PKM2进入细胞核并与β-catenin结合,从而促进CCND1基因转录。此外,K62的去乙酰化还能影响PKM2酶活性和葡萄糖代谢通量。这些结果提示,HDAC8/PKM2信号也可能会成为治疗肿瘤的新靶点[49]。
4.3、 组蛋白降解与DNA损伤修复
DNA双链断裂是一种高毒性的DNA损伤,可触发相邻染色质区域的非蛋白水解性泛素化修饰,从而产生DNA损伤修复因子的结合位点[50]。这取决于E3泛素连接酶RNF8和RNF168以及UBC13的顺序作用,其中UBC13是一种E2泛素结合酶,可特异性地产生K63连接的泛素链[51]。而RNF168可催化组蛋白H2A的泛素化并导致修复因子(如53BP1)的募集。
RNF8通过其FHA结构域与磷酸化的适配器蛋白MDC1发生相互作用,并在DSB位点上快速组装,泛素化组蛋白H2A和H2Ax。伴随着DSB区域相关泛素化修饰水平的增加,随之而来的是更多的DNA损伤修复蛋白,如53BP1和BRCA1等被募集到DSB区。研究发现,RNF8的敲除或其FHA结构域或环区结构域突变会显着抑制DSB区相关蛋白的泛素化修饰,并抑制53BP1和BRCA1在DSB区域的富集[52]。这些数据表明,MDC1介导的、RNF8执行的组蛋白泛素化对DNA损伤修复蛋白在DSB区的富集至关重要,并以此来保护基因组的完整性和稳定性。
5 、结语
在过去的十几年中,人们对组蛋白降解的分子机制做了很多深入的研究和探讨。组蛋白的降解与组蛋白氨基酸残基的修饰方式之间关系密切,不同的修饰具有不同的生理功能。这些修饰过的氨基酸残基通过结合下游的效应器蛋白来驱动不同的下游生物学功能,如基因表达、染色质的形成、蛋白质的降解等,以维持细胞正常的生命活动。而且各种修饰之间相互影响、相互制约,可以交叉调节,是一个动态的过程。组蛋白氨基酸残基的翻译后修饰是调节基因组遗传功能的关键环节之一,是组蛋白动态反应和相互作用的一个重要分子生物学基础,同时也是细胞信号网络调控的重要靶点。组蛋白的降解机制不仅影响着组蛋白的含量、染色质结构、基因表达、DNA复制,还参与糖酵解的调控、肿瘤的发生、DNA的损伤修复等。但到目前为止,我们对组蛋白降解调控的机制及相关因子还知之甚少,我认为其实在很长时间内,组蛋白残基修饰与降解的研究并未引起足够重视,我们需要对组蛋白降解的重要性重新进行定位,它不仅直接与遗传信息的传递息息相关,还会导致各种相关的疾病。如果有大量的与疾病相关的组蛋白修饰谱蛋白组研究成果,我觉得将会发现大量新的疾病标志物和疾病特异药靶蛋白。这个非常有趣且有意义的方向等待我们去继续研究。
参考文献
[1]LUGER K,MADER A W,RICHMOND R K,et al.Crystal structure of the nucleosome core particle at 2.8 A resolution[J]. Nature,1997. ,389(6648):251-60.
[2]EVANS A. Greetings from the planet croton[J].Mol Cell,2015,58(2):195-6.
[3]STRAHL B D,ALLIS C D.The language of covalent histone modifications[J].Nature ,2000,403(6765):41-5.
[4]CHAN J C,MAZE 1.Nothing is yet set in (Hi)stone:novel post-translational modifications regulating Chromatin function[J].Trends Biochem Sci,2020,45(10):829-44.
[5]HECHT A,LAROCHE T,GRUNSTEIN M,et al.Histone H3 and H4 N-termini interact with SIR3 and SIR4 proteins:a molecular model for the formation of heterochromatin in yeast[J].Cell, 1995,80(4):583-92.
[6]EDMONDSON D G,SMITH M M,ROTH S Y.Repression domain of the yeast global repressor Tup1 interacts directly with histones H3 and H4[J].Genes Dev,1996, 10(10):1247-59.
[7]LUGER K,MADER A W,RICHMOND R K,et al.Crystal structure of the nucleosome core particle at 2.8 A resolution[J].Nature, 1997 ,389(6648):251-60.
[8]SEILER D M,ROUQUETTE J,SCHMID V J,et al.Double-strand break-induced transcriptional silencing is associated with loss of tri-methylation at H3K4[J].Chromosome Res,2011,19(7):883-99.
[9]CLAGUE M J,URBE s.Ubiquitin:same molecule different degradation pathways[J].Cell,2010,143(5).682-5.
[10]LU Z,HUNTER T.Degradation of activated protein kinases by ubiquitination[J] Annu Rev Biochem,2009,78(1):435-75.
[11]BASSERMANN F,EICHNER R,PAGANO M.The ubiquitin proteasome system-implications for cell cycle control and the targeted treatment of cancer[J]. Biochim Biophys Acta,2014, 1843(1):150-62.
[12]KOROLCHUK V 1,MENZIES F M.RUBINSZTEIN D C.Mechanisms of cross-talk between the ubiquitin-proteasome and autophagy-lysosome systems[J].FEBS Lett,2010,584(7):1393-8.
[13]BEHRENDS C,HARPER J W.Constructing and decoding unconventional ubiquitin chains[J] Nat Struct Mol Biol,2011,18(5):520-8.
[14]XIA Y,YANG W,FA M,et al. RNF8 mediates histone H3 ubiquitylation and promotes glycolysis and tumorigenesis[J].J Exp Med,2017,214(6):1843-55.
[15]HUEN M S,GRANT R,MANKE 1.et al.RNF8 transduces the DNA-damage signal via histone ubiquitylation and checkpoint protein assembly[J]. Cell,2007,131(5):901-14.
[16]CHACKALAMANNIL S,WANG Y,GREENLEE W J.et al.Discovery of a novel,orally active himbacine based thrombin receptor antagonist (SCH 530348) With potent antiplatelet activity[J].J Med Chem, 2008,51(11):3061-4.
[17]LIU Y,WANG Y,CHEN S.et al.The nucleolus functions as the compartment for histone H2B protein degradation[J.iScience,2021,24(4):102256.
[18]SONG Z,WU M.Identification of a novel nucleolar localization signal and a degradation signal in Survivin-deltaEx3:a potential link between nucleolus and protein degradation[J] Oncogene ,2005 .24(16):2723-34.
[19]GUAN Y,HUANG D.PENG J,et al.Phosphorylation of Def regulates nucleolar p53 turnover and cell cycle progression through Def recruitment of Calpain3[J].PLoS Biol,2016,14(9):e1002555.
[20]MATEO F,VIDAL-LALIENA M,BACHS O,et al.Degradation of cyclin A is regulated by acetylation[J].Oncogene ,2009,28(29).2654-66.
[21]ROBERT T,VANOLI F,FOIANI M,et al.HDACs link the DNA damage response processing of double-strand breaks and autophagy[J].Nature,2011, 471(7336):74-9.
[22]VAN LOOSDREGT J,VERCOULEN Y,COFFER P J,et al.Regulation of Treg functionality by acetylation-mediated Foxp3 protein stabilization[J]. Blood,2010,115(5):965-74.
[23]ORTEGA J,HEYMANN J B,STEVEN A C,et al.The axial channel of the 20S proteasome opens upon binding of the PA200 activator[J]J Mol Biol,2005,346(5):1221-7.
[24]HAMMOUD S S,NIX D A,CAIRNS B R,et al.Distinctive chromatin in human sperm packages genes for embryo development[J]. Nature,2009,460(7254).473-8.
[25]MILLS N C,VAN N T,MEANS A R.Histones of rat testis Chromatin during early postnatal development and their interactions with DNA[J] Biol Reprod,1977,17(5):760-8.
[26]GAUCHER J.REYNOIRD N,KHOCHBIN S.et al.From meiosis to postmeiotic events.the secrets of histone disappearance[J].FEBS J,2010,277(3):599-604
[27]MURR R,LOIZOU J I,HERCEG Z,et al.Histone acetylation by Trrap-Tip60 modulates loading of repair proteins and repair of DNA double-strand breaks[J].Nat Cell Biol,2006,8(1):91-9.
[28]QIAN M X,PANG Y,QIU X B,et al.Acetylation-mediated proteasomal degradation of core histones during DNA repair and spermatogenesis[J]. Cell,2013,153(5):1012-24.
[29]GUPTA R,KUMAR P.Computational analysis indicates that PARP1 acts as a histone deacetylases interactor sharing common lysine residues for acetylation,ubiquitination,and SUMOylation in Alzheimer's and Parkinson's disease[J] ACS Omega,2021 ,6(8):5739-53.
[30]JIANG T X,MA S,QIU X B,et al. Proteasome activator PA200 maintains stability of histone marks during transcription and aging[J]. Theranostics ,2021,11(3)-1458-72.
[31]ZHANG Z H,JIANG T X,QIU X B,et al. Proteasome subunit a4s is essential for formation of spermatoproteasomes and histone degradation during meiotic DNA repair in spermatocytes[J].J Biol Chem, 2021,296:100130.
[32]LI B,CAREY M,WORKMAN J L.The role of Chromatin during transcription[J].Cell,2007, 128(4):.707-19.
[33]GROTH A.ROCHA W,ALMOUZNI G. Chromatin challenges during DNA replication and repair[J].Cell,2007,128(4):721-33.
[34]SUN Z W,ALLIS C D.Ubiquitination of histone H2B regulates H3 methylation and gene silencing in yeast[J]. Nature,2002 ,418(6893):104-8.
[35]L ATHAM J A.DENT S Y.Cross-regulation of histone modifications[J].Nat Struct Mol Biol,2007,14(11):1017-24.
[36] STRAHL B D.ALLIS C D.The language of covalent histone modifications[J]. Nature ,2000, 403(6765):41-5.
[37]TURNER B M.Histone acetylation and an epigenetic code[J] Bioessays, 2000,22(9):836-45.
[38]NAKAYAMA J,RICE J C,GREWAL S I,et al.Role of histone H3 lysine 9 methylation in epigenetic control of heterochromatin assembly[J] 2001,292(5514):110-3.
[39]REA S,EISENHABER F,JENUWEIN T,et al.Regulation of chromatin structure by site-specific histone H3 methyltransferases[J]. Nature,2000,406(6796).593-9.
[40]NAKANISHI S,LEE J S,SHILATIFARD A.Histone H2BK123 monoubiquitination is the critical determinant for H3K4 and H3K79 trimethylation by COMPASS and Dot1[J].J Cell Biol,2009, 186(3):371-7.
[41]CHEUNG W L,AJIRO K,ALLIS C D,et al.Apoptotic phosphorylation of histone H2B is mediated by mammalian sterile twenty kinase[J].Cell, 2003,113(4):507-17.
[42]THORNE A W,KMICIEK D,CRANE-ROBINSON C ,et al.Patterns of histone acetylation[J]. Eur J Biochem, 1990,193(3):701-13.
[43]SHIL ATIFARD A.Chromatin modifications by methylation and ubiquitination:implications in the regulation of gene expression[J] Annu Rev Biochem,2006,75(1):243-69.
[44]SUKA N,SUKA Y,GRUNSTEIN M.et al.Highly specific antibodies determine histone acetylation site usage in yeast heterochromatin and euchromatin[J].Mol Cell,2001 ,8(2):473-9.
[45]AHN S H,DIAZ R L,ALLIS C D,et al.Histone H2B deacetylation at lysine 11 is required for yeast apoptosis induced by phosphorylation of H2B at serine10[J.Mol Cell,2006 24(2):211-20.
[46] LATHAM J A,DENT S Y.Cross-regulation of histone modifications[J].Nat Struct Mol Biol,2007, 14(11):1017-24.
[47]YANG W,XIA Y,LU Z,et al.EGFR-induced and PKCε monoubiquitylation-dependent NF-KB activation upregulates PKM2 expression and promotes tumorigenesis[J].Mol Cell,2012. 48(5):771-84.
[48]L ATORRE-MURO P,BAEZA J,CARRODEGUAS J A.et al.Self-acetylation at the active site of phosphoenolpyruvate carboxykinase (PCK1) controls enzyme activity[J].J Biol Chem,2021 ,296: 100205.
[49]ZHANG R SHEN M,LIU J,et al.HDAC8-dependent deacetylation of PKM2 directs nuclear localization and glycolysis to promote proliferation in hepatocellular carcinoma[J] Cell Death Dis,2020,11(12):1036.
[50]THORSLUND T,RIPPLINGER A.MAILAND N.et al.Histone H1 couples initiation and amplification of ubiquitin signalling after DNA damage[J].Nature,2015,527(7578):389-93.
[51]HOFMANN R M,PICKART C M.Noncanonical MMS2-encoded ubiquitin-conjugating enzyme functions in assembly of novel polyubiquitin chains for DNArepair[J].Cell, 1999,96(5):645-53.
[52]VEMPATI R K,HALDAR D.DNA damage in the presence of chemical genotoxic agents induce acetylation of H3K56 and H4K16 but not H3K9 in mammalian cells[J].Mol Biol Rep,2012,39(1):303-8 .





