
Lactobacillus paraplantarumL-XM1是分离自卡萨莫迪娜那不勒斯风味萨拉米香肠的一株Ⅱb类细菌素产生菌。乳杆菌Ⅱ类细菌素的合成通常由群体感应系统调控,目前已知的主要有plnABCD、plnNC8IF-HK-D、orf4-sapK-sapR和orfY-sppK-sppR等系统。经PCR扩增,从L.paraplantarumL-XM1中克隆到了群体感应系统编码基因的同源基因plnBCD,了解plnBCD编码蛋白的分子特征及其结构,对群体感应调控机制的研究及运用具有重要的意义。
Man等用生物信息学方法研究了L.plantarumKLDS1.0391的群体感应系统,Cui等对L.acidophilus群的群体感应系统进行了生物信息学分析,葛菁萍等对副干酪乳杆菌的感应调节蛋白基因序列进行了分析,但尚未见对L.paraplantarum群体感应系统组分进行生物信息学分析的相关报道。本研究运用序列同源性比较及功能域分析等生物信息学手段对L.paraplantarumL-XM1的plnBCD基因进行理化性质分析预测、保守性功能域分析、二级结构预测以及空间结构同源建模,并对其功能进行预测,旨在丰富产Ⅱ类细菌素乳杆菌群体感应调控系统的生物信息库,为该菌群体感应调控机制的研究奠定基础,为发现新的群体感应调控系统提供参考。
1 材料与方法
1.1 数据来源
本研究使用的类植物乳杆菌L-XM1(中国农业大学应用微生物学实验室分离并保存)plnBCD数据来源于NCBI,GenBank accession number:JQ974915-JQ974917。
1.2 生物信息学分析
利用NCBI网站上的BLASTN软件对plnBCD进行序列同源性分析。运用DNAMAN软件对plnBCD核苷酸序列进行翻译,然后将编码的氨基酸序列进行同源性分析(NCBI网站上的BLASTP软件)、蛋白质的分子质量和等电点预测、保守功能结构域检索(NCBI网站上的RPS-Blast工具以及EBI网站上的InterProscan工具)、跨膜螺旋区域分析(TMHMM Server v.2.0)、疏水性分析、磷酸化位点预测、亚细胞定位以及蛋白质二级结构预测。采用Swiss-model软件对L.paraplantarum L-XM1plnBCD的保守性功能域进行空间结构建模。
2 结果与分析
2.1 plnB生物信息学
组氨酸蛋白激酶是群体感应系统的重要组成部分,一方面它与群体感应信号分子结合启动群体感应调控,另一方面,它将感应调节蛋白磷酸化,引起下游应答。对HPK空间结构的研究,无疑将有利于更进一步解析群体感应系统各组分的间相互作用机制。
相似性比对结果发现,L.paraplantarumL-XM1plnB与L.plantarum WCFS1以及C11中的组氨酸蛋白激酶编码基因plnB同源性最高,达到97.51%,与L.plantarumJ51组氨酸蛋白激酶编码基因plnB的同源性也高达97.29%。菌株L-XM1的plnB与L.pentosus MP-10的组氨酸蛋白激酶编码基因plnB同源性为70.59%,与L.sakei中调控细菌素合成的组氨酸蛋白激酶编码基因sppK的同源性为23.82%。
plnB基因共编码442个氨基酸,其氨基酸序列与L.plantarum细菌素合成相关的组氨酸蛋白激酶(GenBank accession No.FJ809773.1)的同源性为97%。
序列分析表明,plnB编码蛋白PlnB含有38个强碱性氨基酸(K、R);45个强酸性氨基酸(D、E)。预测分子质量为50 454.7,等电点pI=5.59,为一疏水、稳定蛋白,GRAVY(Grand average ofhydropathicity)值为0.301,不稳定系数为31.04。该蛋白具有21个磷酸化位点,符合大多数组氨酸蛋白激酶的基本性质。保守功能结构域检索结果显示,在PlnB的C端存在一个结合ATP的激酶活性功能域HATPase_c,跨膜区分析显示,PlnB是一个膜蛋白,有6个跨膜区(图1),定位于细胞质膜上,与前人对组氨酸蛋白激酶的描述一致。【图1】

以上结果显示,PlnB具有典型的组氨酸蛋白激酶的结构特点,故可以认为PlnB具有组氨酸蛋白激酶的功能。该蛋白在群体感应系统中,作为信号分子的受体,可以通过自身的磷酸化和去磷酸化,起到信号转导的作用。二级结构预测结果表明L.paraplantarumL-XM1的PlnB蛋白主要由α螺旋、延伸链、β转角和无规卷曲组成。其中α螺旋占64.71%、延伸链占15.61%、β转角占4.98%、无规卷曲占14.71%。
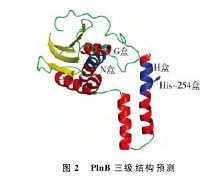
采用SWISS-MODEL server的自动选择模式预测PlnB的三维结构,程序自动选择Thermotogamaritima Sensor histidine kinase(PBD code:2c2aA)作为模板。同源建模得到的从第246到第442位氨基酸残基的三维结构见图2,与模板蛋白的序列相似性为16.071,与二级结构预测结果基本一致,从而进一步验证了结构的正确性。该结构域由5个α-螺旋和5个反向平行的β片层组成,符合群体感应系统组氨酸蛋白激酶的结构特征。该结构域有3个位于C末端的激酶催化区域的盒:H盒、N盒和G盒,围绕形成了一个结合ATP的口袋,位于H盒的His-254为磷酸化位点,是磷酸信号转导的中心区域,这与前人对HPK10亚家族保守区域的描述相一致。已报道的产II类细菌素乳酸菌群体感应系统中的组氨酸蛋白激酶组分均为HPK10亚家族成员,故可认为plnB为群体感应系统的组氨酸蛋白激酶。【图2】
2.2 plnC生物信息学
感应调节蛋白可接受来自组氨酸蛋白激酶His残基上的磷酸基团,被磷酸化的感应调节蛋白激活子PlnC结合到启动子上,激活目的基因的表达。
通过比对发现,plnC基因序列与L.plantarumWCFS1中编码感应调节蛋白激活子的lp_0417(GenBank accession No.AL935263.2)相似度为98.66%。plnC基因共编码247个氨基酸,其氨基酸序列与L.plantarum中细菌素合成相关的感应调节蛋白激活子(GenBank accession No.FJ809773.1)的同源性为97%。
序列分析表明,plnC编码蛋白PlnC含有34个强碱性氨基酸(K、R);35个强酸性氨基酸(D、E)。预测分子质量为28 567.9,等电点pI=6.21,为一亲水、稳定蛋白,GRAVY值为-0.319。不稳定系数为34.69。该蛋白具有16个磷酸化位点。
保守功能结构域检索结果显示,PlnC具有2个结构域,分别为REC、LytTR。在PlnC的N端具有磷酸受体结构域REC,包括1个激活位点,1个磷酸化位点,1个分子间识别位点和二聚体接触面,预测为群体感应系统中的应答调控蛋白或信号传递蛋白。在其C端有一个LytTR结构域,具有结合DNA靶序列的功能,起到转录调节的作用。
PlnC不具有跨膜区,定位于细胞质中。以上结果显示,PlnC具有典型的感应调控因子的结构特点,该蛋白在群体感应系统中,可以通过自身的磷酸化和去磷酸化,起到信号转导的作用。
二级结构预测结果表明L.paraplantarumL-XM1的PlnC蛋白主要由α螺旋、延伸链、β转角和无规卷曲组成。其中α螺旋占47.77%、延伸链占25.51%、β转角占5.26%、无规卷曲占21.46%。
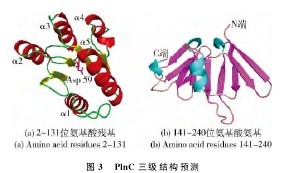
用SWISS-MODEL server的自动选择模式对PlnC的三维结构进行预测,程序自动选择Klebsiellapneuoniae mrkE蛋白的调节结构域(PBD code:2qv0B)和Staphylococcus aureus AgrA LytTR结构域(PBD code:3bs1A)作为模板。同源建模得到的从第2~131位氨基酸残基的三维结构见图3(a),与模板蛋白的序列相似性为24.615%,该结构域由5个α-螺旋和5个平行的β片层组成,其Asp-59残基是组氨酸蛋白激酶磷酸基团传递的受体位点,当磷酸化后,其构象发生变化,引起下游应答。同源建模得到的从第141~240位氨基酸残基的三维结构见图3(b),与模板蛋白的序列相似性为21%,该结构由10个反向平行的β片层和1个α-螺旋结构组成,便于与DNA分子结合。图3显示,根据同源建模得到的PlnC的三维结构具有典型的群体感应系统中感应调节蛋白组分的结构特征。【图3】
2.3 plnD生物信息学
被磷酸化后的感应调节蛋白抑制子PlnD可特异性地结合到目标启动子上,抑制目的基因的表达。
登陆NCBI进行BLAST比对发现,plnD基因序列与L.plantarumWCFS1中编码感应调节蛋白抑制子的lp_0418(GenBank accession No.AL935263.2)相似度为95.70%,与L.plantarumNC8中调控细菌素合成的PlnD(GenBankaccession No.EHS83009)同源性为96%。
plnD基因共编码247个氨基酸,其氨基酸序列与L.plantarum中细菌素合成相关的感应调节蛋白抑制子(GenBank accession No.FJ809773.1)的同源性为96%,故预测其为群体感应系统重要组分。
PlnD序列分析表明,该蛋白含有35个强碱性氨基酸(K、R);36个强酸性氨基酸(D、E)。预测分子质量为28 607.8,等电点pI=6.35,为一亲水、稳定蛋白,GRAVY值为-0.333,不稳定系数为28.74,该蛋白具有11个磷酸化位点。
保守功能结构域检索结果显示,该蛋白具有3个结构域,分别为REC、LytTR、LytT。二级结构预测结果表明L.paraplantarumL-XM1的PlnD蛋白主要由α螺旋、延伸链、β转角和无规卷曲组成。
其中α螺旋占47.37%、延伸链占25.91%、β转角占4.86%、无规卷曲占21.86%。采用SWISS-MODEL server的自动选择模式对PlnD的三维结构进行预测,以Klebsiella pneumoniae mrkE感应调节子结构域(PBD code为2qv0B)和Staphylococcusaureus AgrA LytTR结构域(PBD code为3bs1A)作为模板同源建模,得到从第2~130位氨基酸残基的三维结构见图4(a),与模板蛋白的序列相似性为24.031%,该结构域由5个α-螺旋和5个平行的β片层组成。该结构域第59位的Asp为磷酸化位点,这与Diep等的研究结果一致。同源建模得到的从第140~240位氨基酸残基的三维结构见图4(b),与模板蛋白的序列相似性为26.733%,该结构由10个反向平行的β片层和1个α-螺旋结构组成。以上结构特征与大多数群体感应调控系统的感应调节蛋白相同。【图4】

应用序列同源性比较及功能域分析等生物信息学手段对plnB、plnC和plnD编码蛋白进行预测分析发现,plnB编码蛋白具有典型的组氨酸蛋白激酶的结构特征,plnC和plnD编码蛋白具有典型的感应调节蛋白的结构特征,与Straume等的报道一致。故可以预测plnB、plnC和plnD编码蛋白是L.paraplantarumL-XM1群体感应系统的重要组分。
3 讨论
群体感应是调控细菌素合成的重要系统,了解群体感应系统重要组分的结构及与之相关的辅助信息,可为继续研究PlnBCD蛋白的结构与功能奠定基础,这对于解析群体感应调控机制,以及更好地调控细菌素合成具有重要的作用。
在对生物大分子结构和功能特性认识的基础上,用生物信息学方法通过计算机模拟和计算来“预测”出这些信息或提供与之相关的辅助信息,已成为目前研究的热点。本研究借助生物信息学工具对plnB、plnC和plnD编码蛋白进行了理化性质、结构和功能的分析预测,发现plnB编码蛋白具有典型的组氨酸蛋白激酶的结构特征,plnC、plnD编码蛋白具有典型的感应调节蛋白的结构特征,推测plnBCD是L.paraplantarumL-XM1群体感应系统重要组分。在产细菌素乳酸菌中,研究得较为透彻的双组份系统,多数与自诱导肽组成群体感应系统,参与细菌群体感应,其HPK为HPK10亚族,RR为LytR亚族,L.paraplantarum L-XM1中plnBCD即属于此类。
调控乳杆菌Ⅱ类细菌素合成的群体感应系统的生物信息学研究虽然已有报道,但仅限于植物乳杆菌、副干酪乳杆菌等少数菌种,对L.paraplantarum群体感应系统生物信息学研究尚未见报道,L-XM1菌株群体感应系统组分的生物信息学分析对于丰富乳杆菌群体感应系统的序列特征、理化性质、空间结构等具有重要的理论意义和实际价值。目前,对乳杆菌群体感应系统组分的空间结构的研究相对较少,多数研究是通过借鉴其他菌,如金黄色葡萄球菌、大肠埃希菌等的双组份系统。类植物乳杆菌L-XM1PlnBCD的生物信息学研究,将为PlnBCD的功能研究提供理论依据,为新的群体感应调控系统的发现提供参考。
参考文献
[1]Maldonado-Barragán A,Caballero-Guerrero B,Lucena-PadrósH,et al.Induction of bacteriocin production by coculture iswidespread among plantaricin-producing Lactobacillus plantarumstrains with different regulatory operons[J].Food Microbiol,2013,33(1):40-47
[2]Axelsson L,Holck A.The genes involved in production of andimmunity to sakacin A,a bacteriocin from Lactobacillus sakeLb706[J].J Bacteriol,1995,177(8):2125-2137
[3]Eijsink V G H,Brurberg M B,Middelhoven P H,et al.Induction of bacteriocin production in Lactobacillus sake by asecreted peptide[J].J Bacteriol,1996,178(8):2232-2237
[4]Man L L,Meng X C,Zhao R H,et al.The role of plNC8 HK-plnD genes in bacteriocin production in LactobacillusplantarumKLDS1.0391[J].Int Dairy J,2014,34(2):267-274
[5]Cui Y,Qu X.Comparative analysis of two component signaltransduction systems of the Lactobacillus acidophilus group[J].Braz J Microbiol,2011,42(1):147-157
[6] 葛菁萍,赵晓龙,张岚,等.副干酪乳杆菌响应调节蛋白基因克隆与序列分析[J].微生物学杂志,2009,29(1):60-64





