
0、引言
内生真菌(Endophyticfungi)广泛存在于健康植物组织器官中,具有极其丰富的物种多样性和功能多样性。对纯培养物进行鉴定是内生真菌研究中一项必不缺少的基础性工作,但很多菌株在人工培养条件下缺乏(或不易形成)有性、无性繁殖结构,不产孢菌株占有相当高的比例;同时经常会分离到一些稀有、特殊(或新的分类单元)的菌株。据不完全统计,近20年来以内生真菌为标本命名、建立的新属至少有15个。
最近在橡胶树内生真菌资源挖掘中,还建立了一个子囊菌的新纲(Xolonomycetes),足以凸显内生真菌在全球真菌生物多样性组成中的特殊地位。从低等菌中的接合菌、卵菌到高等真菌中的子囊菌和担子菌都可作为植物内生菌。对于研究内生真菌天然产物、生态学功能等领域的专家来说,如何快速、准确鉴定物种(至少在属的水平)是一项既困难又重要的任务。笔者在2010年对中国科学院阜康荒漠生态站周边的荒漠盐生植物内生真菌资源进行了调查研究。本文就其中一株不产孢内生菌菌株的鉴定方法进行了研究和讨论,希望为今后内生真菌基础研究提供一些思路和参考。
1、材料与方法
1.1菌株材料
Tricharinastriispora(菌株保藏号:MUCL41297)由比利时菌种保藏中心(BCCM)馈赠;Tricharinaochroleuca(菌株保藏号:CBS238.85)购自荷兰皇家科学院真菌多样性研究中心(CBS)。小灌木-白刺属(Nitraria)荒漠植物内生真菌菌株BCS-1由作者实验室培养保存。
1.2内生真菌菌株BCS-1培养性状和显微形态观察
将内生真菌菌株BCS-1分别接种在马铃薯固体培养基(PDA)和麦芽粉琼脂培养基(MEA)平板上(直径9cm),20℃黑暗培养3-12d,观察菌落生长速度、培养性状;在光学显微镜(OlympusBX51)下观察和记录显微形态特征。
1.3内生真菌菌株BCS-1的多基因系统发育分析
菌株总基因组DNA提取采用MultisourceGenomicDNAMiniprep试剂盒(Axygen公司)。引物5.8SR(5’-TCGAT-GAAGAACGCAGCG-3’)和LR7(5’-TACTACCACCAA-GATCT-3’)扩增核糖体RNA(rRNA)28S大亚基(LSU)的部分序列;测序引物为LR0R(5’-ACCCGCTGAACTTAAGC-3’)、LR5(5’-TCCTGAGGGAAACTTCG-3’)和LR7。核糖体DNA内转录间隔区(ITS1-5.8S-ITS2)扩增采用引物ITS1F(5’-CTTGGTCATTTAGAGGAAGTAA-3’)和ITS4(5’-TCCTCCGCTTATTGATATGC-3’);RNA聚合酶第二亚基基因(RPB2)片段用RPB2-5f(5’-GAYGAYMGWGAT-CAYTTYGG-3’)和RPB2-7cr(5’-CCCATRGCTTGYTTRC-CCAT-3’)引物扩增。兼并碱基代码为:Y=T/C;M=A/C;W=T/A;R=A/G。PCR体系组成如下(50μl):5μl10×PCRbuffer、3μl25mmol/LMg2+
、4μl25mmol/LdNTP、0.5μl引物(50μmol/L)、5μlDNA模板(10~100ng)、0.3μlEx-Taqpolymerase和31.7μl去离子H2O。PCR扩增反应条件为:94℃预变性4min,每个循环为94℃40s、55℃退火1min(LSU基因调整为58℃、72℃延伸1.5min,最后72℃延伸10min,共35个循环。PCR产物纯化后送上海生工生物工程技术服务有限公司测序。3个菌株、3个基因共9条DNA序列提交至GenBank数据库。
用BLAST搜索工具获得每个基因多个匹配的序列记录,随后进行多序列比对(CLUSTALX软件)。用GENEDOC软件进行适当修正并去除序列两端不准确部分,其中RPB2序列的起始核苷酸以编码正确氨基酸为标准进行界定。用PHYLIP软件包、采用邻接法(neighborjoininganalysis,NJ)构建3个基因的系统发育树。进化树分支上的自展支持值(Bootstrap)由SEQBOOT、DNADIST、NEIGHBOR和CONSENSUS等相关命令获得,重复次数为1000次。
2、结果与分析
2.1内生真菌菌株BCS-1培养性状描述和显微形态特征
BCS-1菌株生长速度较快,20℃黑暗培养3d后,在PDA、MEA培养基上的菌落直径分别达到5.5cm和4.3cm(平均值)。培养1w后菌落即可覆盖整个培养皿,在PDA平板上菌落呈淡褐色,而在MEA平板上呈深褐色,培养后期菌落可形成不明显的“环形带”结构(图1)。
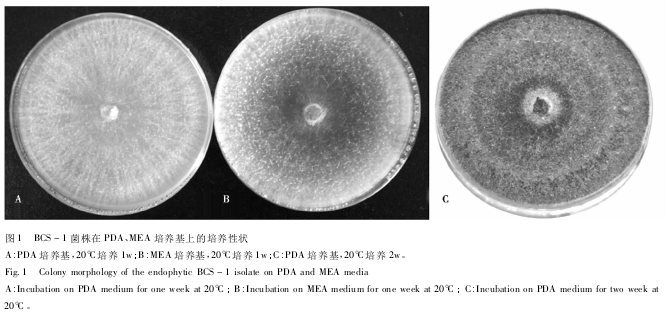
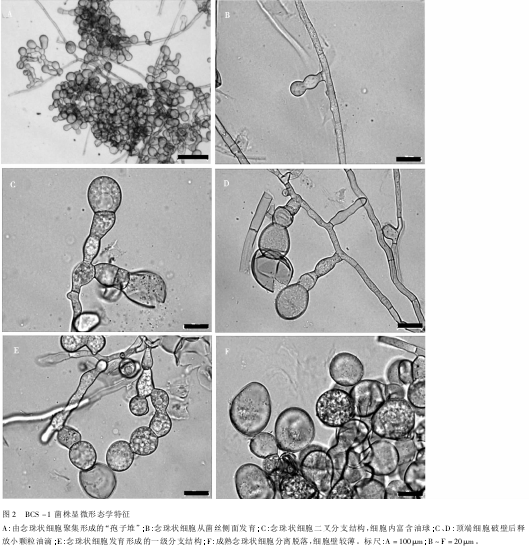
BCS-1菌株在人工培养基上不产生无性繁殖结构(如分生孢子等)。显微观察结果表明,菌丝有隔,宽5~10μm。在菌丝侧面(偶尔在菌丝顶端)近90°方向产生膨大的念珠状细胞(Monilioidcells),这些细胞通常聚集在一起,在菌落表明形成肉眼可见的“孢子堆”(Sporodochia)结构。念珠状细胞呈现多样化形态,有亚球形、柠檬形状、梨形或不规则形。多数细胞透明状,大小不一,富含油球,直径最大可达14μm。一般顶端细胞最大,呈淡褐色,细胞壁薄,油滴较小,细胞破壁后释放出来。念珠状细胞不分枝或二叉分支,少数可形成一级分支(图2)。
2.2基于ITS、RPB2和LSU的内生真菌菌株BCS-1多基因系统发育分析
首先以BCS-1菌株的ITS基因序列在GenBank数据库进行了BLAST搜索,表明BCS-1菌株与子囊菌火丝菌科(Py-ronemataceae)的地孔菌属(Geopora)真菌最接近,然而序列相似性只有90%,表明该菌株可能是稀有种(或潜在新种)。
LSU基因序列的BLAST结果显示前10个最佳匹配记录均隶属于火丝菌科。结合形态学特征并查阅相关文献,初步认为BCS-1菌株与火丝菌科的Tricharina属真菌的无性型(Asco-rhizoctonia)最为接近。为了验证鉴定的准确性,有必要研究该菌株的分子系统学。然而GenBank中有关Tricharina物种的基因数据还十分有限,因此本文选择2个Tricharina物种(标准菌株)为试验材料,以LSU、ITS和RPB2三个基因为标记,分析BCS-1菌株与Tricharina相关种及火丝菌科其它属之间的系统发育关系。
LSU基因系统发育分析表明BCS-1菌株与T.striispora、T.ochroleuca和T.praecox等遗传关系较近,在同一进化分支(自展支持率100%),而与模式种T.gilva遗传关系较远;RPB2和ITS序列的系统发育分析也支持了以上结果(自展支持率100%)。虽然LSU和ITS系统树表明Tricharina不是单谱系类群,与Geopora遗传关系最近,但以上结果确定BCS-1菌株属于Tricharina的一个无性型菌物(图3)。

2.3鉴定结果
根据Tricharina属无性型形态学特征的描述并结合分子鉴定结果,将BCS-1菌株鉴定为Ascorhizoctoniapraecox。根据“一种真菌一个名称”(Onefungus=Onename)命名法规,将BCS-1菌株最后命名为Tricharinapraecox。T.gilva是目前我国报道的唯一一个种,因此T.praecox还是我国的新记录种。
3、讨论
物种鉴定是真菌学研究中的一项基础性工作。菌物分类工作者通常对某一特定类群(科或属的水平)进行详细深入分析,如重要植物真菌病害(镰刀菌、炭疽菌等)或生态型类群(水生真菌、虫生真菌、暗色丝孢菌等),因此积累了丰富的研究材料(如文献资料、实物标本和基因数据等)。植物内生真菌鉴定工作则不同,同一种植物内生真菌的组成可能涵盖了担子菌和子囊菌中的多个科属,而且很多种类在培养基中不产生无性或有性繁殖结构。目前内生真菌的鉴定工作还比较粗放,如何快速、准确鉴定物种值得深入研究。
笔者认为不论内生真菌纯培养物产孢与否,核糖体转录间隔区ITS基因序列分析是内生真菌分子鉴定的不二选择。仅以经典形态学特征为依据进行鉴定费时且不完全可靠,尤其不适用于内生真菌鉴定工作。在本研究中,如果仅根据菌株培养性状和显微特征,可能误将其鉴定为丝核菌类担子菌如角担菌(Ceratobasidiumpseudocornigerum)和(Oliveorhizaana-pauxilla)等。StrobelGA等结合分子证据并比较标准菌株对一株产挥发性气体(碳氢化合物)的内生真菌进行了更正和重新命名。ITS序列已经被公认为是鉴定菌物的通用条形码。与其他基因数据相比较(RPB2、SSU等),GenBank数据库中真菌ITS序列信息是非常丰富的。尽管有研究认为在数据库中有许多序列注释错误或序列本身存在问题,但BLAST搜索结果一般可以给出有益的参考信息,将目的菌株的分类地位缩小范围,最后结合形态学特征(假设能观察到)鉴定到属或种。
当对内生真菌ITS基因序列进行BLAST搜索时,可能存在以下两种特殊情况:第一,与数据库记录匹配程度较低(85~90%)甚至更低,说明目的菌株可能是稀有种类(已知物种但数据库缺少该菌物的ITS序列)或者代表新的分类单元。本文中BCS-1菌株的BLAST结果属于前者;而以内生真菌为标本建立的新属如Muscodor、Edenia、Aurosphaeria和Mi-cronematobotrys等则属于后者。第二,某些真菌ITS基因序列的多个最佳匹配记录在分类地位不并不归在同一个属甚至相差甚远,可能有以下原因:(1)数据库部分匹配序列确属注释错误;(2)目的菌株可能不是单谱系类群(monophyleticgroup);(3)目的菌株与近缘种的ITS序列高度相似,表明此时ITS基因不是理想的条形码或者系统发育分析的优良标记。
以上情况表明基于真菌ITS序列的分子鉴定也有一定的局限性。为了解决这些问题,可以选择进化上更保守的核糖体非编码基因如SSU、LSU或编码基因或特殊分子标记综合分析,依据系谱一致性系统发生“种”识别(GCPSR)原理科学界定物种。如RPB2和翻译延长因子(EF-1α)广泛应用于子囊菌种水平的研究;链格孢过敏元基因Alta1能很好地解决链格孢菌(Alternaria)的系统发育。随着真菌全基因组测序的快速发展,利用生物信息学手段可以将一些进化速率适度的单拷贝编码蛋白基因开发成新的分子标记。
值得注意的是:传统菌物分类通常需要获得独立来源的多个菌株进行分类学特征的调查。然而单个菌株(Singletons)或单个操作分类单元(OTUs)在内生真菌多样性研究中十分普遍;经验还表明很多具有强活性的内生真菌菌株往往只分离到一份标本。在此情况下,通过多个基因的系统学分析并结合几个关键形态学特征进行鉴定是可行的。
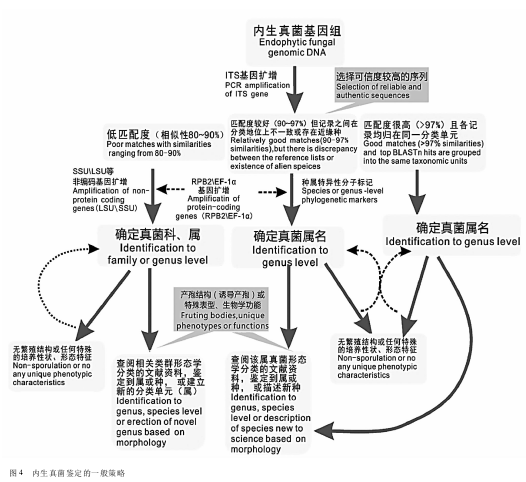
结合本文的研究实例并综合以上讨论,我们归纳了内生真菌菌种鉴定的一般策略:简言之,就是“先分子后形态”(图4)。虽然内生真菌种类繁多,特殊无孢内生菌也相当普遍,鉴定工作给非菌物分类学专家带来一定的挑战性。但随着Tri-chOKEY2、FUSARIUM-IDv.1.0、UNITE、INSDC和真菌生命之树等真菌序列数据库的不断完善,再经过实践积累经验,科学、正确地鉴定植物内生真菌是完全可以做到的。
致谢:感谢中国科学院微生物研究所真菌学国家重点实验室庄文颖教授提供许多原始文献资料!感谢中国科学院新疆生态与地理研究所李彦教授在植物采样中给予的热情帮助!
参考文献:
[1]Guo LD,Huang GR,Wang Y,et al. Molecular identification of whitemorphotype strains of endophytic fungi from Pinus tabulaeformis[J]. Myco-logical Research,2003,107( 6) : 680 - 688.
[2]Gazis R,Miadlikowska J,Lutzoni F,et al. Culture - based study of en-dophytes associated with rubber trees in Peru reveals a new class of Pezizo-mycotina: Xylonomycetes [J]. Molecular Phylogenetics and Evolution,2013,65( 1) : 294 - 304.
[3]Vilgalys R,Hester M. Rapid genetic identification and mapping of enzy-matically amplified ribosomal DNA from several Cryptococcus species [J].The Journal of Bacteriology,1990,172( 8) : 4238 - 4246.
[4]Gardes M,Bruns T D. ITS primers with enhanced specificity for basidio-mycetes - application to identification of mycorrhizae and rusts[J]. Molec-ular Ecology,1993,2( 2) : 113 - 118.
[5]White TJ,Bruns S,Lee S,et al. Amplification and direct sequencing offungal ribosomal RNA genes for phylogenetics[C]/ / Innis MA,GelfandDH,Sninsky JJ,White TL. PCR protocols: a guide to methods and applica-tions. San Diego: Academic Press,1990: 315 - 322.
[6]Liu Y,Whelen S,Hall BD. Phylogenetic relationships among ascomyce-tes: evidence from an RNA polymerase Ⅱ subunit[J]. Molecular Biologyand Evolution,1999,16( 12) : 1799 - 1808.
[7]Yang CS,Korf RP. Ascorhizoctonia gen. nov. and Complexipes emend. ,two genera for anamorphs of species assigned to Tricharina ( Discomycetes)[J]. Mycotaxon,1985,23: 457 -481.
[8]Yang CS,Korf R P. A monograph of the genus Tricharina and of a newsegregate genus,Wilcoxina ( Pezizales) [J]. Mycotaxon,1985,24: 467- 531.
[9]Barrera VA,Romero AI. Tricharina striispora from Argentina and thefinding of its anamorph,Ascorhizoctonia [J]. Mycotaxon,2001,77: 3184
[10]Zhuang W Y,Korf R P. Some new species and new records of Disco-mycetes in China. Ⅲ[J]. Mycotaxon,1989,35: 297 - 312.
[11]Moore RT. The genera of Rhizoctonia - like fungi: Ascorhizoctonia,Cer-atorhiza gen. nov. ,Epulorhiza gen. nov. ,Moniliopsis,and Rhizoctonia [J].Mycotaxon,1987,29: 91 - 99.
[12]Roberts P. Rhizoctonia - forming fungi: a taxonomic guide[M]. UK:Kew Publishing,1999.
[13]Strobel GA,Tomsheck A,Geary B,et al. Endophyte strain NRRL50072 producing volatile organics is a species of Ascocoryne [J]. Mycolo-gy,2010,1( 3) : 187 - 194.
[14]Schoch CL,Seifert KA,Huhndorf S,et al. Nuclear ribosomal internaltranscribed spacer ( ITS) region as a universal DNA barcode marker for fun-gi[J]. Proceedings of the National Academy of Sciences ( USA ) ,2012,109( 16) : 6241 - 6246.
[15]Hawksworth DL.“Misidentifications”in fungal DNA sequence data-banks[J]. New Phytologist,2004,161: 13 - 15.
[16]Holst - Jensen A,Vralstad T,Schumacher T. On reliability[J]. NewPhytologist,2004,161: 11 - 13.
[17]Guo LD. Molecular diversity and identification of endophytic fungi[C]/ / Gherbawy Y,Voigt K. Molecular identification of fungi. Berlin Hei-delberg: Springer - Verlag,2012: 277 - 296.
[18]Yuan ZL,Rao LB,Chen YC,et al. From pattern to process: speciesand functional diversity in fungal endophytes of Abies beshanzuensis [J].Fungal Biology,2011,115( 3) : 197 - 213.
[19]Worapong J,Strobel GA,Ford E,et al. Muscodor albus anam. gen. etsp. nov. ,an endophyte from Cinnamomum zeylanicum [J]. Mycotaxon,2001,79: 67 - 79.
[20]González MC,Anaya AL,Glenn AE,et al. A new endophytic ascomy-cete from El Eden Ecological Reserve,Quintana Roo,Mexico [J]. Mycot-axon,2007,101: 251 - 260.
[21]Lee SJ,Strobel GA,Eisenman K,et al. Aurosphaeria,a novel coelomy-cetous genus[J]. Mycotaxon,2009,107: 463 - 472.
[22]Sun X,Guo L D. Micronematobotrys,a new genus and its phylogeneticplacement based on rDNA sequence analyses [J]. Mycological Progress,2010,9( 4) : 567 - 574 .
[23]Zhang CL,Wang GP,Mao LJ,et al. Muscodor fengyangensissp. nov. from southeast China: morphology,physiology and production of vol-85





