摘要:不依赖于纯培养的分子生物技术已广泛应用于环境微生物的研究。综述了近年来在荧光原位杂交技术 (Fluorescent in situ hybridization, FISH) 和聚合酶链式反应技术 (Polymerasechainreaction, PCR) 基础上发展起来的几种新分子生物技术的基本原理, 包括FISH-MAR、FISH-microelectrodes、Clone-FISH、SIP、PCR-DGGE-cloning-sequencing、PCR-T-RFLP、Real-time fluorescent quantitative PCR和RT-PCR, 以及其在污水生物处理系统内硝化菌群研究领域中的应用现状。通过这些分子生物技术, 可以识别污水生物处理系统内的硝化菌群;建立硝化菌群动态变化与工艺运行参数之间的相关关系;从微生物学角度对系统运行状态给予最直接、最可靠的分析与证明, 为污水生物处理系统的长期稳定运行奠定理论基础。最后对该领域的未来发展进行了展望。
关键词:硝化菌群; 分子生物技术; 荧光原位杂交; 聚合酶链式反应; 稳定同位素探测; 污水生物处理;
Application of Molecular Biological Techniques for Analyzing Nitrifier Communities in Wastewater Treatment
Abstract:Molecular biological techniques have provided a cultivation-independent means for the identification of environmental microorganisms.The molecular biological techniques based on fluorescent in situ hybridization (FISH) and polymerase chain reaction (PCR) were introduced, including FISH-MAR, FISH-microelectrodes, Clone-FISH, SIP, PCRDGGE-cloning-sequencing, PCR-T-RFLP, real-time fluorescent quantitative PCR and RT-PCR.The mechanisms and applications of these techniques in the investigation of nitrifier communities in wastewater treatment systems were studied.By using these techniques, the nitrifier communities could be identified in the systems, and the correlation between the dynamic variation in the nitrifier communities and the operating parameters of the systems could be established, and the real state of the system operation could also be analyzed and proved directly and reliably.The results from this study provide theoretical supports for the long-range stable operation of the wastewater treatment systems, and this paper puts forward some suggestions for the future application of those techniques in this field.
Keyword:nitrifier community;molecular biological technique;fluorescent in situ hybridization (FISH) ;polymerase chain reaction (PCR) ;stable isotope probing (SIP) ;biological wastewater treatment;
污水生物处理系统内的微生物学研究是污水生物处理中最基本、最重要的内容, 是污水生物处理系统高效稳定运行的保障。传统的微生物学研究方法过于依赖纯培养技术来进行菌种分离, 该方法对于研究污水生物处理系统内的微生物有着严重的缺陷。首先, 污水生物处理系统内的微生物菌群是由多种微生物构成、具有一定空间分布的聚集体。这些微生物相互依存、相互竞争, 具有复杂的种群关系。当进行菌种分离培养后, 将无法获取自然条件下微生物菌群结构与空间分布的信息。其次, 由于微生物的基因表达和代谢活动受外界环境因素的影响较大, 菌种的分离培养将会改变微生物的生理特性和生化特性, 从而降低对实际工程实践的指导意义。此外, 纯培养需要较长的时间, 而且有些菌种 (如亚硝酸盐氧化菌中的Nitrospira.) 目前还无法通过纯培养技术实现纯种分离。
近年来, 分子生物技术的快速发展为污水生物处理系统内微生物研究提供了新的分析方法和手段。污水生物脱氮包括硝化和反硝化两大阶段。其中硝化菌群是硝化阶段的功能微生物, 包括将NH4+-N氧化为NO2--N的氨氧化菌 (Ammonia oxidizing bacteria, AOB) 和将NO2--N氧化为NO3--N的亚硝酸盐氧化菌 (Nitrite oxidizing bacteria, NOB) .AOB和NOB在污水生物处理系统内的生物活性和菌群分布的稳定性, 是保证污水生物脱氮系统稳定运行的关键因素之一。因此, 采用分子生物技术对不同工艺、水质条件下的AOB和NOB的菌群结构、活性等进行研究分析, 能够为污水生物脱氮系统的长期稳定运行提供有力保障。目前, 以FISH和PCR为代表的分子生物技术已广泛应用于污水生物处理系统内硝化菌群结构和生物活性的研究中, 国外已有大量的相关文献报道, 但在国内还处于起步研究阶段。
1、基于FISH技术的分析方法
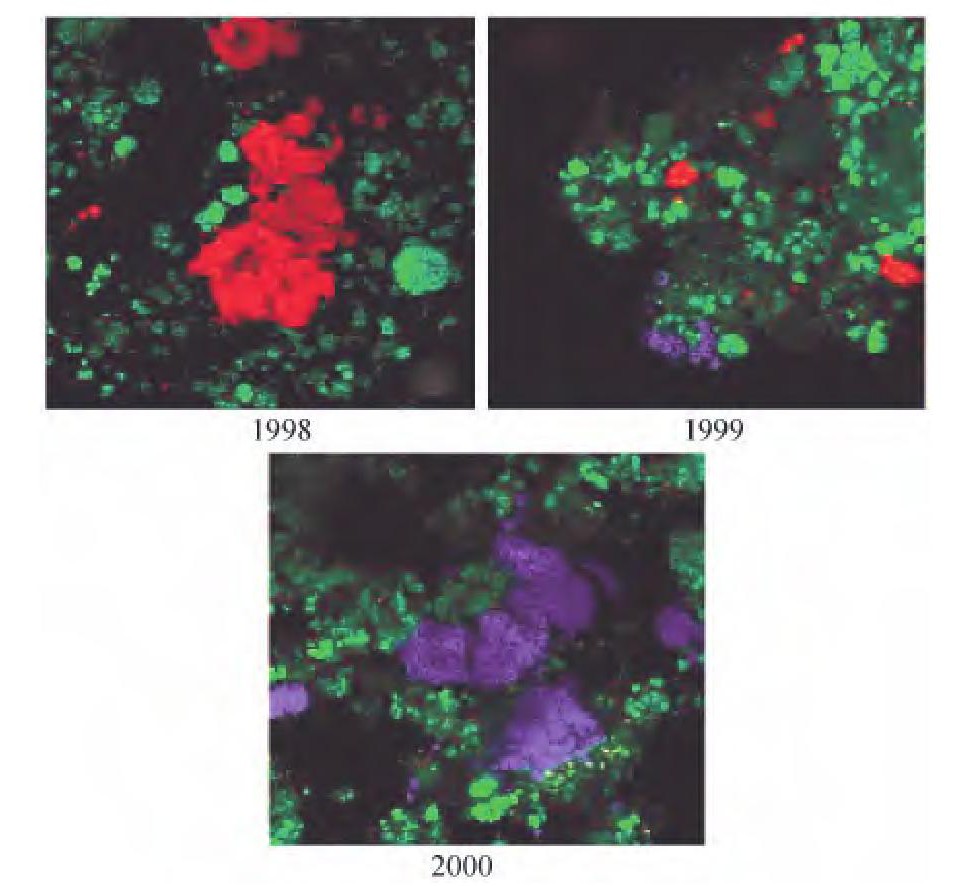
FISH技术是根据目标微生物16S r RNA基因中的特异性序列, 设计相应的、带有荧光染料的寡核苷酸探针, 按照碱基序列互补原则, 将探针直接原位杂交到目的基因上, 杂交成功的探针就会产生荧光信号。然后通过共聚焦激光扫描显微镜 (Confocal laser scanning microscopy, CLSM) 或者荧光显微镜对目的基因进行定性和相对定量分析。典型的FISH图片如图1所示。通过选取系统发育中保守性不同的特异性序列, 就可在种、属水平上对微生物进行检测。因此, FISH技术还可用于研究微生物种群的结构动态变化。自从Amann等最初将FISH应用于环境微生物的原位分析[1]以来, 该方法已被用于污水生物处理系统内丝状菌[3]、聚磷菌[3]、硝化菌[4~5]、厌氧氨氧化菌[6]等的原位检测。近几年, 研究者在普通FISH的基础上发展了几种更为先进的分子生物学分析方法, 用于对污水生物处理系统内硝化菌群的研究:FISH-MAR (Microautoradiography, 显微放射自显影) 技术、FISH-microelectrodes (Microelectrodes, 微电极) 技术和Clone-FISH技术。
1.1 FISH-MAR
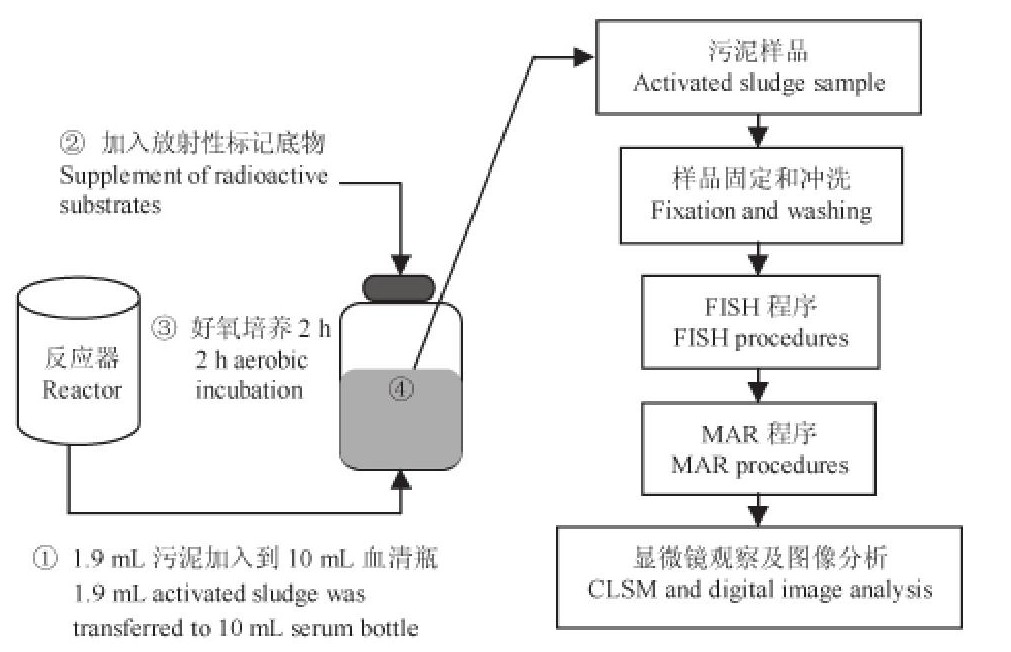
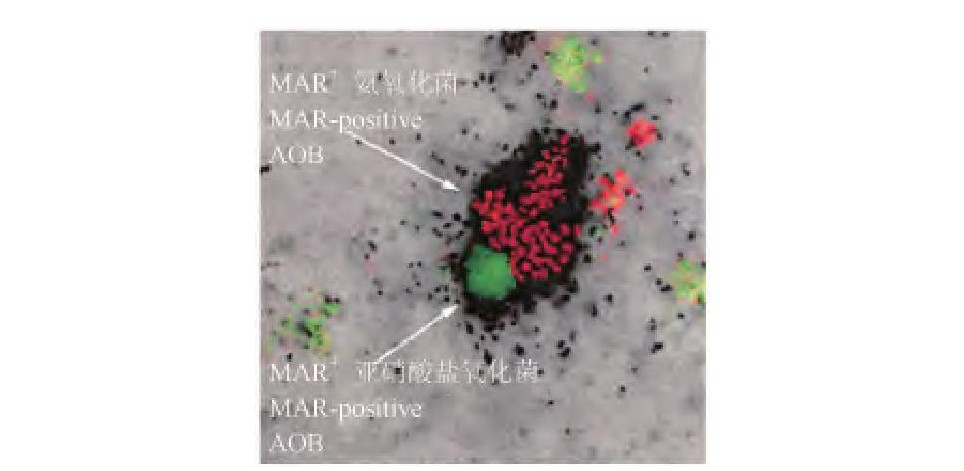
单独采用FISH技术, 并不能获取微生物种群的生物代谢活性和生理功能的信息。而单独采用MAR技术, 却无法对微生物种群的组成及其分布加以识别。Lee等首先将这两种技术结合, 建立了FISH-MAR方法[7].该方法能够以显微照片的形式, 清晰、直观地显示出复杂的微生物群体的菌群结构及其原位空间分布情况, 而且同时能够反映出不同微生物种群的生物代谢活性。该方法的基本流程[7]可总结如图2, 其核心思想是将待分析的微生物样本放置在经放射性物质标记的有机或无机底物中进行培养, 其中能够吸收放射性底物的细胞将在放射自显影的显微图片上呈现黑色的影像 (MAR阳性, MAR+) , 而没有吸收放射性底物的细胞则没有影像显示。对于异养型微生物通常采用14C标记的有机底物, 如14C-乙酸;对于无机自养型硝化细菌, 通常采用Na H14CO3.图3为一典型的Fish-MAR显微图片[8], 图中的黑色细胞为吸收了Na H14CO3的化能自养型细菌, 红色细胞为通过FISH探针检测到的AOB, 二者的细胞数比例表示化能自养型细菌中AOB所占的比例;绿色细胞是FISH探针检测到的NOB.
Okabe课题组采用FISH-MAR方法对自养型硝化生物膜中的微生物代谢进行了研究。研究者首先将自养型硝化生物膜在含有Na H14CO3的底物中培养6 h, 其中自养硝化菌呈MAR+, 异养菌呈MAR-.再将含MAR+硝化菌和MAR-异养菌的生物膜在仅含有NH4+的溶液中培养10 d, 结果发现多数异养菌呈现MAR+, 说明异养菌能够利用14C标记的硝化菌细胞裂解产物或代谢产物, 从而揭示了在碳源受限的自养型硝化生物膜中微生物在底物利用上的相互作用关系[8~9].该研究指出在碳源受限的硝化生物膜中硝化菌与异养菌各占50%, 其中α-Proteobacteria 23%、γ-Proteobacteria 13%、绿色非硫细菌9%、Cytophaga Flavobacterium Bacteroides 2%、不能确定的微生物3%.Gieseke等也采用FISH-MAR方法对生物膜中的硝化及碳源同化进行了原位分析, 进而研究不同微生物种群之间碳的转化及同化[10].
虽然FISH-MAR是一种研究微生物代谢及微生物种群结构的有效方法, 但该方法不适于检测目标微生物含量较低的样品。由于其检测手段取决于目标微生物的荧光信号和放射自显影, 所以当活性污泥中目标微生物的细胞数低于104cell/m L时, 由于目标微生物的r RNA含量过低, 将导致无法区分目标微生物与背景的荧光信号, 从而造成无法检测。所以, 当硝化菌群没有成为系统的优势菌群时FISH-MAR将难以取得检测结果。
1.2 FISH-microelectrodes
利用微电极技术能够检测活性污泥絮体或生物膜内部微小生境的环境要素, 并通过这些环境要素的变化反映微生物代谢活性。FISH-microelectrodes方法将FISH技术和微电极技术二者有机地结合在一起, 该方法能够建立微生物原位空间分布、代谢活性与环境要素之间的相互关系[11].其中微电极能够测量的参数主要有O2、H2S、NO3-、NO2-、NH4+和p H等。
目前FISH-microelectrodes技术主要集中应用在对生物膜中微生物种群的研究。其中对于具有硝化功能的生物膜, 该方法能够确定沿着生物膜厚度的垂直方向硝化菌群的原位分布及代谢活性[12~13].Okabe课题组采用该方法研究了自养型硝化生物膜和处理市政污水的生物膜中AOB和NOB的原位空间分布[14].结果表明这两种生物膜系统中Nitrosomonas是优势AOB, Nitrospira-like是优势NOB, 没有检测到Nitrobacter.Nitrosomonas遍布整个生物膜, 而Nitrospira-like以细胞聚集体的形式分布在AOB周围。该生物膜的氨氧化区主要集中在生物膜的外层, 亚硝酸盐氧化区位于氨氧化区下面。采用FISH-microelectrodes方法也能够很好地评价生物扩增和生物刺激对生物膜系统硝化功能的影响, 其中FISH用于分析硝化菌群时间上的动态变化, 微电极测量反映原位硝化活性[15].生物扩增是将富集培养的硝化菌投加于系统中, 该方法能够提高系统内硝化菌活性及丰度, 从而快速启动系统的硝化功能。生物刺激是通过提高或投加某种底物, 从而促进或抑制微生物的生长。有研究者试图提高硝化系统启动阶段的底物中NH4+和NO2-浓度, 想藉此来刺激AOB和NOB的生长, 但实验结果表明该操作并没有加快系统硝化功能的启动, 其中NO2-氧化过程并没有受到影响, 但是AOB增殖却到了受抑制。也有研究者采用FISH-microelectrodes方法研究活性污泥系统中反硝化作用与硫酸盐还原作用的相互影响, 反硝化菌与硫酸盐还原菌对有机碳源的竞争[16].
1.3 Clone-FISH
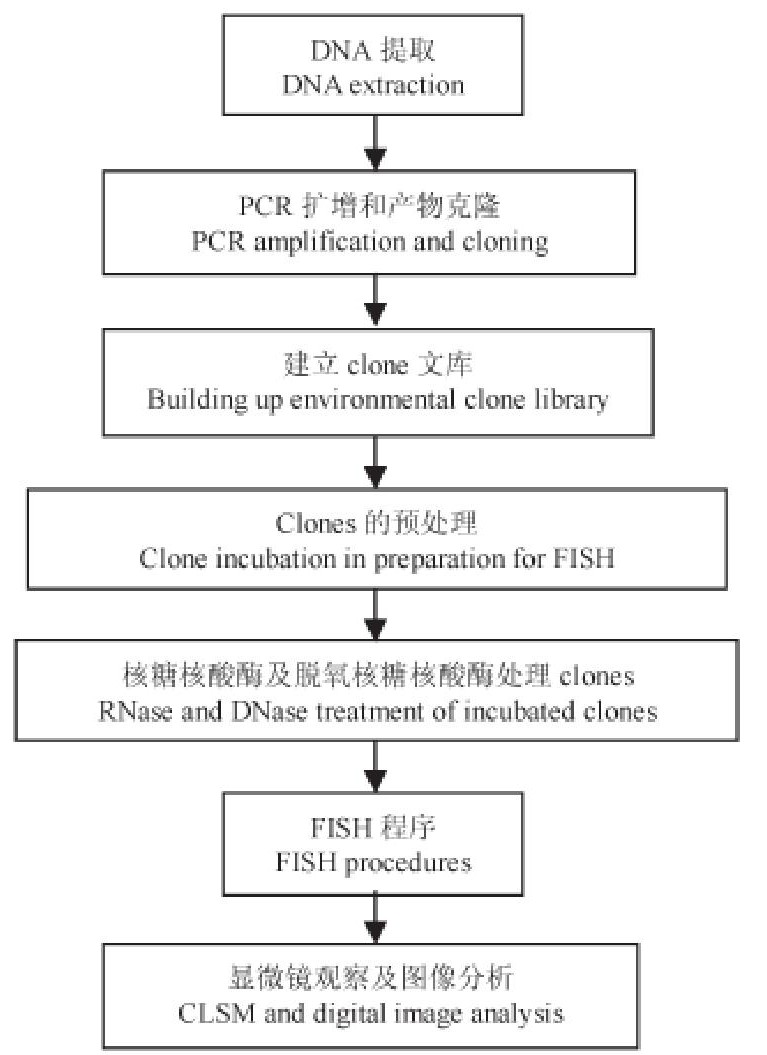
由于含有目的基因片段的克隆子 (Clones) 同样适用于FISH检测, 因此诞生了Clone-FISH检测方法, 即采用FISH筛选阳性Clones或检验探针的有效性[17].Clone-FISH的实验程序总结如图4.
目前将Clone-FISH方法直接用于硝化菌群的研究鲜有报道。该方法的应用主要体现在两方面:一是检验针对硝化菌群的探针的特异性和有效性[18].探针是FISH应用的前提和基础, 以16S r RNA克隆子代替纯培养的细胞作为阳性对照检验探针的特异性及优化杂交条件, 尤其是当某些硝化菌群无法进行纯培养时, Clone-FISH解决了检验探针特异性的难题。二是用FISH筛选克隆文库中的阳性克隆子, 检验目的基因片段是否成功转化[19].
2、基于PCR技术的分析方法
PCR是一种选择性体外扩增DNA或RNA片段的分子生物技术。PCR技术解决了从环境样品中提取的硝化菌DNA含量通常过低而不能进行相关分析的难题。目前多种基于PCR技术的衍生技术已经广泛应用于对污水生物处理系统内硝化菌群的研究, 并取得了大量研究成果, 深化了人们对硝化菌群的认识。
2.1 amo A与16S r RNA基因
将PCR技术应用于对硝化菌群的研究中, 首先要选取一段具有硝化菌群系统进化标记特征的DNA序列作为目的分析序列, 然后根据该序列的保守区设计相应的探针和引物。其中, 对AOB进行研究分析时所选取的分子标记基因一般是AOB的16S r RNA基因和amo A.
对16S r RNA基因的研究给整个分子生物学研究领域带来了革命性的影响, 并扩展了人们对微生物系统发育的认知。16S r RNA基因具有高度的保守性, 但不同微生物的16S r RNA基因的某些区域也存在着差异。这些差异可将微生物分为古细菌和细菌两大类, 并能在分类学上对微生物进行更进一步的研究。伯杰氏系统细菌学手册的第2版中对微生物的分类和命名就是基于对不同微生物16S r RNA基因差异的分析。同样不同种AOB的16S r RNA基因在结构上也具有差异性。依据16S r RNA基因的保守性和差异性, 可确定不同种AOB的系统发育的相关性和进化距离。所有种类AOB的基因组中只含有一条16S r RNA基因。
amo A是编码氨单加氧酶 (Ammonia monooxygenase, AMO) 的基因, AMO是AOB所特有的一种胞内酶。编码AMO的基因簇中至少含有3个基因:amo A、amo B和amo C[20].其中amo A的基因产物包含AMO的活性位点, 可以催化铵氧化成羟胺, 并为AOB生长提供能量。不同种类的AOB含有不同拷贝数的amo A, 一般AOB中含有2~3个拷贝数的amo A.Nitrosomonas europaea中含有2个拷贝的amo A[21], 这2个拷贝的amo A的DNA序列上仅有一个核苷酸不同。Hommes通过突变实验证明这2个拷贝的amo A都是功能基因, 但是它们在细胞内的功能和地位却不尽相同[21].
Purkhold[22]、Aakra[23]和Nicolaisen[24]等对不同AOB菌株的amo A和16S r RNA基因进行测序和系统发育树分析后, 发现大部分AOB在分别基于amo A和16S r RNA基因的系统发育树上的分类有着高度的相似性。他们同时也发现, 尽管对AOB的16S r RNA基因和amo A基因进行定性分析时都可以反映出环境中AOB的种类, 但由于不同种AOB之间的16S r RNA基因序列的高度相似性和该类型引物的非特异性[25], 限制了16S r RNA基因类引物对AOB菌群系统发育树分析的精确度。而尽管amo A不是制定系统发育树的因子, 但由于amo A只存在于AOB中, 而且不同种AOB的amo A序列差异度超过其16S r RNA基因的序列差异度, 使得amo A类引物的扩增特异性更强, 因此对AOB菌群遗传差异的分辨能力更高[25,27], 从而能够更精确地分辩出AOB的种属。但无论是amo A类引物, 还是16S r RNA基因类引物, 目前都只针对β-变形菌纲的自养型AOB, 因此在使用这两类引物对AOB进行分析研究时都会低估环境样品中AOB菌群的多样性。
2.2 PCR-DGGE-cloning-sequencing
PCR-DGGE (PCR-denaturing gradient gel electrophoresis, PCR-变性梯度凝胶电泳) 技术是将PCR扩增产物加入到含有线性梯度变性剂 (尿素和甲酰胺) 的聚丙烯酰胺凝胶中进行电泳。PCR扩增出来的DNA片段长度虽然相同, 但是它们的碱基序列却不尽相同。在电泳过程中, 序列不同的DNA片段就会在各自相应的变性剂浓度下变性, 导致其在聚丙烯酰胺凝胶中电泳速度的急剧下降, 从而停留在其相应的变性剂梯度位置上。再通过染色处理, 聚丙烯酰胺凝胶上就会呈现出不同的DNA谱带, 每条DNA谱带就代表了某一序列的DNA片段。为了避免单链DNA、异源双链核酸分子[24]和探针特异性等对分析结果的影响, 研究者通常会在PCR-DGGE后进行克隆 (Cloning) 和测序 (Sequencing) 来进一步确定DNA序列, 进而揭示样品中微生物的菌群结构。这就形成了PCR-DGGE-cloning-sequencing技术。
Cole[28]和Lu x my[29]利用该技术证实了生物膜系统内的微生物多样性要远远大于传统活性污泥法。Ebie[30]和Limpiyakorn[31]分别利用该技术对不同污水处理系统内的硝化菌群进行了研究, 研究结果表明, 尽管监测过程中系统内的AOB含量保持不变, 但是由于受到进水水质和温度等外界条件变化的影响, 污水生物处理系统内的AOB种群结构发生了巨大的变化。Mota研究间歇曝气对硝化细菌菌群结构以及脱氮效率的影响时发现, 好氧时间与缺氧时间的长短会影响硝化菌群结构[32];与传统理论不同的是, 研究者发现脱氮效率与特定的AOB种群含量及AOB的菌群多样性无关。Sundberg在对处理垃圾渗滤液的坡面漫流系统内AOB菌群结构进行了研究, 研究发现该系统内的AOB菌群结构并没有季节性的变化, 但是在系统不同的处理单元内存在不同种类的AOB菌群[33].
通过PCR-DGGE-cloning-sequencing技术能够揭示污水生物处理系统内的硝化菌群结构及其相对含量的变化。但是聚丙烯酰胺凝胶条带中的硝化菌DNA片段通常在500 bp以下, 而当从该片断中获得的序列少于已知序列的85%时, 就不能准确地在系统发育树上将该硝化菌进行分类, 从而无法进一步得到该硝化菌的相关信息。而且由于配制的聚丙烯酰胺凝胶浓度的差异和该方法较低的灵敏度, 限制了该技术的重现性以及对含量较少的微生物种群的检测能力。
2.3 PCR-T-RFLP
PCR-T-RFLP (PCR-terminal restriction fragment length polymorphism, PCR-末端限制性片段长度多态性) 技术是对PCR扩增过程中所使用的引物一端用荧光物质进行标记。当用该引物对样品DNA进行PCR扩增后, PCR的扩增产物就带有了荧光标记。然后选择适当的限制性内切酶对PCR产物进行消化, 就可产生不同长度的限制性片段。传统的T-RFLP技术通常只使用一条带有荧光染料的引物和一种限制性内切酶。随着该技术的改进, 有的研究者会同时使用两条带有不同荧光染料的引物进行PCR扩增, 从而提高目标微生物的分类水平。最后利用DNA自动测序仪和带有其它荧光染料标记的内标物对消化产物的长度进行分析, 并获得峰值图。由于每种微生物带荧光标记的限制性片段长度唯一, 所以峰值图中每一个峰至少代表一种微生物, 每个峰面积与峰总面积的比值代表这种微生物的相对含量[34].相对于PCR-DGGE技术, PCR-T-RFLP技术能在相对较短的时间内对大量样品的菌群结构进行分析。PCR-T-RFLP技术还具有较好的重现性和更高的灵敏度, 从而该技术能够对样品中含量较少的微生物进行研究。
自Hiraishi 2000年首次报道利用PCR-T-RFLP技术分析活性污泥系统内硝化菌群结构[35]以来, 由于该技术在分析微生物群落动态变化上的优势, 国内外诸多学者也相继采用了该技术对不同环境下的硝化菌群进行了研究。Egli采用该技术对启动阶段的短程硝化反应器的硝化菌群结构进行了研究, 发现反应器内的AOB的种群多样性逐渐降低, 而且在不同的温度、p H和SRT条件下AOB的菌群结构是不同的[36].Park利用该技术探讨了DO对活性污泥中AOB菌群结构的影响[37];并研究了奥贝尔氧化沟内AOB的菌群结构[38], 发现由于氧化沟内好氧、缺氧环境的交替使得该系统内Nitrosospira-like的含量大大超出其他污水处理厂, 而且Nitrosospira-like的存在与SND现象有着较好的相关性。
由于PCR-T-RFLP技术要对PCR产物进行纯化和脱盐处理, 所以较为耗时, 费用较高。该技术方法中采用的荧光染料种类以及内标物的长度的不同可能会引起理论与实际片段长度的差异。这些差异虽然可以通过限定扩增片段的长度来解决, 但是与此同时也会使得某些片段得到额外的扩增。此外由于酶切不能人为的控制, 某些微生物物种可能也不存在所需要的酶切位点, 这些因素都会影响该技术的精确性。
2.4 Real-time fl uorescent quantitative PCR
获得样品中硝化细菌数量的信息对于进一步深入研究硝化菌群在污水生物处理过程中的作用是至关重要的。目前, 国内外普遍采用实时荧光定量PCR (Real-time fl uorescent quantitative PCR) 技术对污水生物处理系统内硝化菌群进行定量分析。与传统的末端监测PCR技术不同, 实时荧光定量PCR技术是在PCR反应体系中加入荧光基团, 利用仪器对整个PCR反应过程中的荧光信号进行实时采集, 随着反应的进行, 监测到的荧光信号可以绘制成一条曲线并获得其阀值 (Cycle threshold, Ct) , 该Ct值与环境样品中所含的目标DNA成反比。然后将该Ct值与标准曲线进行比较, 就可以得出该样品中目标DNA的含量。
目前, 实时荧光定量PCR普遍采用两种荧光物质来进行定量分析。一种是带有荧光染料的Taq Man探针, 一种是SYBR Green染料。除了正、反向两条引物外, 由于一条特异性探针的引入, 使得Taq Man探针法相对于SYBR Green染料法, 其对目标DNA的扩增特异性更强。而SYBR Green染料法的费用较为低廉, 而且该方法中所使用的引物较Taq Man探针法更为广泛。无论使用哪种方法首先都要建立目标DNA含量与Ct值之间的标准曲线。目前的研究者大都通过等梯度稀释纯培养目标菌的基因组DNA, 或者PCR扩增片段, 或者含有目标DNA片段的质粒来建立标准曲线。其中利用质粒建立的标准曲线的重现性最好。根据公式1可计算出已知浓度的DNA溶液中所含目标DNA的拷贝数。PCR扩增的效率可以根据建立的标准曲线的斜率得出, 正常的扩增效率应该在80%~85%之间, R2应大于0.95.
式中, MW=DNA碱基对数 (bp) ×660 daltons/bp;Daltons-道尔顿, 质量单位, 1克约为6×1023daltons.
与传统的PCR技术和FISH技术相比, 由于实时荧光定量PCR具有灵敏度高、重现性好、同时处理的样品量大等特点, 研究者已经利用实时定量PCR技术考察了不同的污水处理工艺、运行参数对硝化菌群数量、结构[39]的影响以及硝化细菌的数量和污水处理厂脱氮效果之间的关系[40~41].但实时荧光定量PCR过于依赖DNA的提取效率[42]和探针、引物的特异性, 在很大程度上影响了该技术对硝化细菌定量的准确性。Harms[43]和Robinson[44]利用实时荧光定量PCR技术对活性污泥法污水处理厂的AOB和NOB菌群进行定量研究时发现, 相对于AOB在污水生物处理系统内的重要作用, 其定量结果偏低。有研究者通过添加已知含量的内标物来评估DNA提取和纯化的效率[45], 从而能够更为准确地对污水生物处理系统中硝化菌群进行定量分析。而将实时荧光定量PCR与FISH等其他分子生物学技术联合使用能更好地反映出硝化细菌的数量[46], 避免单一技术所引起分析误差。
2.5 RT-PCR
RT-PCR (Reverse transcripatase PCR, 逆转录PCR) 技术将RNA的反转录技术和c DNA的PCR扩增技术进行结合。首先模板RNA经反转录酶的作用合成c DNA;再以c DNA为模板, 扩增合成目的片段。作为模板的RNA可以是总RNA、m RNA或体外转录的RNA产物。无论使用何种RNA, 关键是确保提取的RNA中无RNA酶和基因组DNA的污染。
Terahara联合应用RT-PCR技术和T-R FLP技术对启动阶段的污水处理厂内AOB的16S r DNA和16S r RNA的研究表明, AOB胞内的16S r RNA要比16S r DNA优先增长, 并且外界环境条件的变化将会影响AOB菌群16S r RNA的多样性[47].尽管amo A是AMO的活性位点, 但是采用普通的PCR技术对amo A基因进行分析并不能揭示amo A基因乃至AOB的活性。由于amo A m RNA在AOB体内更新速度快, 其半衰期一般仅有几分钟, 所以通过RT-PCR技术对AOB体内的amo A m RNA进行分析, 能够揭示环境因素对AOB活性的影响, 从而及时获得信息来优化污水处理工艺。Aoi通过RT-PCR技术对生物膜反应器中的amo A m RNA的研究表明, 氨氮浓度和p H是影响amo A m RNA转录的重要因素, 其中当p H较低时, 将抑制amo A m RNA的转录[48].Araki也利用Real-time PCR和RT-PCR技术对活性污泥系统不同反应阶段的amo A m RNA的转录水平进行了分析[49].
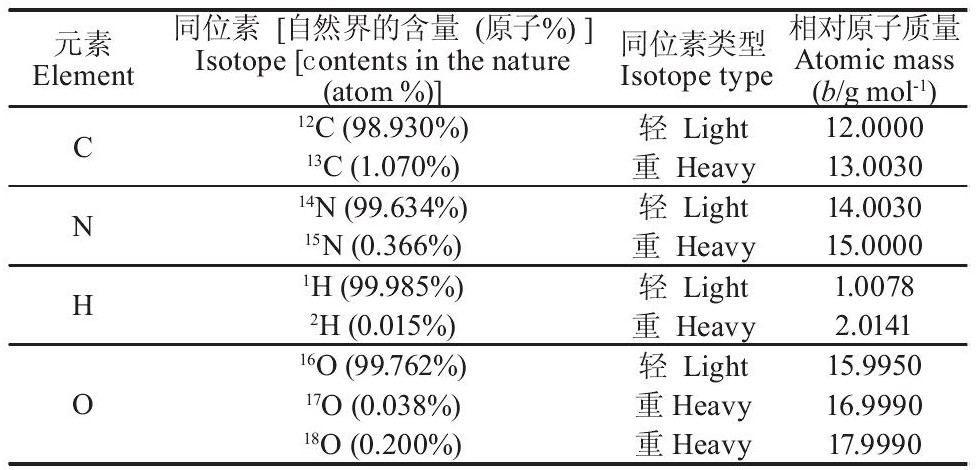
3、SIP
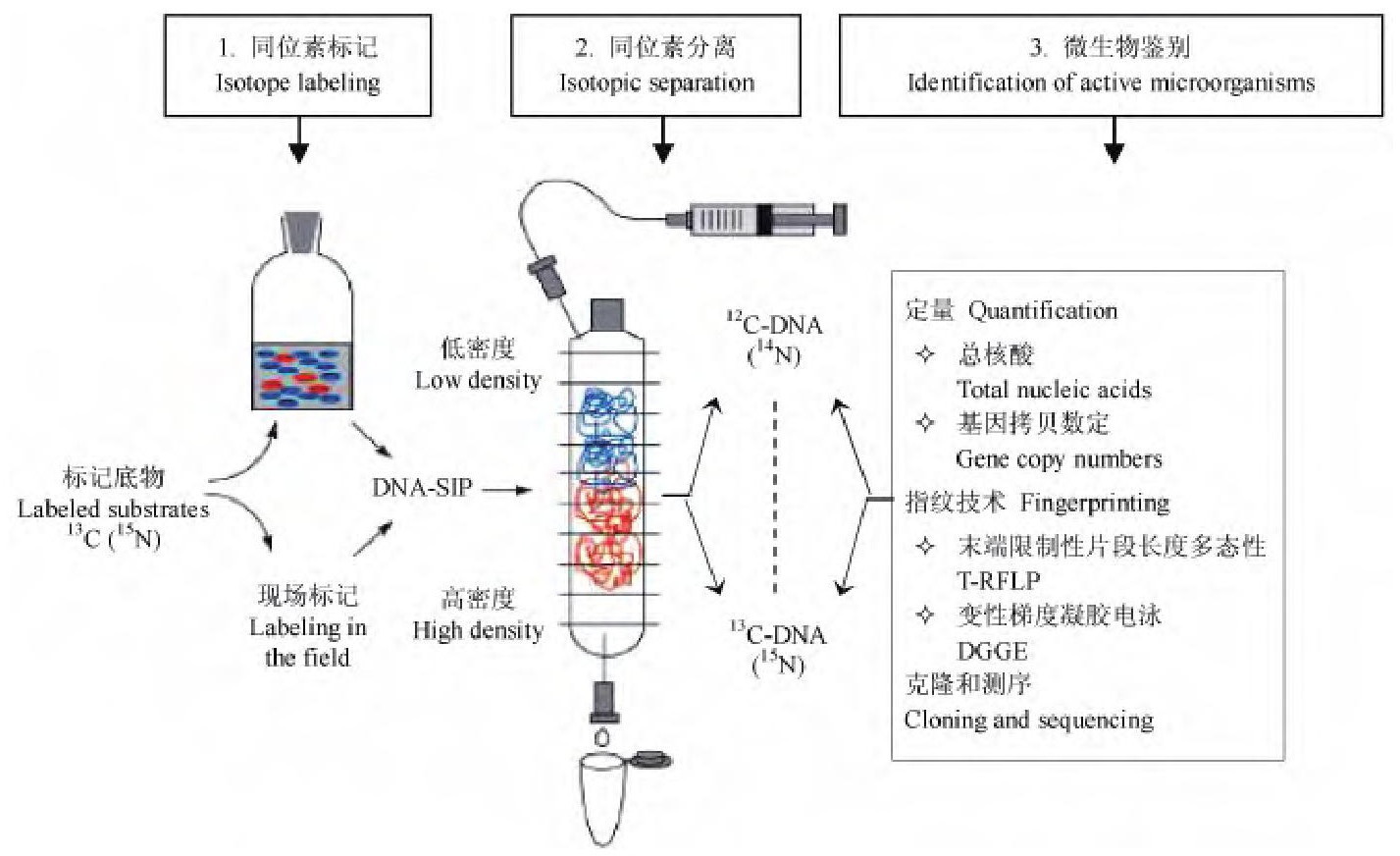
稳定同位素探测 (Stable isotope probing, SIP) 是指利用稳定同位素标记细胞组分, 进而识别出参与某一特定生物代谢过程的微生物种群。该方法实验过程较安全, 没有放射性同位素的危险性。由于核酸是采用分子生物学手段分析微生物的主要对象, 因此将其作为标记物。核酸分子含有碳、氮、氢和氧元素, 可用于核酸标记的稳定同位素如表1所示。根据同位素原子的密度不同区分核酸分子, 例如12C-DNA分子比13C-DNA分子轻。SIP的实验程序[50]总结如图5.首先将稳定同位素标记的底物 (如13C或15N) 与微生物混合培养, 微生物吸收利用该底物合成细胞组分。然后从微生物细胞中提取DNA分子, 吸收了13C或15N底物的DNA分子比12C-DNA或14N-DNA分子重, 利用超速离心机进行密度梯度离心, 分离出高密度的13C或15N-DNA分子。最后对分离出的高密度或低密度DNA分子做进一步的定性或定量分析, 确定微生物种类, 常用的分析方法如图5 (步骤3) 所示。
由于RNA合成速度较快, 而且标记过程无需DNA合成及复制, 一些研究者更倾向于提取RNA, 经密度梯度离心后进行RT-PCR合成c DNA文库, 再进行定量及定性分析[51].目前, 将SIP方法应用于反硝化菌的研究较多[52,53], 而应用于硝化菌的研究则较少。硝化菌属自养微生物, 可采用Na H13CO3培养活性污泥样品, 再提取RNA, 经密度梯度离心分离13C-RNA进行分析。
4、结论与展望
4.1分子生物技术在污水生物处理系统内硝化菌群研究中的应用, 从微生物学角度更本质地揭示了影响污水生物处理系统硝化性能的因素。本文提到的分子生物技术在污水处理中脱氮除磷相关机理和理论的研究中也有着广阔的应用前景, 如短程脱氮、反硝化除磷、同步硝化反硝化及厌氧氨氧化等。通过这些技术可以识别特定生物代谢过程的微生物种群;建立目标菌群动态变化与工艺运行参数之间的相关关系;从微生物学角度对系统运行状态给予最直接、最可靠的分析与证明, 为污水生物脱氮除磷系统的长期稳定运行奠定理论基础, 提供借鉴与指导。
4.2应完善利用分子生物技术对不同类型污水生物处理系统内硝化菌群进行研究时的具体实验方案;同时进行新技术、新手段研究, 从而不断提高分子生物技术方法对污水处理系统中微生物定量、定性的准确性以及检测的特异性, 例如完善Fish-MAR、Clone-FISH、SIP等方法在活性污泥或生物膜样品中的具体操作, 16S r RNA寡核苷酸探针和实时定量PCR引物的设计及特异性研究等, 从而能够更准确地建立微生物与外界环境影响因素的关系。在不久的将来, 分子生物学技术手段将会更加快捷简便, 并将成为污水生物处理系统运行稳定性的常规检测手段。可靠又灵敏的分子生物技术的研发, 也将为实时监测生物反应器的运行开辟新途径。
参考文献
[1]Amann R, Ludwig W, Schleifer KH.Phylogenetic identification and in situ detection of individual microbial cells without cultivation.Microbiol Rev, 1995, 59 (1) :143~169
[2]Wagner M, Amann RI, Kampfer P, Assmus B, Hartmann A, Hutzler P, Springer N, Schleifer KH.Indentifi cation and in situ detection of Gram-negative fi lamentous bacteria in activated sludge.Syst Appl Microbiol, 1994, 17 (3) :405~417
[3]Christensson M, Blackall LL, Welander T.Metabolic transformations and characterisation of the sludge community in an enhanced biological phosphorus removal system.Appl Microbiol Biotechnol, 1998, 49 (2) :226~234
[4]Peng YZ, Yang Q, Liu XH, Zeng W, Mino T, Satoh H.Achieve nitrogen removal via nitrite form municipal wastewater at low temperatures using real-time control to optimize nitrifying communities.Environ Sci Technol, 2007, 41 (23) :8159~8164
[5]Zhang D (张丹) , Xu H (徐慧) , Liu YP (刘耀平) , Zhang Y (张颖) , Chen GX (陈冠雄) , Cleemput O, Willy V.Performance and molecular monitoring of nitrifying bacteria in nitrif ication stage of OLAND system.Chin J Appl Environ Biol (应用与环境生物学报) , 2003, 9 (5) :530~533
[6]Wilderer PA, Bungartzb HJ, Lemmer H, Wagner M, Keller J, Wuertz S.Modern scientifi c methods and their potential in wastewater science and technology.Water Res, 2002, 36 (2) :370~393
[7]Lee N, Nielsen PH, Andreasen KH, Juretschko S, Nielsen JL, Schleifer KH, Wagner M.Combination of fluorescent in situ hybridization and microautoradiography-a new tool for structure-function analyses in microbial ecology.Appl Environ Microbiol, 1999, 65 (3) :1289~1297
[8]Kindaichi T, Ito T, Okabe S.Eco-physiological interaction between nitrifying bacteria and heterotrophic bacteria in autotrophic nitrifying biofi lms as determined by MAR-FISH.Appl Environ Microbiol, 2004, 70 (3) :1641~1650
[9]Okabe S, Kindaichi T, Ito T.Fate of14C-labeled microbial products derived from nitrifying bacteria in autotrophic nitrifying biofi lms.Appl Environ Microbiol, 2005, 71 (7) :3987~3994
[10]Gieseke A, Nielsen JL, Amann R, Nielsen PH, Beer DD.In situ substrate conversion and assimilation by nitrifying bacteria in a model biofi lm.Appl Environ Microbiol, 2005, 7 (9) :1392~1404
[11]Schramm A, Larsen LH, Revsbech NP, Amsing NB, Amann R, Schleifer KH.Structure and function of a nitrifying biofilm as determined by in situ hybridization and the use of microelectrodes.Appl Environ Microbiol, 1996, 62 (12) :4641~4647
[12]Schramm A, Beer DD, Heuvel JC, Ottengraf S, Amann R.Microscale distribution of populations and activities of Nitrosospira and Nitrospira spp.along a macroscale gradient in a nitrifying bioreactor:quantifi cation by in situ hybridization and the use of microsensors.Appl Environ Microbiol, 1999, 65 (8) :3690~3696
[13]Gieseke A, Purkhold U, Wagner M, Amann R, Schramm A.Community structure and activity dynamics of nitrifying bacteria in a phosphate-removing biofi lm.Appl Environ Microbiol, 2001, 67 (3) :1351~1362
[14]Okabe S, Satoh H, Watanabe Y.In situ analysis of nitrifying biofi lms as determined by in situ hybridization and the use of microelectrodes.Appl Environ Microbiol, 1999, 65 (7) :3182~3191
[15]Satoh H, Okabe S, Yamaguchi Y, Watanabe Y.Evaluation of the impact of bioaugmentation and biostimulation by in situ hybridization and microelectrode.Water Res, 2003, 37 (9) :2206~2216
[16]Okabe S, Santegoeds CM, Beer DD.Effect of nitrite and nitrate on in situ sulfi de production in an activated sludge immobilized agar gel fi lm as determined by use of microelectrodes.Biotechnol Bioeng, 2003, 81 (5) :570~577
[17]Schramm A, Fuchs BM, Nielsen JL, Tonolla M, Stahl DA.Fluorescence in situ hybridization of16S rRNA gene clones (Clone-FISH) for probe validation and screening of clone libraries.Environl Microbiol, 2002, 4 (11) :713~720
[18]Simon HM, Dodsworth JA, Goodman RM.Crenarchaeota colonize terrestrial plant roots.Environ Microbiol, 2000, 2 (5) :495~505
[19]Tonolla M, Demarta A, Peduzzi S, Hahn D, Peduzzi R.In situ analysis of sulfate-reducing bacteria related to Desulfocapsa thiozymogenes in the chemocline of meromictic Lake Cadagno (Switzerland) .Appl Environ Microbiol, 2000, 66 (2) :820~824
[20]Liu ZP (刘志培) , Liu SJ (刘双江) .Advances in the molecular biology of nitrifying microorganisms.Chin J Appl Environ Biol (应用与环境生物学报) , 2004, 10 (4) :521~525
[21]Hommes NG, Sayavedra-Soto LA, Arp DJ.Mutagenesis and expression of amo, which codes for ammonia monooxygenase in nitrosomonas europaea.J Bacteriol, 1998, 180 (13) :3353~3359
[22]Purkhold U, Wagner M, Timmermann G, Pommerening A, Koops HP.16S rRNA and amoA based phylogeny of12novel betaproteobacterial ammonia-oxidizing isolates:Extension of the dataset and proposal of a new lineage within the nitrosomonads.Int J Syst Evol Microbiol, 2003, 53 (5) :1485~1494
[23]Aakra A, Utaker JB, Nes IF.Comparative phylogeny of the ammonia monooxygenase subunit A and16S rRNA genes of ammonia-oxidizing bacteria.FEMS Microbiol Lett, 2001, 205:237~242
[24]Nicolaisen MH, Ramsing NB.Denaturing gradient gel electrophoresis (DGGE) approaches to study the diversity of ammonia-oxidizing bacteria.J Microbiol Methods, 2002, 50 (2) :189~203
[25]Rotthauwe JH, Boer W, Liesack W.Comparative analysis of gene sequences encoding ammonia monooxygenase of Nitrosospira sp.AHB1and Nitrosolobus multvormis C-71.FEMS Microbiol Lett, 1995, 133:131~135
[26]Hoshino T, Noda N, Tsuneda S, Hirata A, Inamori Y.Direct detection by in situ PCR of the amoA gene in biofi lm resulting from a nitrogen removal process.Appl Environ Microbiol, 2001, 67 (11) :5261~5266
[27]Rotthauwe JH, Witzel KP, Liesack W.The ammonia monooxygenase structural gene amoA as a functional marker:Molecular fine-scale analysis of natural ammonia-oxidizing populations.Appl Environ Microbiol, 1997, 63 (12) :4704~4712
[28]Cole AC, Shanahan JW, Semmens MJ, LaPara TM.Preliminary studies on the microbial community structure of membrane-aerated biofilms treating municipal wastewater.Desalination, 2002, 146:421~426
[29]Luxmy BS, Nakajima F, Yamamoto K.Analysis of bacterial community in membrane-separation bioreactors byuorescent in situ hybridization (FISH) and denaturing gradient gel electrophoresis (DGGE) techniques.Water Sci&Technol, 2000, 41 (10) :259~268
[30]Ebie Y, Matsumura M, Noda N, Tsuneda S, Hirata A, Inamori Y.Community analysis of nitrifying bacteria in an advanced and compact Gappei-Johkasou by FISH and PCR-DGGE.Water Sci&Technol, 2002, 46 (11) :105~111
[31]Limpiyakorn T, Shinohara Y, Kurisu F, Yagi O.Distribution of ammonia-oxidizing bacteria in sewage activated sludge:Analysis based on16S rDNA sequence.Water Sci&Technol, 2004, 50 (8) :9~14
[32]Mota C, Head MA, Ridenoure JA, Cheng JJ, Reyes FL III.Effects of aeration cycles on nitrifying bacterial populations and nitrogen removal in intermittently aerated reactors.Appl Environ Microbiol, 2005, 71 (12) :8565~8572
[33]Sundberg C, Stendahlb JS, Tonderski K, Lindgren PE.Overland flow systems for treatment of landfi ll leachates-Potential nitrifi cation and structure of the ammonia-oxidising bacterial community during a growing season.Soil Biol Biochem, 2007, 39 (1) :27~138
[34]Wang XD (王晓丹) , Li YH (李艳红) .Advances in studying water microbial ecology by molecular biological techniques.Microbiology (微生物学通报) , 2007, 34 (4) :777~781
[35]Hiraishi A, Iwasaki W, Shinjo H.Terminal restriction pattern analysis of16s rRNA genes for the characterization of bacterial communities of activated sludge.J Biosci&Bioengin, 2000, 90 (2) :148~156
[36]Egli K, Langer C, Siegrist HR, Zehnder AJ, Wagner M, Meer JR.Community analysis of ammonia and nitrite oxidizers during start-up of nitritation reactors.Appl Environ Microbiol, 2003, 69 (6) :3213~3222
[37]Park HD, Noguera DR.Evaluating the effect of dissolved oxygen on ammonia-oxidizing bacterial communities in activated sludge.Water Res, 2004, 38 (14) :3275~3286
[38]Park HD, ReganJM, Noguera DR.Molecular analysis of ammonia-oxidizing bacterial populations in aerated-anoxic Orbal processes.Water Sci&Technol, 2002, 46 (1) :273~280
[39]Limpiyakorn T, Shinohara Y, Kurisu F, Yagi O.Communities of ammonia-oxidizing bacteria in activated sludge of various sewage treatment plants in Tokyo.FEMS Microbiol Ecol, 2005, 54 (2) :205~217
[40]Nakamura Y, Satoh H, Kindaichi T, Okabe S.Community structure, abundance, and in situ activity of nitrifying bacteria in river sediments as determined by the combined use of molecular techniques and microelectrodes.Environ Sci Technol, 2006, 40 (5) :1532~1539
[41]Layton AC, Dionisi H, Kuo HW, Robinson KG, Garrett VM, Meyers A, Sayler GS.Emergence of competitive dominant ammonia-oxidizing bacterial populations in a full-scale industrial wastewater treatment plant.Appl Environ Microbiol, 2005, 71 (2) :1105~1108
[42]Dionisi HM, Harms G, Layton AC, Gregory IR, Parker J, Hawkins SA, Robinson KG, Sayler GS.Power analysis for real-time PCR quantifi cation of genes in activated sludge and analysis of the variability introduced by DNA extraction.Appl Environ Microbiol, 2003, 69 (11) :6597~6604
[43]Harms G, Layton AC, Dionisi HM, Gregory IR, Garrett VM, Hawkins SA, Robinson KG, Sayler GS.Real-time PCR quantification of nitrifying bacteria in a municipal wastewater treatment plant.Environ Sci Technol, 2003, 37 (2) :343~351
[44]Robinson KG, Dionisi HM, Harms G, Layton AC, Gregory IR, Sayler GS.Molecular assessment of ammonia-and nitrite-oxidizing bacteria in full-scale activated sludge wastewater treatment plants.Water Sci&Technol, 2003, 48 (8) :119~126
[45]Okano Y, Hristova KR, Leutenegger CM, Jackson LE, Denison RF, Gebreyesus B, Lebauer D, Scow KM.Application of real-time PCR to study effects of ammonium on population size of ammonia-oxidizing bacteria in soil.Appl Environ Microbiol, 2004, 70 (2) :1008~1016
[46]Hall SJ, Keller J, Blackall LL.Microbial quantification in activated sludge:the hits and misses.Water Sci and Technol, 2003, 48 (3) :121~126
[47]Terahara T, Hoshino T, Tsuneda S, Hirata A, Inamori Y.Monitoringthe microbial population dynamics at the start-up stage of wastewater treatment reactor by terminal restriction fragment length polymorphism analysis based on16S rDNA and rRNA gene sequences.J Biosci&Bioengin, 2004, 98 (6) :425~428
[48]Aoi Y, Shiramasa Y, Tsuneda S, Hirata A, Kitayama A, Nagamune T.Real-time monitoring of ammonia-oxidizing activity in a nitrifying biofi lm by amoA mRNA analysis.Water Sci&Technol, 2002, 46 (1) :439~442
[49]Araki N, Yamaguchi T, Yamazak S, Harada H.Quantifi cation of amoA gene abundance and their amoA mRNA levels in activated sludge by real-time PCR.Water Sci&Technol, 2004, 50 (8) :1~8
[50]Friedrich MW.Stable-isotope probing of DNA:Insights into the function of uncultivated microorganisms from isotopically labeled metagenomes.Curr Opin Biotechnol, 2006, 17 (1) :59~66
[51]Madsen EL.The use of stable isotope probing techniques in bioreactor and fi eld studies on bioremediation.Curr Opin Biotechnol, 2006, 17 (1) :92~97
[52]Ginige MP, Keller J, Blackall LL.Investigation of an acetate-fed denitrifying microbial community by stable isotope probing, full-cycle rRNA analysis, and fluorescent in situ hybridization-microautoradiography.Appl Environ Microbiol, 2005, 71 (12) :8683~8691
[53]Ginige MP, Hugenholtz P, Daims H, Wagner M, Keller J, Blackall LL.Use of stable-isotope probing, full-cycle rRNA analysis, and fluorescence in situ hybridization microautoradiography to study a methanol-fed denitrifying microbial community.Appl Environ Microbiol.2004, 70 (1) :588~596