摘 要: 目的 应用染色体改良高分辨G显带技术, 对四例复杂染色体重排 (CCRs) 携带者进行细胞遗传学分析, 探讨该技术对CCRs的临床应用价值。方法 选取因复发性流产或不孕不育就诊于广东省妇幼保健院医学遗传中心的4对夫妇, 经改良高分辨G显带技术制备外周血染色体, 核型分析并与常规方法进行比较。结果 改良高分辨G显带染色体分辨率达到550条以上核型占比50%以上;核型分析提示4对夫妇中各有一方是CCRs携带者, 涉及3~4条染色体易位;易位染色体涉及3号和12号染色体最多。结论 改良高分辨G显带技术能更好的发现CCRs携带者的多个染色体易位, 避免漏诊。该技术成本较低、操作简单, 适合在基层单位推广。
关键词: 高分辨; G显带; 复杂染色体重排; 染色体;
Abstract: Objective To investigate the cytogenetic analysis of 4 patients with complex chromosomal rearrangements (CCRs) by using chromosome modified high resolution G banding technique, and explore the clinical value of this technique in CCRs. Methods The karyotype of peripheral blood chromosomes was prepared from 4 couples referring for recurrent abortion or infertility in the Medical Genetic Center of Guangdong Women and Children Hospital. The karyotype of peripheral blood was prepared by improved high resolution G banding technique and compared with conventional methods. Results The resolution of the modified high resolution G banding chromosomes was over 50%, and the karyotype analysis showed that one of each couple was CCRs carriers, which involved 3 or 4 chromosome translocations. The most involving chromosomes were 3 and 12. Conclusion Improved high resolution G banding technique can better detect multiple chromosome translocations and avoid missed diagnosis in CCRs carriers. The technology is simple in operation and low cost, and is suitable for promotion in grassroots units.
Keyword: High resolution; G banding; Complex chromosomal rearrangement; Chromosome;
复杂染色体重排 (complex chromosomal rearrangements, CCRs) 是一种染色体结构性异常, 通常涉及两条或更多的染色体, 且至少有3个或以上断裂点, 染色体重新排列组合并伴随遗传物质的交换[1]。CCRs携带者临床表现多样, 包括完全正常的表型、复发性流产、不孕不育、智力低下、发育迟缓、先天畸形等[2]。CCRs在表型正常的人群中少见, 但在复发性流产、不孕不育、不良生育史等患者中相对较多。CCRs的发生机制复杂, 携带者或是新发, 或是家族性遗传[3,4]。由于CCRs涉及重排的染色体和断点的数量增加, 配子发生不平衡的百分比和子代受影响的风险越高[1,5]。携带者生育先天性多发畸形 (multiple congenital abnormalities, MCA) 患儿风险与涉及的染色体、受累数目和断裂点位置等有关[5]。所以对表型正常的CCRs携带者做出准确核型诊断, 对孕前咨询和胎儿染色体异常的风险评估非常重要。常规染色体G显带在分辨率低的情况下, 辨别CCRs存在困难, 容易漏诊。因此对该类患者进行针对性的细胞遗传学诊断显得非常关键。目前大多基层实验室开展染色体检测的分辨率在320条带左右, 对CCRs难以识别。本研究应用改良的高分辨G显带技术, 对4例CCRs携带者进行细胞遗传学分析并探讨该技术对CCRs的临床应用价值。

1、 对象与方法
1.1、 研究对象
选取因复发性流产或不孕不育就诊于广东省妇幼保健院医学遗传中心的夫妇4对共8人, 签署知情同意书后抽取外周血行改良高分辨G显带染色体分析。病例1, 30岁女性婚后2次早期胚胎停育。病例2, 28岁女性, 早期自然流产3次。病例3, 33岁男性婚后多年不育, 精液常规检测正常。病例4, 39岁男性, 畸、弱精子症, 其妻早期自然流产1次。4对夫妻均否认近亲结婚, 除上述外其他既往病史无特殊。
1.2、 方法
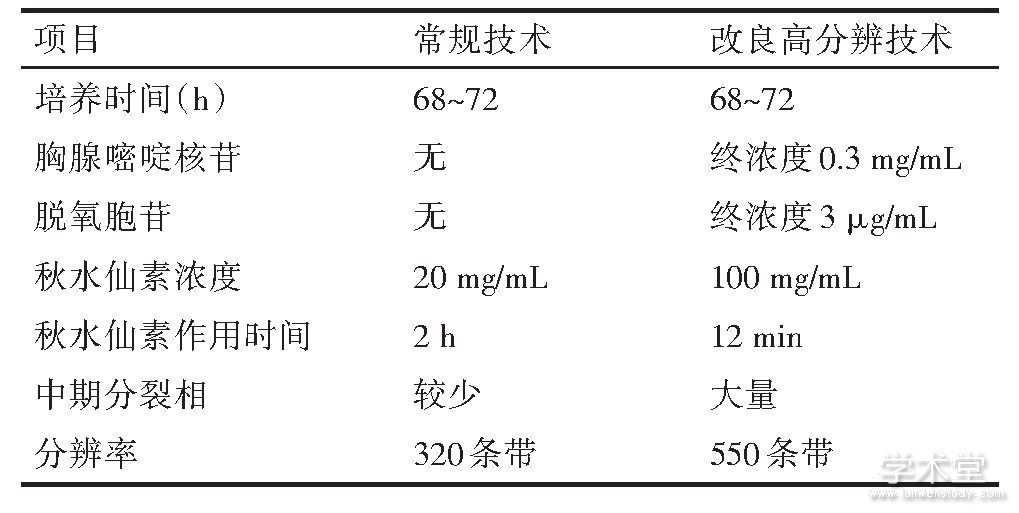
抽取2 m L静脉肝素抗凝血, 接种于2瓶外周血培养基, 置于37℃培养箱培养72 h。在终止培养前16 h加入胸腺嘧啶核苷100μL (终浓度达到0.3 mg/m L) , 前6 h加入脱氧胞苷 (终浓度达到3μg/m L) 。收获前加入50μL高浓度 (100 mg/m L) 的秋水仙素 (终浓度为2μg/m L) 作用12 min, 经常规低渗、预固定、固定、制片 (4张/人) 、吉姆萨染色, 全自动染色体扫描仪选取、拍摄核型, 按《人类细胞遗传学国际命名体制ISCN (2016) 》分析和诊断染色体核型[6]。常规技术与改良高分辨G显带技术的方法比较见表1。
表1 常规技术与改良高分辨G显带技术的方法比较
2、 结果
2.1、 制备结果
所有对象进行改良高分辨G显带技术均获得成功, 每例均可至少制片4张, 每张玻片至少80个核型, 分辨率在550条带核型数占50%以上。分析4对夫妇外周染色体G显带核型结果如下:
病例1核型为:46, XX, t (3;12;18) (18qter→18q21.1::3p25→3qter;12pter→12q22::3p25→3pter;18pter→18q21.1::12q22→12qter) , 其丈夫核型正常, 见图1。
图1 病例1改良高分辨染色体核型
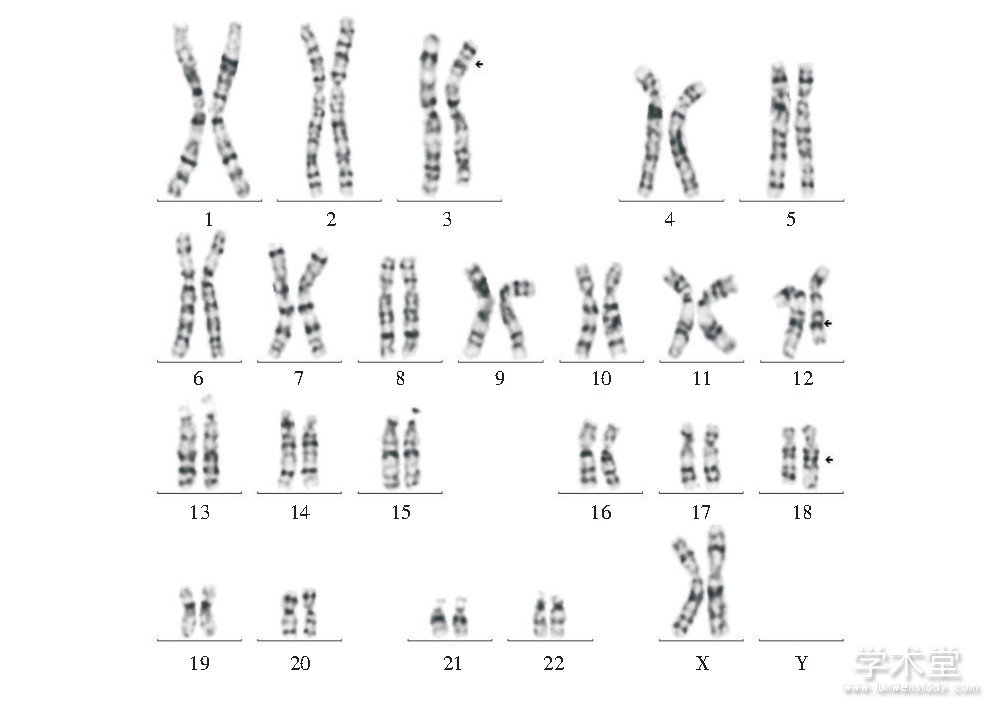
Fig.1 Modified high resolution chromosome karyotype of case 1
病例2核型为:45, XX, t (3;12) (3pter→3q21::12q24.1→12qter;12pter→12q24.1::3q21→3qter) , der (13;14) (q10;q10) , 其丈夫核型正常, 见图2。
图2 病例2改良高分辨染色体核型
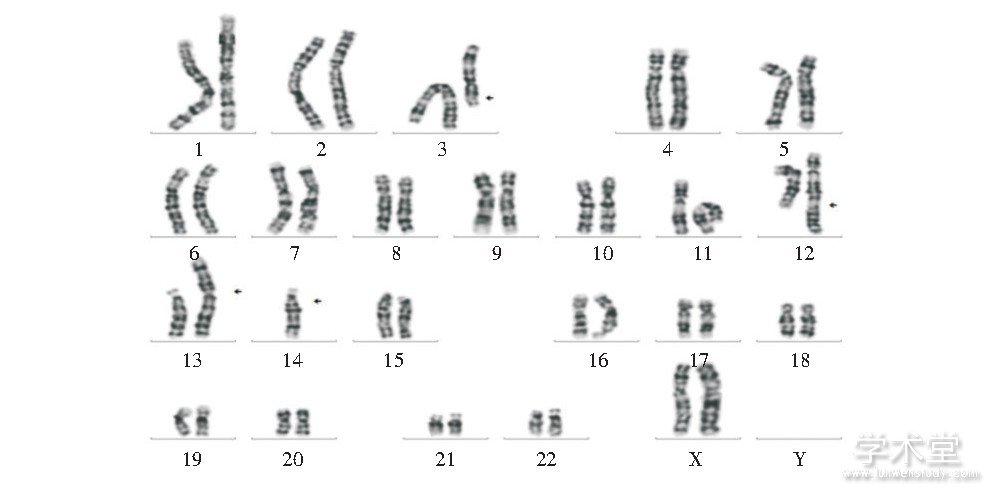
Fig.2 Modified high resolution chromosome karyotype of case 2
病例3核型为:46, XY, der (1) (1pter→1q43::3q25→3q26.2::12q24.1→12qter) , der (3) (pter→q25::q26.2→qter) , der (12) (12pter→12q24.1::1q43→1qter) , 其妻子核型正常, 见图3。
图3 病例3改良高分辨染色体核型
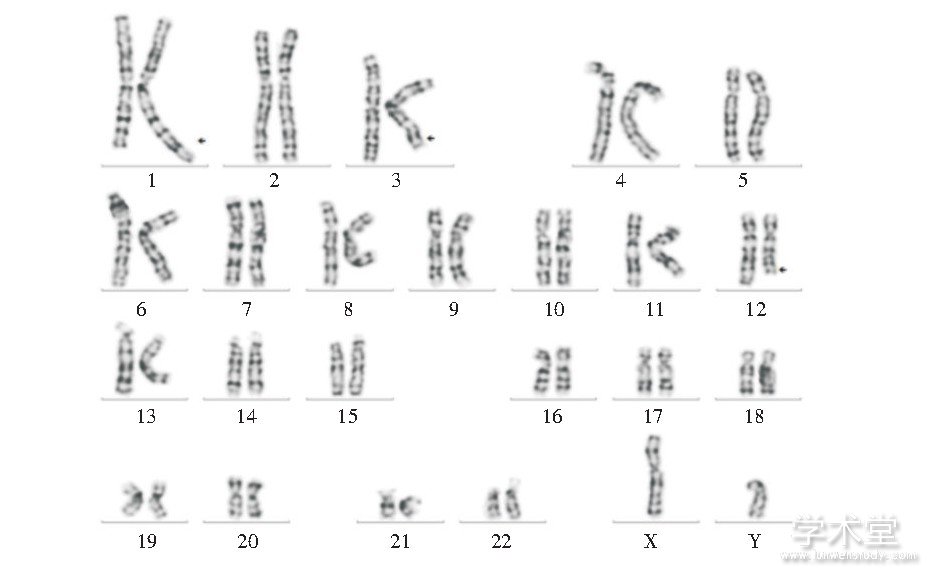
Fig.3 Modified high resolution chromosome karyotype of case 3
病例4核型为:46, XY, der (5) (6qter→6q23::5p15.1→5qter) , der (6) (6pter→6q22::7p21→7pter) , der (7) (5pter→5p15.1::6q23→6q22::7p21→7qter) , 其妻子核型正常, 见图4。
2.2、 4例CCRs携带者异常染色体特点分析
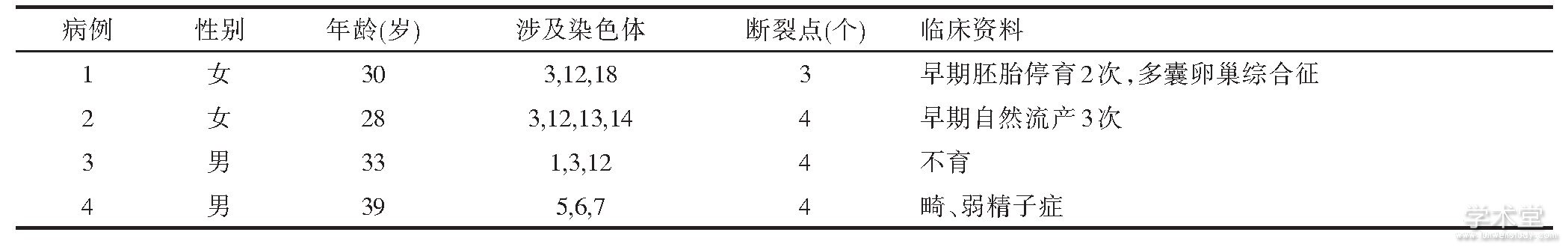
结合改良高分辨G显带分析结果, 比较4例CCRs携带者细胞遗传特点及临床资料, 发现4对夫妇均存在复发性流产或不孕不育病史, 并且其中一方染色体异常, 另一方染色体正常。染色体异常一方, 存在3~4个染色体断裂点, 涉及3~4条染色体易位, 其中以3号和12号染色体最多见, 见表2。
图4 病例4改良高分辨染色体核型
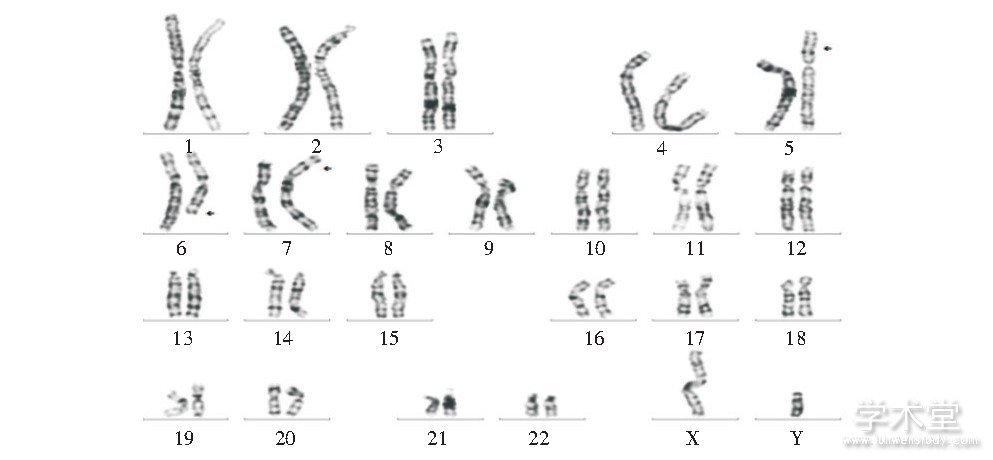
Fig.4 Modified high resolution chromosome karyotype of case 4
表2 四例CCRs携带者细胞遗传特点及临床资料
3、讨论
常规染色体制备方法于细胞培养68~72 h, 在收获前直接加低浓度秋水仙素作用2 h后直接低渗固定, 制备的染色体条带分辨率约为320条, 对核型分析技术相对较低。由于操作简单, 为广大细胞遗传实验室应用。但该分辨率对染色体微小结构异常、复杂重排容易漏诊, 导致CCRs涉及的染色体数目和断点容易发生错误。而高分辨技术始于1976年Yunis[7]的细胞周期同步化方法, 后经许多学者改良成经典的高分辨技术, 但操作步骤繁琐, 收获的染色体分辨率太高, 在850条以上, 对核型分析技术要求非常高, 因此并不适合常规开展。本研究在常规技术基础上改良高分辨技术, 操作上仅通过适时加入胸腺嘧啶核苷和脱氧胞苷, 并调整秋水仙素浓度和作用时间, 即可达到临床诊断CCRs的目的。实验中过量的胸腺嘧啶核苷能可逆的抑制DNA合成, 使大量细胞静止在G1/S期, 再加入脱氧胞苷使细胞同步化进入分裂中期, 从而收获大量足够染色体分裂相。秋水仙素能破坏细胞分裂周期中出现的纺锤体, 使细胞终止在分裂中期, 同时能收缩染色体。如秋水仙素作用时间太长, 可造成染色体过短。本研究在常规技术上, 采用增加秋水仙浓度并缩短作用时间的方法, 即获得大量长度适中的染色体分裂相;在参考胸腺嘧啶核苷双阻断同步化高分辨技术的基础上, 改良为单阻断且不需离心去除阻断剂直接释放进入细胞中期, 简便实验操作, 且收获到的染色体分裂相达550条带占50%以上, 而又不至于过高的分辨率, 可满足临床上检测CCRs的要求。
CCRs通常涉及2条以上的染色体断裂, 导致染色体片段交换[8], 迄今报告病例约为300多例[5,8,9,10]。3条或者以上的染色体复杂重排, 理论上在减数分裂中至少会形成32种配子, 其中只有1种平衡易位配子, 1种正常配子, 其他均为部分单体或部分三体等非平衡型配子, 此类配子受精后不能正常发育, 结局多为自然流产[11]。CCRs的发生机制尚未完全阐明。染色体在各种内外因素的作用下受到破坏, 导致DNA双链断裂, 断裂的染色体在DNA修复功能的作用下发生非同源末端连接, 从而形成了染色体的重排, 如果涉及到3条或以上的染色体, 则形成了CCRs[12]。约70%的CCRs携带者表型正常, 20%~25%有先天畸形和精神障碍, 5%~10%在介入性产前诊断中被检测到[13]。70%~75%的CCRs为新发, 30%为家族性遗传, 新发的CCRs携带者中表型正常和异常的比例大致相同[14]。
Kausch等[15]根据CCRs的结构, 将其分为三型:Ⅰ型为三相重排CCRs, 即3条染色体上的3个片段发生断裂、易位、重组, 形成3条衍生染色体, 一般断裂点为3个;Ⅱ型为特殊CCRs, 包括一系列的复杂重组 (易位、倒位、插入、复制、缺失) , 涉及的断裂点一般比衍生染色体的数目多, 在减数分裂时增加额外的复杂性;Ⅲ型为双重相互易位CCRs, 包括2对或以上的染色体之间的相互易位, 通过减数分裂单独分离, 产生正常和衍生染色体。Ⅰ型CCRs最常见, 约占30%[16], 且随着断裂位点和涉及染色体数目的增加, CCRs携带者生育异常后代的几率越大[17]。本研究中病例1属于Ⅰ型, 涉及3条染色体3个断裂点, 患者有复发性流产史, 其两次胚胎停育可能是减数分裂后产生不平衡配子, 导致胚胎染色体异常引起流产。病例2属于Ⅲ型, 涉及4条染色体4个断裂点, 为罗氏易位合并平衡易位, 患者生育正常染色体后代的理论概率为 (1/6×1/18) 。病例3为Ⅱ型, 涉及3条染色体4个断裂点, 患者不孕很可能是正常配子几率太低。病例4属于Ⅱ型, 涉及3条染色体4个断裂点, 患者畸、弱精子症, 据报道常染色体结构畸变可影响精子的形成和质量[18], 其妻流产过一次且之后一直未再孕, 可能与此有关。Vermeulen等[19]报道约30%的CCRs有7q21.1断裂点, Zhang等[20]报道2、3、4、7、11等染色体容易形成CCRs, 本研究中4个病例中有3例含有3号和12号染色体的CCRs, 占75%, 推测12号染色体也许含有CCRs热点区域。
随着分子遗传技术飞速发展, 荧光原位杂交、染色体微阵列分析、二代测序 (next-generation sequencing, NGS) 等促进了染色体断裂点的特征描述和精确划分[5], 可发现更多的染色体微小易位, 促进遗传物质重排的诊断[21]。目前NGS对CCRs的详细研究已证明是有成效的[22], 因此对CCRs的研究也成为热点。同时也明确提出传统的细胞遗传核型分析仍是不可缺少的技术[9,23], 从而对传统细胞遗传的高分辨技术提出了更高的质量要求。本研究改良高分辨G显带技术成本低、方便操作、容易掌握, 尤其适合基层单位开展, 及时发现CCRs携带者, 避免过度检查和治疗。
参考文献
[1]Madan K.Balanced complex chromosome rearrangements:reproductive aspects.A review[J].Am J Med Genet A, 2012, 158A (4) :947-963.
[2]Nguyen MH, Morel F, Pennamen P, et al.Balanced complex chromosome rearrangement in male infertility:case report and literature review[J].Andrologia, 2015, 47 (2) :178-185.
[3]Alesi V, Orlando V, Genovese S, et al.Interstitial 10q21.1q23.31duplication due to meiotic recombination of a paternal balanced complex rearrangement:cytogenetic and molecular characterization[J].Cytogenet Genome Res, 2017, 151 (4) :179-185.
[4]de Pagter MS, van Roosmalen MJ, Baas AF, et al.Chromothripsis in healthy individuals affects multiple proteincoding genes and can result in severe congenital abnormalities in offspring[J].Am J Hum Genet, 2015, 96 (4) :651-656.
[5]Pellestor F, Anahory T, Lefort G, et al.Complex chromosomal rearrangements:origin and meiotic behavior[J].Hum Reprod Update, 2011, 17 (4) :476-494.
[6]吴菁, 尹爱华, 傅文婷, 等.改进同步化法制备外周血染色体500条G显带技术的临床应用研究[J].中华医学遗传学杂志, 2013, 30 (3) :377-378.
[7]Yunis JJ.High resolution of human chromosomes[J].Science, 1976, 191 (4233) :1268-1270.
[8]Poot M, Haaf T.Mechanisms of origin, phenotypic effects and diagnostic implications of complex chromosome rearrangements[J].Mol Syndromol, 2015, 6 (3) :110-134.
[9]Aristidou C, Theodosiou A, Ketoni A, et al.Cryptic breakpoint identified by whole-genome mate-pair sequencing in a rare paternally inherited complex chromosomal rearrangement[J].Mol Cytogenet, 2018, 11:34.
[10]廖亚平, 王春景, 梁猛, 等.平衡复杂染色体重排携带者的遗传与生育情况分析[J].遗传, 2017, 39 (5) :396-412.
[11]Zhang T, Sun Y, Chen Z, et al.Traditional and molecular chromosomal abnormality analysis of products of conception in spontaneous and recurrent miscarriage[J].BJOG, 2018, 125 (4) :414-420.
[12]Tsai AG, Lieber MR.Mechanisms of chromosomal rearrangement in the human genome[J].BMC Genomics, 2010, 11 Suppl 1:S1.
[13]De Gregori M, Ciccone R, Magini P, et al.Cryptic deletions are a common finding in“balanced”reciprocal and complex chromosome rearrangements:a study of 59 patients[J].J Med Genet, 2007, 44 (12) :750-762.
[14]Patsalis PC.Complex chromosomal rearrangements[J].Genet Couns, 2007, 18 (1) :57-69.
[15]Kausch K, Haaf T, K?hler J, et al.Complex chromosomal rearrangement in a woman with multiple miscarriages[J].Am JMed Genet, 1988, 31 (2) :415-420.
[16]Kousseff BG, Papenhausen P, Essig YP, et al.Complex chromosome rearrangement with ankyloblepharon filiforme adnatum[J].J Med Genet, 1993, 30 (2) :167-170.
[17]Giardino D, Corti C, Ballarati L, et al.Prenatal diagnosis of a de novo complex chromosome rearrangement (CCR) mediated by six breakpoints, and a review of 20 prenatally ascertained CCRs[J].Prenat Diagn, 2006, 26 (6) :565-570.
[18]Martin RH.Cytogenetic determinants of male fertility[J].Hum Reprod Update, 2008, 14 (4) :379-390.
[19]Vermeulen S, Menten B, Van Roy N, et al.Molecular cytogenetic analysis of complex chromosomal rearrangements in patients with mental retardation and congenital malformations:delineation of7q21.11 breakpoints[J].Am J Med Genet A, 2004, 124A (1) :10-18.
[20]Zhang Y, Dai Y, Tu Z, et al.Array-CGH detection of three cryptic submicroscopic imbalances in a complex chromosome rearrangement[J].J Genet, 2009, 88 (3) :369-372.
[21]林欣怡, 宋新明, 李颖, 等.染色体微小易位致智力障碍家系的遗传学研究[J].热带医学杂志, 2018, 18 (5) :574-577, 602.
[22]Schluth-Bolard C, Labalme A, Cordier MP, et al.Breakpoint mapping by next generation sequencing reveals causative gene disruption in patients carrying apparently balanced chromosome rearrangements with intellectual deficiency and/or congenital malformations[J].J Med Genet, 2013, 50 (3) :144-150.
[23]Aristidou C, Koufaris C, Theodosiou A, et al.Accurate breakpoint mapping in apparently balanced translocation families with discordant phenotypes using whole genome mate-pair sequencing[J].PLoS One, 2017, 12 (1) :e0169935.





