摘 要: 胶原酶具有很强的特异性,其降解胶原的能力在动物细胞外基质的整体降解中起着关键作用。首先介绍了微生物胶原酶的分类和结构,简述了目前微生物胶原酶活性测定、胶原酶基因克隆、表达和纯化表征的技术及方法,然后综述了胶原酶在皮革工业、医药工业和细胞分离等领域的应用及其前景,最后对胶原酶现存在的问题也做了简要的概括,并对未来的发展方向进行了展望。
关键词: 微生物; 胶原酶; 分类; 应用; 研究进展;
Abstract: Collagenase is highly specific and plays a key role in the overall degradation of animal extracellular matrix due to its ability to degrade collagen. This review introduced the classification and structure of collagenase, and expounded the techniques and methods used in the activity determination, gene cloning, expression, purification and characterization of microbial collagenase. The application and prospect of collagenase was then summarized in leather industry, medical industry, cell separation and other fields. Finally, the existing problems for collagenase were briefly summarized, and the future development direction was prospected.
Keyword: microorganism; collagenase; classification; application; research progress;
前言
胶原蛋白是动物结缔组织(如皮肤、骨骼、软骨、腱、血管)的主要结构蛋白。它是由三条主要重复Gly-X-Y三个氨基酸序列的α链组成(X和Y通常代表脯氨酸和羟脯氨酸),它们使每个链保持左螺旋,具有明显的蛋白四级结构[1]。胶原蛋白的水解对动物的许多生理功能是必不可少的,包括组织重建、形态形成和伤口愈合等[2]。例如:随着年龄的增长,人体合成胶原数量会呈下降趋势。但有一些身体部位对其需求却在增加,胶原蛋白多肽就可以应对这一状况。胶原蛋白多肽是胶原蛋白的水解产物,在日化工业、功能食品等领域均具有很高的利用价值。此外,胶原蛋白多肽在医学和药学领域也具有多种生物活性,可以治疗骨质疏松症、胃溃疡、高血压等多种疾病,也可以用于制备皮肤的保湿剂[3],因此胶原蛋白多肽在商业上的应用价值正在迅速提升。
目前,胶原蛋白水解方法有酸法、碱法和酶法,但是酸法水解和碱法水解会破坏氨基酸,与这些方法相比,酶法水解反应温和,对氨基酸破坏力小,安全可靠,无污染,而且它们具有高度的特异性,其中胶原酶更是可以在中性p H条件和接近于体表温度的情况下发挥作用,在极低的浓度下仍具有很高的活性。随着科学技术的发展,人们发现很多微生物中都含有胶原酶,特别是相对于动物来源的胶原酶来说,更易于表达和纯化。而微生物胶原酶独特的水解活性,使得其在生物技术、医药、食品、酿造、肉类加工、化妆品、动物饲料等领域中有着广泛的应用[4,5,6,7]。
1 、胶原概述
胶原蛋白是生物高分子,也是哺乳动物体内含量最多、分布最广的功能性蛋白,占体内蛋白质总量的25%~30%,在某些生物体内占的比例甚至高达80%[8]。胶原蛋白分子呈棒状,长约300 nm,直径在1.5 nm左右,相对分子质量约200~300 k Da。胶原蛋白由3条α链组成,每条链主要由重复的三个氨基酸Gly-X-Y序列组成,羟脯氨酸残基可形成氢键稳定胶原蛋白分子,它的数量直接影响了胶原蛋白纤维的热稳定性和机械强度。这三条α链通过氢键、共价交联及范德华力以右手螺旋形式缠绕成三股螺旋结构,使胶原蛋白具有很高的拉伸强度[9,10]。
胶原蛋白作为生物材料具有很低的免疫原性,在不含端肽的情况下免疫原性更低[11];胶原蛋白本身是构成细胞外基质的骨架的主要分子,其三股螺旋结构及交联所形成的纤维或网络对细胞具有稳定和支持作用,并为细胞的增值生长提供适当的环境,所以其与宿主细胞及组织之间有良好的相互作用[12];胶原蛋白能被蛋白酶水解,故其具有良好的生物降解性[13];胶原蛋白还具有止血性能[14,15],通过促进血小板凝聚和血浆结块可以有效实现止血。胶原蛋白所具有的良好生物学性能,使其在医学(美容)、食品(营养学)、皮革(通过改性促进制革过程中化工材料吸收)等领域有着十分广泛的应用。
2、 胶原酶
胶原酶通常被认为是特异性水解天然胶原蛋白和水溶性变性胶原蛋白的酶。从胶原酶来源的角度出发,胶原酶一般可分为植物胶原酶、动物胶原酶和微生物胶原酶(如图1所示)。据报道,从姜根茎(丁基纤维)[16],无花果的乳胶[17]中已经分离鉴定出了胶原酶,而且植物胶原酶优先在特定位点切割天然胶原蛋白。动物胶原酶通常分布在脊椎动物体内,其可以在天然胶原蛋白的三个α链上的单个肽键水解胶原,从X-Gly键上分子的四分之三长度切割胶原蛋白的三螺旋结构。
图1 胶原酶的分类
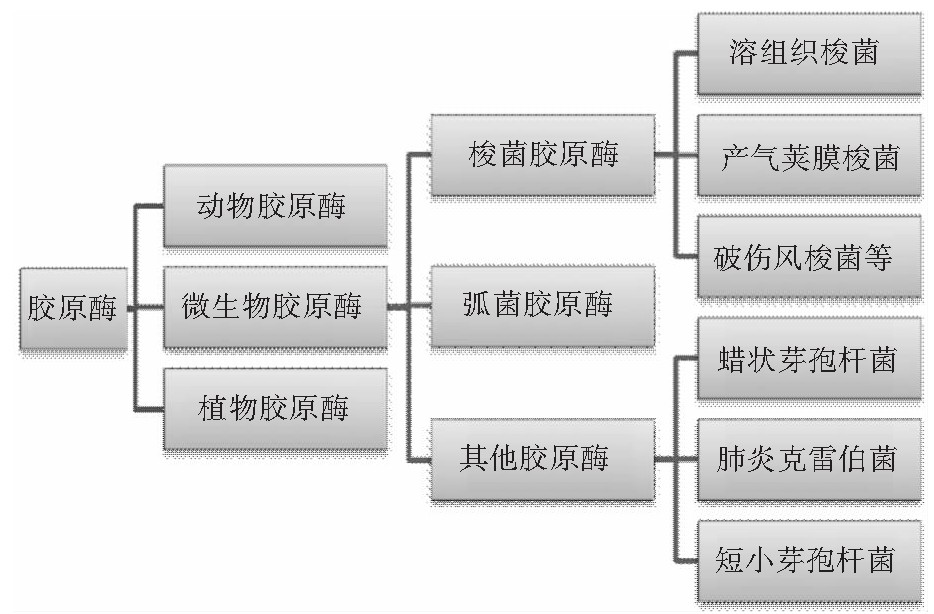
Fig.1 Classification of microbial collagenase
微生物胶原酶实际上是一种金属内肽酶,依靠于Zn2+、Ca2+等离子发挥活性。它不但可以水解变性的可溶性胶原蛋白,而且可以水解天然不溶性胶原蛋白[18]。与动物胶原酶相比,微生物胶原酶底物种类更广泛,水解底物更彻底。有的微生物胶原酶可以与胶原蛋白的多个位点发生反应,产生大小均匀的5个残基的短肽。但是动物胶原酶只能与胶原蛋白N端3/4处Gly-Leu或Gly-lle肽键发生反应,产生一个3/4肽段和1/4肽段[19]。微生物胶原酶更容易获得,是因为微生物可以将胶原酶分泌到胞外,通过发酵可以大量生产,而动物胶原酶还需要进行组织培养和提取,工序繁琐且成本很高。
3 、微生物胶原酶的分类
微生物胶原酶在肽酶数据库里属于M9家族,其根据氨基酸序列和催化功能的不同被分为M9A和M9B两个亚家族,其中来源于梭菌和弧菌的胶原酶被广泛研究[20]。
3.1 、梭菌胶原酶
梭菌胶原酶属于M9B亚家族,其中研究最充分的是来源于溶组织梭菌的胶原酶。1937年,马斯曼(Maschmann)首次发现并报道了第一种梭菌胶原酶。随后,科学家们进行了更加深入的研究。根据其对不同底物(胶原蛋白,明胶,合成底物等)的相对活性、稳定性以及胶原酶的一级、二级和三级结构特征,溶组织梭菌胶原酶被分为两类,即I类和II类胶原酶[21]。其中I类胶原酶对天然胶原具有高水解活性,对FALGPA合成胶原肽具有中等水解活性,而II类胶原酶对天然胶原具有中等水解活性,对FALGPA合成肽具有高水解活性。Matsushita研究小组在溶组织梭菌染色体中发现了两个胶原酶基因col G和col H,其中I类胶原酶由col G基因翻译,而II类胶原酶由col H基因翻译[22,23]。除此之外,科学家还发现了其他梭菌物种胶原酶及其编码基因。例如:产气荚膜梭菌的col A基因[24]和破伤风梭菌的col T基因[25]等。
溶组织梭菌胶原酶分子是一种多结构域的蛋白质,两类胶原酶col G和col H在结构上也存在着差异。col G胶原酶由S1、S2、S3a和S3b等4个结构域组成,col H胶原酶由S1、S2a、S2b和S3等4个结构域组成[26]。其中S1是胶原酶结构域中的催化结构域(AD),S2和S3分别代表PKD和CBD结构域,PD是肽酶结构域(如图2所示)。研究表明,col G胶原酶中S3b含有结合胶原蛋白三螺旋结构区域,但是不能与缺乏三螺旋的相似肽和明胶(变性胶原)结合。来自col H胶原酶的S3结构与来自col G胶原酶的S3b结构相似,它们共享30%的氨基酸序列,S3b中参与底物结合的9个残基中,只有3个芳香残基被保存在S3中,根据序列对比,S3b中的一个Ca2+的天冬氨酸(Asp927)被S3中的丝氨酸(Ser896)所取代[27]。PKD结构域和CBD结构域对于三螺旋胶原蛋白的水解不是必需的,但是对于较大的胶原实体(例如胶原纤维)却是必需的[28]。I类和II类胶原酶的作用位点是胶原蛋白α链中重复Gly-X-Y序列中的Y-Gly键,且有研究证明这两类胶原酶在胶原蛋白水解中具有协同作用[29]。
3.2、 弧菌胶原酶
弧菌胶原酶属于M9A亚家族。最初,弧菌金属蛋白酶被分为I类和II类金属蛋白酶。在I类金属蛋白酶中,锌结合基序包括HEXXH-E序列,而II类金属蛋白酶具有一致的HEXXH序列和一个谷氨酸作为位于HEXXH基序下游的第三个锌结合配体[30]。后来根据氨基酸序列、酶分子质量、底物特异性和功能,把这两类弧菌金属蛋白酶又重新分为三类[31](如图3所示),第三类蛋白酶在羧基末端包含一个PKD结构域和一个PPC结构域(前肽酶结构域),关于弧菌蛋白酶中的PPC结构域的结构和功能还是未知的,但也有文献指出,来自创伤弧菌的PPC结构域与IV型胶原可以相互作用[32]。根据序列分析和结构域排列证据表明,仅来自第二类(MEROPS M09.004)和第三类(MEROPS M09.001)的弧菌金属蛋白酶被视为胶原酶[33,34]。弧菌胶原酶水解胶原的第一步,优先切割三螺旋胶原蛋白的距N末端四分之三位置的X-Gly肽键,而不是动物胶原酶的Gly-Leu或Gly-Ile肽键。事实上,与梭菌胶原酶一样,弧菌胶原酶在Leu-Gly键上水解合成肽Pz-Pro-Leu-Gly-Alu-D-Arg。
图2 colG和colH胶原酶结构图
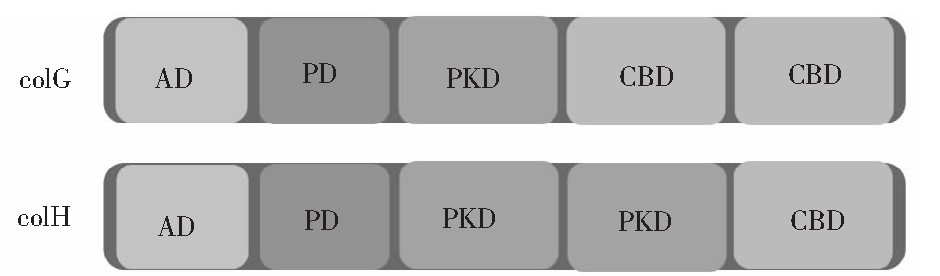
Fig.2 The domain structures of col G and col H Collagenase
图3 三类弧菌金属蛋白酶结构图
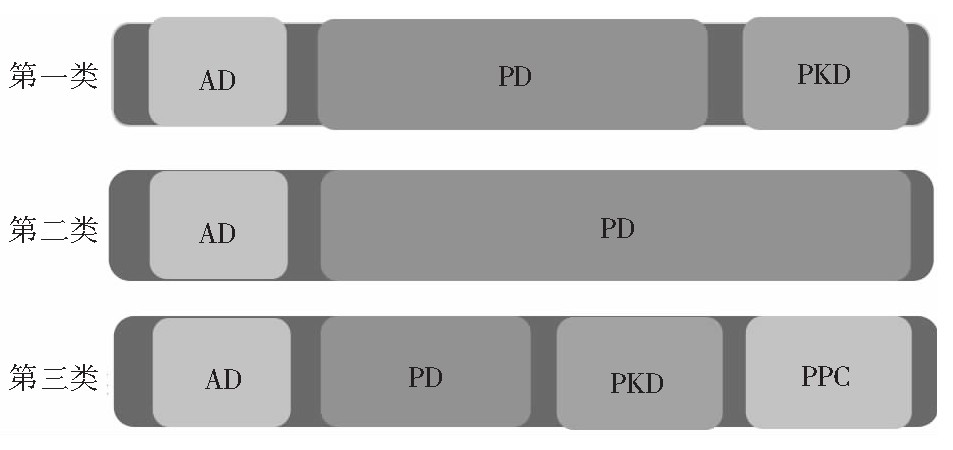
Fig.3 The domain structures of three types of metallopro-teinases from vibrio
3.3 、其他来源
除梭菌胶原酶和弧菌胶原酶,短小芽孢杆菌[36]、蜡样芽孢杆菌[37]、枯草芽孢杆菌[38]、白色念珠菌[39]等都可产生胶原酶,但是这些微生物产生胶原酶的晶体结构还未被全部解析,作用位点尚未明确,Punta[40]等人发现了在众多微生物胶原酶中,它们所包含的结构域的种类和数量各不相同,但部分结构域的结构具有相似性。Abfalter等人[41]以蜡样芽孢杆菌ATCC 14579 col A的全长氨基酸序列作为目标序列,利用BLAST进行序列比对得到蜡样芽孢杆菌中Y93-K850和col G具有49.8%的同源性,故采用SWISS-MODEL以col G为指定模板对Y93-K850进行了同源建模,发现此模型包括激活结构域、催化亚结构域和辅助催化亚结构域,这些发现可以使人们更深入地了解微生物胶原酶的功能和机制,以明确它们的应用方向。因此,对微生物胶原酶及其编码基因进行更多的研究是很有必要的。
4 、微生物胶原酶的活性测定
微生物胶原酶在众多工业领域具有很高的应用价值,因此测定胶原酶的活性变得十分重要。最常见是利用茚三酮显色法测定胶原酶的活性,将水溶性或水不溶性胶原蛋白用作胶原酶水解的底物,用分光光度计测量底物胶原生成的氨基酸数量来确定胶原酶活性。此方法一般将1min内在特定条件下能水解底物产生相当于1μg氨基酸的酶量定义为1个酶活力单位。除此之外,合成肽N-(3-[2-fury]acryloyo)-Leu-Gly-Pro-Ala(FALGPA)也可以作为有效测定胶原酶活性的底物,而且相比其他底物反应更为迅速[42]。Zhang等人还利用14C,2H或3H放射性标记或荧光标记的胶原作为底物,来定量评估胶原酶活性[43]。茚三酮显色法易受环境因素影响,而合成肽和荧光标记胶原蛋白作为底物需要高成本并导致放射性废物的产生。所以在众多测定法中,依然没有一种既简单可靠又经济的方法测定胶原酶活性。
5 、微生物胶原酶的基因克隆、纯化和表征
基因的克隆和核苷酸序列检测为解析酶的结构和功能提供了帮助和便利。现在,科学家已经从一些微生物中纯化出了微生物胶原酶,并且已经克隆了它们的相应基因并对其进行了测序。溶组织梭菌的col G[44]、col H[45]和破伤风杆菌的col T基因[45]都已被成功克隆,并在大肠杆菌中重组表达;霍氏弧菌的胶原酶基因也已被克隆,并确定了其完整核苷酸序列[46]。微生物胶原酶纯化的方法有硫酸铵沉淀、超滤、镍柱层析、凝胶过滤层析、离子交换层析等[47]。但由于纯化成本很高,也就限制了微生物胶原酶在一些工业领域的应用。
微生物胶原酶的表征可以从他们的分子质量、动力学参数以及p H、热稳定性、金属离子对酶活力的影响等方面入手。测定酶分子质量的方法有十二烷基硫酸钠聚丙烯酰胺凝胶电泳法、凝胶渗透色谱法、高效液相色谱与电喷雾质谱联用技术等;改变底物浓度时,反应速率会随之变化,这时可利用双倒数作图法计算得出动力学参数;微生物胶原酶在反应中的最适p H、最适温度和其他影响因素可以通过测定酶活力来确定,其中金属离子在改善微生物胶原酶活性中具有重要作用,有的离子起促进作用,如Ca2+、Zn2+,而有的离子会有抑制作用,如Fe2+、Mn2+[31],如表1所示。
表1 不同离子对不同来源胶原酶活性的影响
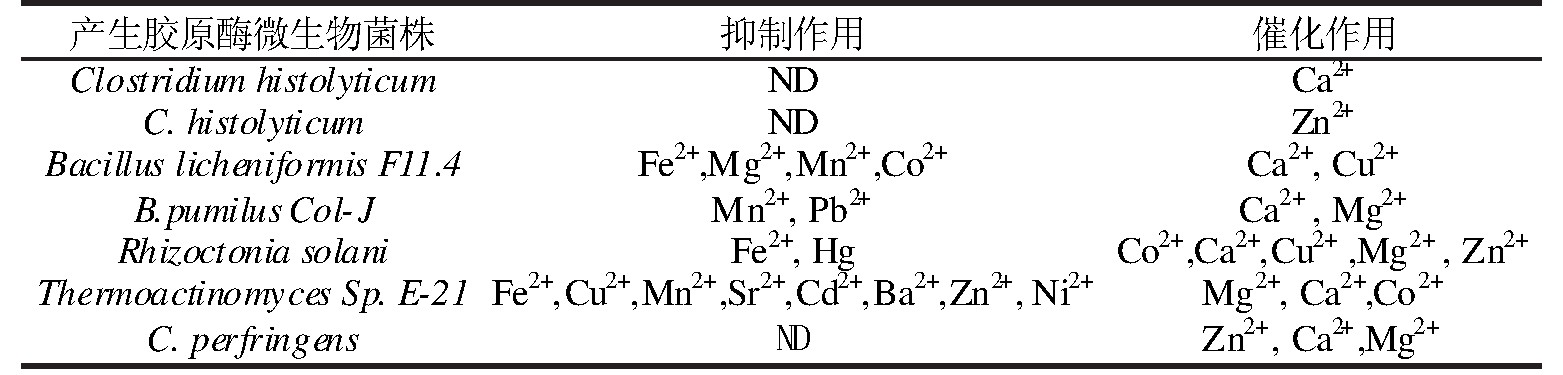
ND=not determined
6、 微生物胶原酶的应用
6.1、 皮革工艺的应用前景
胶原酶在皮革工业中有很多潜在的应用前景。皮革工业有浸水、脱脂、脱毛浸灰、脱灰、软化、浸酸、鞣制、中和、复鞣、染色加脂等诸多繁琐程序,并且使用化工材料繁杂,对环境有很多负面影响。皮革产业面临着开发环保高效制革工艺的巨大压力,以此满足现代污染和排放法规的要求。因此寻找皮革和皮革化学加工中的环保替代品是皮革工业面临的巨大挑战。在这方面,作为化学加工试剂替代品的酶法工艺获得了一致的推荐,因为它们不仅能生产出质量更好的产品,而且减少了危险和污染化学品的使用[48]。染色是皮革工业的一项重要工艺,而皮革工业废水中存在的未用完的染料往往对生物修复有抗性,这是一个重要问题。皮革鞣制后,人们发现使用微生物胶原酶会导致纤维状皮革网络的开放,促进染料在基质中的扩散,使用这种酶可以在保证皮革强度不变的情况下使皮革更柔软,而且染料的吸收率可达99%,同时也提升了皮革的丰满度、颗粒平滑度、手感和外观,所以它们被用作生物催化剂来改善染料的染色过程[49],减少了皮革废水中染料的排放。另外制革过程中的脱毛步骤也对环境造成了严重的污染,因此寻找生态友好的替代品是很有必要的。Sivasubramanian等人[50]提出了将微生物胶原酶用于脱毛工艺,有助于减轻污染负荷和胶原蛋白损伤。然而,微生物胶原酶的生产成本和适用性限制了其在脱毛过程中的商业应用。
6.2、 医药工业的应用
微生物胶原酶在医药工业中具有重要地位。胶原蛋白占人体蛋白质的三分之一,反映了它在健康和疾病中的特殊作用。微生物胶原酶已直接应用于临床治疗,如烧伤清创[51]、青光眼[52]、腰椎间盘突出[53]等。
掌筋膜挛缩症(DD病)是一种进行性发展的纤维素增殖性疾病,主要是掌筋膜增厚和手指腱膜增厚引起的掌指关节和指间关节的功能性障碍和屈曲挛缩。酶在DD患者中的首次应用是在1965年[54],当时使用的是胰蛋白酶和透明质酸酶的混合物。后来明确了沉积的蛋白是未成熟的Ⅲ型胶原蛋白后,使用胶原酶比筋膜切除手术更方便,因为它是具有胶原特异性的。2010年美国食品药品监督管理局(FDA)批准溶组织梭菌胶原酶可以用于DD病的治疗。在具体应用时,细胞水平上,控制胶原酶剂量和作用时间去抑制患者掌部成纤维细胞的扩散、附着和增殖,转录水平上,控制胶原酶剂量可对多种ECM成分、细胞因子和生长因子进行抑制[55]。Badalamente等人[56]严格遵循要求选择了35个成年患者,有的患者MP(掌指关节)挛缩程度在20°~100°之间,有的患者PIP(近侧指间关节)挛缩程度在20°~80°之间,排除使用四环素,胶原酶过敏等因素,给被选择的患者注射胶原酶(最多注射3次),最后跟踪记录了其中21名患者的恢复过程,以挛缩程度恢复到5°作为结点,注射1~24个月后,86%MP患者和100%PIP患者对治疗效果比较满意,且根据CMS评分达到79分可以说明胶原酶对DD患者治疗效果良好。
在一些研究中,患者认为接受胶原酶治疗和筋膜切开术没有差异,而且相比筋膜切开术,胶原酶的治疗成本较低[57]。Naam[58]的研究结果表明,使用胶原酶治疗的DD患者恢复工作或日常活动时间相比使用手术治疗的时间要短一些(胶原酶组对比手术组:1.9 d对比37.4 d)。另外还有许多报告研究证明:采用胶原酶治疗DD病后,复发率较低,严重不良事件并不多见。因此,采用注射胶原酶的方法对DD患者进行治疗是可行的,应予以鼓励。
6.3、 细胞分离方面的应用
细胞培养一般有原代培养、细胞菌株和细胞系等几种培养类型,其中酶经常用于在传代过程中保持体外细胞培养的组织分解和细胞解离[59]。胰蛋白酶通常用于哺乳动物细胞的组织解离和分离,在实验中被广泛用于重新悬浮附着在培养皿壁上的细胞的分离。贴壁细胞传代培养的首要步骤是当细胞达到高度融合时将细胞从基质中分离出来[60]。然而,胰蛋白酶在赖氨酸和精氨酸残基的羧基进行特异性裂解[61],其蛋白水解活性可能通过裂解细胞表面生长因子受体或膜蛋白而损伤细胞。因此,为了获得最大产量的活单细胞,选择适当的酶是至关重要的。另一方面,胶原含量高的纤维组织对胰蛋白酶水解具有抵抗力,所以在这些组织中使用胰蛋白酶是无效或有害的[62]。
微生物胶原酶在细胞纤维组织或敏感组织分离过程中很有价值[63]。Sun等人[64]通过0.01%微生物胶原酶和33%Percoll密度梯度离心从小鼠和人类肝脏样品中体外分离和纯化免疫细胞,使其具有更高的细胞产量和活力。Tanaka等人[65]设计了仅由催化结构域组成的重组62 k Da胶原酶进行细胞分离,具有很高的生产效率,这证明了这种重组胶原酶在生理条件下是稳定和活跃的,而且与来自溶组织梭菌的标准胶原酶产品相比,它具有更高的水解胶原蛋白活性,并能切割更多种类的胶原蛋白。
7 、结束语
目前,科学界对于胶原酶的各种名词定义、分类和鉴定都存在着巨大的争议。由于酶的本质是蛋白质,所以胶原酶在表征实验和参与反应的过程中易受温度、p H等条件的影响,在提取微生物胶原酶的过程中,也会有很多副产物,同时因为成本高也难以实现工业化。而在微生物胶原酶的应用上,医学领域应该做好临床监测,跟踪调研病人的恢复情况和复发率;在食品领域,需要寻找非致病菌株中产生重组胶原酶将其纯化,避免病毒存在妨碍这些酶在食品工业中的安全使用,而且未来的研究应该集中在生物活性胶原多肽、胶原水解物的生产和新型功能性食品的开发上;皮革领域需要在提高产品质量的同时还要重视环境和生态问题,采用非化学方式的生物、物理处理方式助推皮革产业的生态化。因此,筛选高效的产胶原酶的微生物菌株和开发低成本培养基以及通过不同的培养方法经济地生产具有新特性的胶原酶,对于皮革产业的生态化和持续发展也将具有十分重要的意义。
参考文献
[1] Adhikari A S,Glassey E,Dunn A R.Conformational dynamics accompanying the proteolytic degradation of trimeric collagen I by Collagenases[J].Journal of the American Chemical Society,2012,134(32):13259-13265.
[2] Bilek S E,Bayram S K.Fruit juice drink production containing hydrolyzed collagen[J].Journal of Functional Foods,2015,14:562-569.
[3] Lima C A,Freitas A C V,Lima J L,et al.Two-phase partitioning and partial characterization of a Collagenase from Penicillium aurantiogriseum URM4622:Application to collagen hydrolysis[J].Biochemical Engineering Journal,2013,75:64-71.
[4] Daboor S M,Budge S M,Ghaly A E,et al.Isolation and activation of Collagenase from fish processing waste[J].Advances in Bioscience and Biotechnology,2012,3 (3):191-203.
[5] Daboor S M,Budge S M,Ghaly A E,et al.Extraction and purification of Collagenase enzymes:A critical review[J].American Journal of Biochemistry and Biotechnology,2010,6(4):239-263.
[6] Jain R,Jain P C.Production and partial characterization of Collagenase of Streptomyces exfoliatus CFS 1068 using poultry feather[J].Indian Journal of Experimental Biology,2010,48(2):174-178.
[7] Kaur S P,Azmi W.Cost effective production of a novel Collagenase from a non-pathogenic isolate Bacillus tequilensis[J].Current Biotechnology,2013,2(1):17-22.
[8] Zuber M,Zia F,Zia K M,et al.Collagen based polyurethanes-A review of recent advances and perspective[J].International Journal of Biological Macromolecules,2015,80:366-374.
[9] 焦昀.羟基化修饰对胶原热稳定性影响研究[D].山东农业大学,2015.
[10] Gonzalez E A,Bielajew B J,Hu J C,et al.Engineering self-assembled neomenisci through combination of matrix augmentation and directional remodelin[J].Acta Biomaterialia,2020,109:73-81.
[11] Gharenaz N M,Movahedin M,Mazaheri Z.Three-dimensional culture of mouse spermatogonial stem cells using a decellularised testicular scaffold[J].Cell Journal,2020,21(4):410-418.
[12] Yuan J J,Zhou Q,Wang P,et al.Phosphorylation of silk fibroin via maillard reaction and its behavior of biomimetic mineralization[J].Fibers and Polymers,2019,20 (8):1616-1623.
[13] Jiang X J,Wang Y,Fan D C,et al.A novel human-like collagen hemostatic sponge with uniform morphology,good biodegradability and biocompatibility[J].Journal of Biomaterials Applications,2017,31(8):1099-1107.
[14] Slezak P,Monforte X,Ferguson J,et al.Properties of collagen-based hemostatic patch compared to oxidized cellulose-based patch[J].Journal of Materials Science-Materials in Medicine,2018,29(6):71.
[15] Yuan H B,Chen L,Hong F F.A biodegradable antibacterial nanocomposite based on oxidized bacterial nanocellulose for rapid hemostasis and wound healing[J].ACS Applied Materials&Interfaces,2020,12(3):3382-3392.
[16] Kim M,Hamilton S E,Guddat L W,et al.Plant Collagenase:unique colla enolytic activity of cysteine proteases from ginger[J].Biochimica Et Biophysica Acta-General Subjects,2007,1770(12):1627-1635.
[17] Raskovic B,Bozovic O,Prodanovic R,et al.Identification,purification and characterization of a novel collagenolytic serine protease from fig (Ficus carica var.Brown Turkey) latex[J].Journal of Bioscience&Bioengineering,2014,118(6):622-627.
[18] Santra M,Luthra-Guptasarma M.Assaying Collagenase activity by specific labeling of freshly generated N-termini with fluorescamine at mildly acidic p H[J].International Journal of Peptide Research and Therapeutics,2020,26(2):775-781.
[19] Gomez D E,Alonso D F,Yoshiji H,et al.Tissue inhibitors of Metalloproteinases:structure,regulation and biological functions[J].European Journal of Cell Biology,1997,74 (2):111-122.
[20] Rawlings N D,Barrett A J,Thomas P D,et al.The MEROPSdatabase of proteolytic enzymes,their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database[J].Nucleic Acids Research,2018,46(D1):D624-D632.
[21] Duarte A S,Correia A,Esteves A C.Bacterial Collagenases-A review[J].Critical Reviews in Microbiology,2016,42(1):106-126.
[22] Matsushita O,Jung C M,Katayama S,et al.Gene duplication and multiplicity of Collagenases in Clostridium histolyticum[J].Journal of Bacteriology,1999,181(3):923-933.
[23] Zhang Y Z,Ran L Y,Li C Y,et al.Diversity,structures and collagen-degrading mechanisms of bacterial collagenolytic proteases:An overview[J].Applied&Environmental Microbiology,2015,81(18):6098-6107.
[24] Obana N,Nomura N,Nakamura K.Structural requirement in Clostridium perfringens Collagenase m RNA 5'leader sequence for translational induction through small RNA-m R-NA base pairing[J].Journal of Bacteriology,2013,195(12):2937-2946.
[25] Ducka P,Eckhard U,Sch?nauer E,et al.A universal strategy for high-yield production of soluble and functional clostridial Collagenases in E.coli[J].Applied Microbiology and Biotechnology,2009,83(6):1055-1065.
[26] Sekiguchi H,Uchida K,Matsushita O,et al.Basic fibroblast growth factor fused with tandem collagen-binding domains from Clostridium histolyticum Collagenase col G increases bone formation[J].Biomed Research International,2018,2018:8393194.
[27] Kumar P G,Nidheesh T,Suresh P V.Comparative study on characteristics and in vitro fibril formation ability of acid and pepsin soluble collagen from the skin of catla (Catla catla)and rohu (Labeo rohita)[J].Food Research International,2015,76(3):804-812.
[28] Bauer R,Wilson J J,Philominathan S T L,et al.Structural comparison of col H and col G collagen-binding domains from Clostridium histolyticum[J].Journal of Bacteriology,2013,195(2):318-327.
[29] Eckhard U,Brandstetter H.Polycystic kidney disease-like domains of clostridial Collagenases and their role in collagen recruitment[J].Biological Chemistry,2011,392 (11):1039-1045.
[30] Rawlings N D,Waller M,Barrett A J,et al.Merops:the database of proteolytic enzymes their substrates and inhibitors[J].Nucleic Acids Reseaech 2014,42 (D1):D503-D509.
[31] Pal G K,Pv S.Microbial Collagenases:challenges and prospects in production and potential applications in food and nutrition[J].RSC Advances,2016,6(40):33763-33780.
[32] Miyoshi S.Vibrio vulnifificus infection and Metalloprotease[J].Indian Journal of Dermatology,2006,33:589-595.
[33] Kim S K,Yang J Y,Cha J.Cloning and sequence analysis of a novel Metalloprotease gene from Vibrio parahaemolyticus04[J].Gene,2002,283(1-2):277-286.
[34] Bauer R,Janowska K,Taylor K,et al.Supporting information for article:Structures of three polycystic kidney disease-like domains from Clostridium histolyticum Collagenase col G and col H[J].Acta Crystallographica.Section D:Biological Crystallography,2015,71:565-577.
[35] Thring T S A,Hili P,Naughton D P.Anti-Collagenase,anti-elastase and anti-oxidant activities of extracts from 21plants[J].BMC Complementary and Alternative Medicine,2009,9:27.
[36] Wu Q,Li C,Li C Y,et al.Purification and characterization of a novel Collagenase from Bacillus pumilus Col-J[J].Applied Biochemistry and Biotechnology,2010,160(1):129-139.
[37] Chen S H,Ma M H,Fu X.Analyzing structural and functional characteristics of Collagenase from Bacillus cereus MH19 via in silico approaches[J].Current Proteomics,2020,17(3):200-212.
[38] Tran L H,Nagano H.Isolation and characteristics of Bacillus subtilis CN2and its Collagenase production[J].Journal of Food Science,2002,67(3):1184-1187.
[39] Lima C A,Rodrigues P M B,Porto T S,et al.Production of a Collagenase from Candida albicans URM3622[J].Biochemical Engineering Journal,2009,43(3):315-320.
[40] Punta M,Coggill P C,Eberhardt R Y,et al.The Pfam protein families database[J].Nucleic Acids Reseacch,2012,40(D1):D290-D301.
[41] Abfalter C M,Schoenauer E,Ponnuraj K,et al.Cloning,purification and characterization of the Collagenase col A expressed by Bacillus cereus ATCC 14579[J].Plos One,2016,11(9):e0162433.
[42] Yoshida E,Noda H.Isolation and characterization of Collagenases I and II from Clostridium histolyticum[J].Biochimica Et Biophysica Acta,1965,105(3):562-574.
[43] Zhang Y F,Fu Y,Zhou S F,et al.A straightforward ninhydrin-based method for Collagenase activity and inhibitor screening of Collagenase using spectrophotometry[J].Analytical Biochemistry,2013,437(1):46-48.
[44] Eckhard U,Schoenauer E,Nüss D,et al.Structure of Collagenase G reveals a chew-and-digest mechanism of bacterial collagenolysis[J].Nature Structural&Molecular Biology,2011,18(10):1109-1114.
[45] Ducka P,Eckhard U,Sch觟nauer E,et al.A universal strategy for high-yield production of soluble and functional clostridial Collagenases in E.coli[J].Applied Microbiology and Biotechnology,2009,83(6):1055-1065.
[46] Ohbayashi N,Matsumoto T,Shima H,et al.Solution structure of lostridial Collagenase H and its calcium-dependent global conformation change[J].Biophysical Journal,2013,104(7):1538-1545.
[47] Eckhard U,Huesgen P F,Brandstetter H,et al.Proteomic protease specificity profiling of clostridial Collagenases reveals their intrinsic nature as dedicated degraders of collagen[J].Journal of Proteomics,2014,100:102-114.
[48] Kaushik R,Saran S,Isar J,et al.Statistical optimization of medium components and growth conditions by response surface methodology to enhance lipase production by Aspergillus carneus[J].Journal of Molecular Catalysis B-Enzymatic,2006,40(3-4):121-126.
[49] Kanth S V,Venba R,Madhan B,et al.Studies on the influence of bacterial Collagenase in leather dyeing[J].Dyes&Pigments,2008,76(2):338-347.
[50] Sivasubramanian S,Manohar B M,Rajaram A,et al.E-cofriendly lime and sulfide free enzymatic dehairing of skins and hides using a bacterial alkaline protease[J].Chemosphere,2008,70(6):1015-1024.
[51] Pham C H,Collier Z J,Fang M,et al.The role of Collagenase ointment in acute burns:a systematic review and meta-analysis[J].Journal of Wound Care,2019,28(2):S9-S15.
[52] Song M,Lee S,Choe D,et al.Clinical and biological evaluations of biodegradable collagen matrices for glaucoma drainage device implantation[J].Investigative Opthalmology&Visual Science,2017,58(12):5329-5335.
[53] Changhuai L U,Zhi J L,Hong B Z,et al.Mechanism of p38mitogen activated protein kinase signaling pathway on promoting the hypertrophy of human lumbar ligamentum flavum via transforming growth factorβ1/connective tissue growth factor[J].Chinese Journal of Reparative and Reconstructive surgery,2019,33(6):330-335.
[54] Bassot J.Treatment of Dupuytren’s(下转第27页)disease by isolated pharmacodynamic“exeresis”or“exseresis”completed by a solely cutaneous plastic step[J].Lille Chirurgical,1965,20:38-44.
[55] Warwick D,Arandes-Renu J M,Pajardi G,et al.Collagenase Clostridium histolyticum:emerging practice patterns and treatment advances[J].Journal of Plastic Surgery and Hand Surgery,2016,50(5):251-261.
[56] Badalamente M A,Hurst LC.Efficacy and safety of injectable mixed Collagenase subtypes in the treatment of Dupuytren's Contracture[J].Journal of Hand Surgery-American Volume,2007,32A(6):767-774.
[57] Nydick J A,Olliff B W,Garcia M J,et al.A comparison of percutaneous needle fasciotomy and Collagenase injection for Dupuytren Disease[J].Journal of Hand Surgery-American Volume,2013,38A(12):2377-2380.
[58] Naam N H.Functional outcome of Collagenase injections compared with fasciectomy in treatment of Dupuytren’s contracture[J].Hand (New York,N.Y.),2013,8 (4):410-416.
[59] Duarte A S,Rosa N,Duarte E P,et al.Cardosins:a new and efficient plant enzymatic tool to dissociate neuronal cells for the establishment of cell cultures[J].Biotechnology and Bio-(上接第22页)engineering 2007,97(4):991-996.
[60] Huang H L,Hsing H W,Lai T C,et al.Trypsin-induced proteome alteration during cell subculture in mammalian cells[J].Journal of Biomedical Science,2010,17:36.
[61] Olsen J V,Ong S E,Mann M.Trypsin cleaves exclusively C-terminal to arginine and lysine residues[J].Molecular&Cellular Proteomics,2004,3(6):608-614.
[62] Yu Z X,Visse R,Inouye M,et al.Defining requirements for Collagenase cleavage in collagen type III using a bacterial collagen system[J].Journal of Biological Chemistry,2012,287(27):22988-22997.
[63] Santra M,Luthra G M.Assaying Collagenase activity by specific labeling of freshly generated N-Termini with fluorescamine at mildly acidi p H[J].Inrernational Journal of Peptide Research and Therapeutic,2020,26(2):775-781.
[64] Sun C,Luo Q B,Lu X X,et al.Isolation and purification of primary Kupffer cells from mouse liver[J].Chinese Journal of Cellular&Molecular Immunology,2016,32(8):1021-1025
[65] Tanaka K,Okitsu T,Teramura N,et al.Recombinant Collagenase from Grimontia hollisae as a tissue dissociation enzyme for isolating primary cells[J].Scientific Reports,2020,10(1):3927.





