摘要:核酸结构设计因简单的碱基配对法则而获得预测结构的能量支持,且具备更强的折叠合成结构,受到了广泛的关注.然而,核酸碱基的化学多样性缺乏,使得核酸结构在功能上多样性比蛋白质弱,因而限制了其在实际中的应用.本文聚焦核酸分子工程,尤其是对核酸结构以及分子间相互作用的研究;基于核酸结构的空间可寻址性,实现了多种材料在核酸结构上的位点可控的修饰;基于对核酸分子序列的设计,还实现了核酸分子间反应动力学参数的连续微调.此外,设计构建了合成分子化学反应网络、分子机器及核酸基生物材料,并将这种绿色生物材料应用于生物分子识别、生物膜表面工程及生物催化等方向.
关键词:合成核酸分子工程;生物分子识别;细胞表面工程;生物催化;
Abstract:The simple rule of base-pairing of nucleic acids enables nucleic acid structures to be designed via powerful energy-based prediction tools. Thus, nucleic acid structures have attracted considerable attention owing to their ability to fold into a variety of synthetic structures. However, the lack of chemical persity of nucleic acid bases makes nucleic acid structures less functionally perse than proteins, thereby limiting their practical applications. This review focuses on the underlying technology of nucleic acid molecular engineering, especially on the studies of nucleic acid structures and their molecular interactions. As nucleic acid structures are fully spatially addressable, a persity of particles could be linked to designated positions on the surface of nucleic acid structures. Additionally, the intermolecular reaction kinetics of nucleic acids could be continuously fine-tuned by rational design of nucleic acid sequences. This review also summarizes the development of synthetic molecular networks, dynamic molecular machines, and nucleic acid-based biomaterials, as well as the application of these as green biomedical devices in biomolecular recognition, cell surface engineering, and biocatalysis.
Keyword:synthetic nucleic acid molecular engineering; biomolecular recognition; cell surface engineering; biocatalysis;
0 引言
核酸分子作为自然界生物遗传信息的载体,具有独特的化学或结构特性,例如DNA双链遵循Watson-Crick碱基互补配对原则、序列可编程性和优异生物相容性等[1,2].同时,利用限制性核酸酶、连接酶和聚合酶,研究人员能够实现对核酸分子的剪切、连接、复制和延伸等操作[1,2].近年来,核酸分子合成技术的不断进步,以及其价格的降低,促进了核酸纳米技术领域的迅速发展[3,4].核酸纳米技术可大致分为结构纳米技术和动态纳米技术[5].一方面,结构纳米技术通过核酸分子自下而上的自组装,实现了不同尺寸的二维和三维结构的构建[6].这些结构在纳米尺度上具有空间可寻址性,可以在体外对生物分子进行位点特异性锚定[5,7,8].另一方面,动态核酸纳米技术基于可编程的核酸分子间相互作用或分子内构象变化,可以构建具有自主移动或处理信息能力的分子马达或电路[5,7].对核酸分子反应动力学的定量研究,能够预测单链核酸分子的折叠路径以及核酸分子间的反应路径,这是核酸纳米技术取得成功的一个关键因素[9].长期以来,核酸纳米技术取得的进步推动了多个领域的发展,例如基因编辑[10]、智能药物递送[11,12,13,14]和绿色催化[4]等.这些进步使人们看到了核酸分子器件在“智能疗法”中的巨大应用潜力,尤其是用于生物诊断.已经有研究表明,利用核酸分子器件能够对多个生物标志物进行同时检测,并实现准确、快速的疾病诊断[15,16].目前,已经发展的多种体外诊断(in vitro diagnostics,IVD)方法也涉及合成核酸分子器件,例如基于DNA折纸和人工基因电路的埃博拉病毒检测试纸[17].这些能够用于护理点的简单、快速的体外诊断方法的涌现,揭示了合成DNA分子潜在的经济价值.
本文总结了合成核酸分子工程在生物医学应用中的进展.首先,简要概述了对核酸结构的合理设计和修饰,描述了对核酸分子间反应和分子运动的动力学的调控方法,阐述了功能切换电路的设计.然后,总结了合成核酸分子工程3个方向的应用:利用合成核酸分子的生物分子识别能力,构建核酸传感器,实现生物标志物的检测和疾病的诊断;核酸分子电路用于细胞表面工程化,以实现对细胞行为的调控;在生物催化中,核酸分子用于构建模拟酶和优化酶的催化活性.最后,提出了合成核酸分子工程当前面临的瓶颈,并指出了其未来的发展方向.
1 对核酸结构以及分子间相互作用的研究
自20世纪80年代Seeman设计并构建了第一个DNA纳米结构以来[18],自组装DNA纳米结构不断涌现.2006年DNA折纸的问世[19],在结构核酸纳米技术领域具有里程碑的意义.在数百条短的订书钉链的辅助下,一条长的DNA支架链折叠形成DNA折纸结构(图1(a))[19,20,21,22,23,24,25].基于DNA分子的序列可编程性和互补链间的程序化杂交,通过上述方法,理论上能够构建任意复杂形状的二维、三维结构,如笑脸、立方体和半球形等.平面DNA折纸结构的尺寸受到支架链的长度的限制,通常为8 000~10 000 nm2.通过合理的设计,多个DNA折纸结构能够作为模块进行拼接,合成的结构能够达到毫米尺度.在DNA折纸结构中,每一条订书钉链经过设计,其位置可轻易确定.因此,折纸结构具有空间可寻址能力,可以将修饰分子锚定在指定的位置.每40~50 nm2内存在1个可寻址点.
基于其空间可寻址的特性,研究人员能够从设计层面上改造DNA折纸结构.在DNA折纸结构上,通过找到预设的可寻址点或具有一定间距的多个可寻址点所在的订书钉链,可对其进行功能化修饰[8].通过对订书钉链的修饰,诸如延伸出捕获链或连接生物素,多种分子或结构能够被锚定在DNA折纸结构表面,使其获得新的功能(图1(b))[8,11,26].例如,通过精确控制捕获链在棒状DNA折纸结构上的位置,可实现对平行金纳米棒簇三维空间配置的控制[27].
在结构核酸纳米技术发展的同时,动态核酸纳米技术也取得了巨大的进步.基于DNA链置换反应,AND和OR等基础逻辑门以及DNA分子电路等计算器件被构建.链置换反应的过程:在toehold区域的触发下,一条DNA单链取代DNA双链中的一条链,并与另一条链杂交.通过调整toehold区域的长度和序列,可实现对链置换反应速率6个数量级范围的调控.在此基础上,本文课题组提出了精细调控DNA链置换反应动力学的策略[28].在这一策略中,能够发生分子内构象变化的发夹结构作为分子开关模块被插入到toehold区域与分支迁移域之间,通过调控发夹结构茎和环的长度实现了链置换反应动力学的连续微调.进一步利用协同变构的DNA开关,设计了DNA I/O(input/output)控制器,实现了DNA链置换反应动力学的非线性调控,希尔系数可达2.70[29].
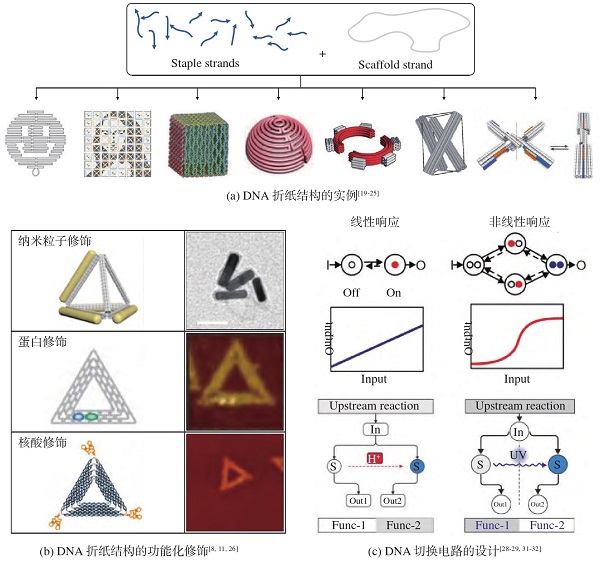
图1 核酸纳米技术的发展
通过对序列的合理设计,上游DNA链置换反应的产物能够作为输入,用来触发下游反应的发生,从而实现反应的级联.基于此,研究人员设计并构建了具有特定功能的DNA电路[30].将DNA电路进一步与刺激响应材料结合,构建了对外界刺激具有响应能力的DNA功能切换电路(图1(c)).该工作以光控DNA功能切换电路作为一个经典的例子[31],将紫外光(Ultraviolet,UV)响应的光致变色分子(螺吡喃)引入到反应体系中.在初始状态下,三链体结构的DNA开关能够与输入链发生反应;在紫外光照射下,螺吡喃发生变构并诱导三链体结构转化为双链结构,通过阻碍与输入链的反应实现功能的转化.此外,研究人员还设计并构建了p H响应的功能切换电路[32,33].
目前,研究人员将动态DNA纳米技术与静态DNA纳米技术相结合,实现了具有动态行为的分子机器,如能够开闭的DNA六边形桶状结构[34]、局域化分子电路[35]及DNA步行者[36]等.
2 在生物医学领域的应用
基于其优异的生物相容性和低细胞毒性,核酸分子在体内应用中显示出独特的优势[37].结合其易于修饰、可编程等特点,基于核酸的分子机器在生物医学领域表现出极大的应用潜力.本文从3个方面来简述合成DNA分子工程的应用潜力:生物分子识别、生物膜表面修饰和生物催化.
2.1 生物分子识别
生物分子识别依赖于识别元件和靶标之间的特异性结合[38,39].经核酸适配体或官能团修饰,核酸结构能够作为识别元件靶向多种目标,包括核酸、蛋白、离子,甚至整个细胞.此类具有生物分子识别能力的DNA结构可以应用于生物传感等领域[40,41,42,43,44,45,46,47,48,49,50,51,52,53,54,55,56,57,58,59,60].
受马达蛋白的启发,研究人员创造出了人工分子机器人—DNA步行者.其中,行走元件能够在驱动力的作用下沿着步行轨道稳定、渐进地运动[2].将DNA识别元件与步行者的行走元件相结合,识别元件捕获靶标后,引发信号的产生,随着步行者的多步行走,进一步实现信号的放大,这一过程可用于构建高灵敏生物传感器.基于此,本文课题组设计并构建了一种随机DNA步行者,它以一条DNA单链作为行走元件,以核酸外切酶Ⅲ为驱动力,在DNA链修饰的金纳米粒子上移动[61].当行走元件结合到一条轨道链上之后,轨道链会在酶的作用下被水解,从而产生信号.而释放的行走元件会结合到下一条轨道链上,通过多次循环实现信号放大.在此基础上,进一步构建了双步行者系统,目标细菌能够与识别元件结合,从而释放两类行走链,分别触发两类步行者上信号的产生及扩增,实现快速灵敏的生物传感[62].此外,有研究报道使用双链特异性核酸酶作为驱动力,以吸附在Mo S2纳米片上的DNA发夹作为行走轨道,发展了能够用于mi RNA识别和检测的RNA步行者[63,64].更进一步地,基于单壁碳纳米管的RNA步行者也被开发并应用于mi RNA识别和胞内成像[65].为了实现对靶标的超高通量检测,研究人员将随机DNA步行者与基于液滴的微流体策略结合,采用基于荧光种类和强度的二维编码策略,构建了具有超多重检测能力的生物传感器,理论上可以识别和区分9种细菌的511种不同组合(图2)[66].
![随机DNA步行者-液滴平台用于细菌的超多重检测[66]](http://www.xueshut.com/uploads/allimg/230307/2-23030G64441305.jpg)
图2 随机DNA步行者-液滴平台用于细菌的超多重检测[66]
2.2 生物膜表面修饰
细胞膜是细胞与其环境之间发生交互的主要介质[67].开发细胞膜上的分子工程策略可以用于调节细胞与环境之间的交互作用.目前,调控细胞膜表面受体的常用工具是基因编辑.基因编辑是一种能够对生物基因组进行修饰的强有力的工具,利用编辑工具对基因“改写”,从而改变细胞膜表面受体的表达.但这种方法操作复杂,存在脱靶的可能性,并且可能会造成基因组结构突变.相比于基因编辑,利用细胞表面工程也能够实现相似的功能,并且可规避上述问题.研究人员已经发展了各种策略来实现细胞表面工程[68].例如,利用合成细胞黏附肽(arginylglycylaspartic acid,RGD)将DNA电路修饰在细胞膜表面,通过改变输入模式可调控细胞在载玻片表面的黏附和分离行为(图3(a))[69].此外,基于杂交链式反应,研究人员发展了一种基于DNA电路的细胞表面工程策略,可以对细胞-细胞相互作用进行可编程调控,从而选择性地控制多细胞自组装路径(图3(b))[70].近期,基于DNA概率电路,研究人员提出了一种可精确调节细胞膜上的人工受体聚集程度的策略(图3(c)),这一策略可以调控自然杀伤细胞与癌细胞之间相互作用,从而促进有效的癌细胞杀伤[71].该研究为精确控制细胞识别和发展基于细胞的免疫治疗提供了有效工具.除对细胞膜外表面进行人工核酸分子修饰之外,基于核酸的细胞膜内表面工程化进一步扩展了功能性核酸分子在细胞生物学中的应用,这一方面的研究也引起了广泛关注.例如,邢航课题组提出了一种基于融合性球形核酸的可在空间上控制膜表面工程化的仿生策略[72].依赖这一膜融合策略,可以精准控制DNA链在细胞膜内表面或外表面的高效修饰,并以此实现了对细胞间通信的调节以及细胞内代谢物的监测.
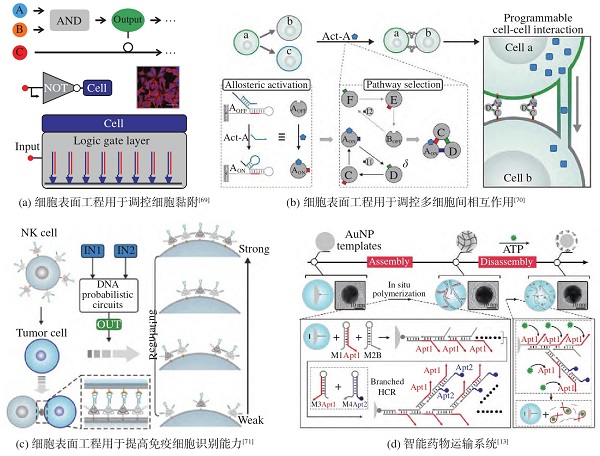
图3 细胞表面工程和智能药物运输
适配体等靶向元件能够与细胞膜上蛋白发生特异性相互作用,将靶向元件与药物载体结合,构建智能药物递送系统,这类系统能够特异性识别肿瘤细胞,实现药物的靶向递送,从而提高药物的运输效率、降低药物的全身毒性.基于此,研究人员开发了一种DNA开关调控的水凝胶,用于控制肿瘤杀伤药物在特定癌细胞中的释放[13].体系以金纳米粒子作为模板,DNA链在模板表面发生杂交链式反应,形成球形核酸水凝胶.抗癌药物能够插入到DNA双链当中.当识别到靶细胞中ATP(adenosine triphosphate)时,DNA双链解旋并释放药物(图3(d)).此外,适配体修饰的DNA折纸结构[11]和金属有机框架(metal-organic framework,MOF)-生物矿化DNA纳米球[12]也被开发用于智能药物运载系统.
2.3 生物催化
生物催化是在酶或生物有机体等催化剂的作用下进行化学转化的过程,它有助于合成化学品、药物和生物燃料等,对于生物医学等领域的发展至关重要.生物催化是一种环境友好和经济有效的催化方法,有望替代传统金属催化方法.常见的生物酶包括蛋白、DNA和RNA.为了实现高效的生物催化,Zhong等[73]发展了一种具有与酶相似活性的新型DNA纳米纤维水凝胶(G4模拟酶),可用于H2O2和四甲基联苯胺的氧化还原催化(图4(a)).G4模拟酶通过鸟嘌呤核苷与KB(OH)4自组装,并在自组装过程中插入血红素,具有显着的类酶活性.
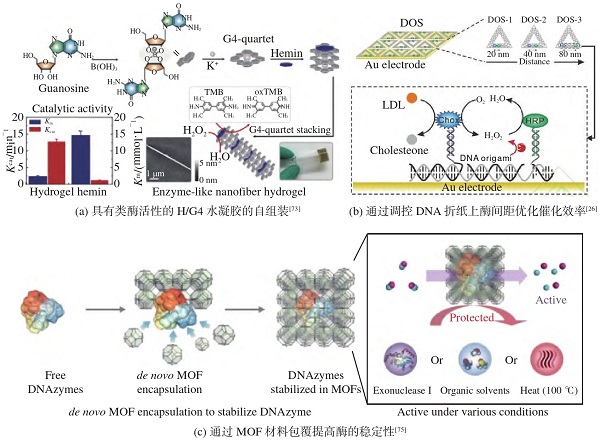
图4 基于合成核酸分子的生物催化
设计构建高效的生物催化剂可以加速化学反应.然而,在工业化应用中,生物催化剂还需要满足两个重要条件:高化学转化速率和对极端条件的耐受性(包括高温或低温,有机溶剂和抑制性试剂等).研究人员通过调控两个酶分子的间距有效地提高了级联催化反应的转化率(图4(b))[26].基于DNA折纸结构的可编程性和空间可寻址性,两种酶以不同的间距被锚定在平面核酸结构上,以优化酶级联催化反应的效率.此外,研究人员还使用多壳金属有机框架,在纳米尺度上对酶进行空间组装,提高酶的级联催化活性[74].这种金属有机框架还能够作为区室化的反应器,有效避免反应串扰.另一方面,使用MOF封装脱氧核糖核酸酶(DNAzymes),可以提高酶的稳定性,使其在有机溶剂或高温条件下依旧具有良好的酶催化活性(图4(c))[75].
3 结论
本文总结了合成核酸分子工程在生物医学应用中的进展.本研究专注于核酸分子底层技术,尤其是对静态核酸结构的合理设计和修饰,以及对核酸分子间反应和分子运动动力学的调控方法,并且进一步展示了功能切换电路的设计.最后阐述了合成核酸分子工程用于生物分子识别、生物膜表面工程和生物催化这3个方向的研究进展.
合成核酸分子工程的蓬勃发展为生物医学领域带来了巨大的机遇,但仍然存在一些障碍和发展瓶颈.主要包括:(1)受到稳定性和体内分子串扰的影响,目前功能化DNA分子虽然能够识别广泛的靶标,但在生物体液中的检测能力仍然有限.为了解决这一问题,研究人员将支架结构引入到生物检测当中[76],以提高体系的稳定性.(2)基于DNA的药物运输系统存在细胞摄取率不高、细胞摄取的机制不明确等问题.为此,研究人员针对药物载体的种类、尺寸和形状等对细胞摄取的影响进行了研究[77].(3)当核酸分子工程应用于生物领域时,需要在体系中加入特定的工具酶或者对探针链进行荧光标记,这无疑会增加体系的成本.为了解决这一问题,非常有必要发展等温非酶扩增方法和无标记方法.(4)对于DNA纳米技术而言,当DNA电路规模扩展到一定程度或DNA纳米结构大到一定尺度时,信号泄露和DNA链错误折叠的概率都会急剧增加,这会导致电路的错误输出,并且影响合成结构的产率和完整性.因此,改进和完善DNA电路或结构的设计和制备技术,对于构建更复杂的DNA电路和组装更复杂的DNA结构具有重要的意义,也是未来核酸分子工程的发展方向.
尽管核酸分子工程仍面临诸多挑战,但不可否认的是,其在生物医学领域发挥着越来越重要的作用,尤其是生物分子的检测以及疾病的诊断和治疗方面.同时,研究人员也在不断拓展核酸分子工程在热点领域(如生物安全、表观遗传学等)的应用.相信未来经过几十年的发展,在先进功能性材料的推动下,DNA设计、合成、修饰及组装技术的发展将达到一个新的水平,有望应用于更广泛的领域.
参考文献
[1] LIU S,JIANG Q,WANG Y,et al. Biomedical applications of DNA-based molecular devices[J] Advanced Healthcare Materials, 2019, 8(10):e1801658.
[2] CHEN J,LUO Z,SUN C,et al.Research progress of DNA walker and its recent applications in biosensor[J]. TrAC Trends in Analytical Chemistry,2019, 120:115626.
[3] PAYNE R J,WINSSINGER N.Editorial overview: Synthetic biomolecules[J]. Current Opinion in Chemical Biology,2018. 46:A3-A4.
[4] ZHANG D Y,SEELIG G.Dynamic DNA nanotechnology using strand-displacement reactions[J].Nature Chemistry,2011 ,3(2):103-113.
[5] SEEMAN N C,SLEIMAN H F.DNA nanotechnology[J].Nature Reviews Materials,2017,3(1):1-23.
[6] HUANG P,WANG J,JIAO L,et al. A*time frozen“technique in microchannel used for the thermodynamic studies of DNA origami[J].Biosensors and Bioelectronics ,2019, 131:224-231.
[7] DELUCA M,SHI Z,CASTRO C E,et al.Dynamic DNA nanotechnology: Toward functional nanoscale devices[J].Nanoscale Horizons,2020,5(2):182-201.
[8] MAN T,JI W,LIU X,et al.Chiral metamolecules with active plasmonic transition[J] ACS Nano,2019,13(4):4826-4833.
[9] ZHU D,SONG P,SHEN J,et al.Polya-mediated DNA assembly on gold nanoparticles for thermodynamically favorable and rapid hybridization analysis[J] Analytical Chemistry,2016 ,88(9):4949-4954.
[10] SUN W,JI W,HALL J M,et al.Self-assembled DNA nanoclews for the efficient delivery of CRISPR-Cas9 for genome editing[J] Angewandte Chemie-International Edition,2015. 54(41): 12029-12033.
[11] CAO M,SUN Y,XIAO M,et al.Multivalent aptamer-modified DNA origami as drug delivery system for targeted cancer therapy[J]. Chemical Research in Chinese Universities 2019,36(2):254-260.
[12] WANG P,XIAO M,PEI H,et al.Biomineralized DNA nanospheres by metal organic framework for enhanced chemodynamic therapy[J].Chemical Engineering Journal,2021 ,415:129036.
[13] XIAO M,LAl W,WANG F,et al.Programming drug delivery kinetics for active burst release with DNA toehold switches[J].Journal of the American Chemical Society,2019, 141(51):20354-20364.
[14] JI W,LI X,XIAO M,et al.DNA-scaffolded disulfide redox network for programming drug-delivery kinetics[J].Chemistry-AEuropean Journa1,2021,27(34):8745-8752.
[15] LOPEZ R,WANG R,SEELIG G.A molecular multigene classifier for disease diagnostics[J].Nature Chemistry,2018, 10(7):746-754.
[16] ZHANG C,ZHAO Y,XU X,et al.Cancer diagnosis with DNA molecular computation[J] Nature Nanotechnology, 2020, 15(8):709-715.
[17]张浩千,娄春波台成生物学的发展、挑战及应对[J]科学与社会,2014 .4(4):26-33.
[18] KALLENBACH N R,MA R I,SEEMAN N C. An immobile nucleic acid junction constructed from oligonucleotides[J].Nature, 1983 ,305(5937):829-831.
[19] ROTHEMUND P W.Folding DNA to create nanoscale shapes and patterns[J].Nature. 2006, 440(7082):297-302.
[20] TIKHOMIROV G,PETERSEN P,QIAN L.Fractal assembly of micrometre-scale DNA origami arrays with arbitrary patterns[J].Nature,2017,552(7683):67-71.
[21] ANDERSEN E S,DONG M,NIELSEN M M,et al.Self-assembly of a nanoscale DNA box with a controllable lid[J]. Nature, 2009 459(7243):73-76.
[22] DIETZ H,DOUGLAS S M,SHIH W M.Folding DNA into twisted and curved nanoscale shapes[J] Science ,2009,325(5941):725-730.
[23] LIEDL T,HOGBERG B.TYTELL J,et al. Self-assembly of three-dimensional prestressed tensegrity structures from DNA[J]. Nature Nanotechnology,2010,5(7):520-524.
[24] HAN D,PAL S,NANGREAVE J,et al.DNA origami with complex curvatures in three-dimensional space[J] Science, 2011 ,332(6027):342-346.
[25] GERLING T,WAGENBAUER K F,NEUNER A M,et al.Dynamic DNA devices and assemblies formed by shape-complementary,non-base pairing 3D components[J] Science ,2015,347(6229).1446-1452.
[26]余紫荆.肖明书,裴昊,等基于DNA折纸的酶级联催化用于低密度脂蛋白的电化学检测[J.分析化学,2022 ,50(6):850-858.
[27] YU H,MAN T,JI W.et al. Controllable self-assembly of parallel gold nanorod clusters by DNA origami[J.Chinese Chemical Letters,2019.30(1):175-178.
[28] LAI W,REN L,TANG Q,et al.Programming chemical reaction networks using intramolecular conformational motions of DNA[J].ACS Nan0,2018,12(7):7093-7099.
[29] LAI W,XIONG X,WANG F,et al.Nonlinear regulation of enzyme-free DNA circuitry with ultrasensitive switches[J] ACSSynthetic Biology,2019,8(9):2106-2112.
[30] XIONG X,ZHU T,ZHU Y,et al.Molecular convolutional neural networks with DNA regulatory circuits[J] Nature Machine Intelligence .2022(4):.625-635.
[31] XIONG X.XIAO M,LAI W,et al.Optochemical control of DNA-switching circuits for logic and probabilistic computation[J] Angewandte Chemie-International Edition,202 1,60(7):3397-3401.
[32] TANG Q,LAI W,WANG P,et al.Multi-mode reconfigurable DNA-based chemical reaction circuits for soft matter computing and control[J]Angewandte Chemie-International Edition,2021,60(27):15013-15019.
[33] JI W,LI D,LAI W,et al.pH-operated triplex DNA device on MoS2 nanosheets[J].Langmuir,2019,35(14):5050-5053.
[34] DOUGLAS S M,BACHELET 1,CHURCH G M J S.A logic-gated nanorobot for targeted transport of molecular payloads[J].Science,2012,335(6070)-831-834.
[35] CHATTERJEE G,DALCHAU N,MUSCAT R A,et al.A spatially localized architecture for fast and modular DNA computing[J] Nature Nanotechnology,2017,12(9):920-927.
[36] THUBAGERE A J,LI W,JOHNSON R F,et al.A cargo-sorting DNA robot[J].Science ,2017,357(6356):1095-1096.
[37] XIAO M, GAO L,CHANDRASEKARAN A R,et al. Bio-functional G-molecular hydrogels for accelerated wound healing[J].Materials Science and Engineering:C ,2019, 105:110067.
[38] MAN T,LAI W,ZHU C,et al.Perovskite mediated vibronic coupling of semiconducting SERS for biosensing[J]. Advanced Functional Materials ,2022 ,32(32):2201799.
[39] TANG Q,PLANK T N,ZHU T,et al.Self-assembly of metallo-nucleoside hydrogels for injectable materials that promote wound closure[J].ACS Applied Materials&Interfaces ,2019, 11(22):19743-19750.
[40] WANG X,YAN L,YU Z,et al.Aptamer-functionalized fractal nanoplasmonics-assisted laser desorption/ionization mass spectrometry for metabolite detection[J] .ChemPlusChem,2022,87(1):e202100479.
[41] SONG L,XIAO M,L AI W,et al. Intracellular logic computation with framework nucleic acid-based circuits for mRNA imaging[J].Chinese Journal of Chemistry,2021 ,39(4):947-953.
[42] WANG X,XIAO M,ZOU Y,et al.Fractal SERS nanoprobes for multiplexed quantitative gene profiling[J]. Biosensors and Bioelectronics ,2020,156:112130.
[43] YU H,XIAO M,LAI W,et al.A self-calibrating surface-enhanced Raman scattering-active system for bacterial phenotype detection[J] .Analytical Chemistry,2020,92(6):4491-4497.
[44] LAI W,XIAO M,YANG H,et al.Circularized blocker-displacement amplification for multiplex detection of rare DNA variants[J].Chemical Communications ,2020 ,56(82):12331-12334.
[45] MAN T,LAI W,XIAO M,et al.A versatile biomolecular detection platform based on photo-induced enhanced Raman spectroscopy[J].Biosensors and Bioelectronics, 2020, 147:111742.
[46] XIAO M,LAI W,MAN T,et al.Rationally engineered nucleic acid architectures for biosensing applications[J] Chemical Reviews,2019,119(22):11631-11717.
[47] SHEN J,LIANG L,XIAO M,et al.Fractal nanoplasmonic labels for supermultiplex imaging in single cells[J] Journal of the American Chemical Society,2019, 141(30):11938-11946.
[48] SU Y,LI D,LIU B,et al. Rational design of framework nucleic acids for bioanalytical applications[J]. ChemPlusChem,2019 ,84(5):512-523.
[49] QU X,XIAO M,LI F,et al.Framework nucleic acid-mediated pulldown microRNA detection with hybridization chain reaction amplification[J].ACS Applied Bio Materials,2018, 1(3):859-864.
[50]王飞.钟睿博,唐倩等ATP触发快速响应的线性DNA凝胶[J].高分子学报.2018(5):553-558.
[51] WANG X,LAI W,MAN T,et al. Bio-surface engineering with DNA scaffolds for theranostic applications[J]. Nanofabrication,2018,4(1):1-16.
[52] ZHONG R,XIAO M,ZHU C,et al.Logic catalytic interconversion of G-molecular hydroge[J] ACS Applied Materials&Interfaces,2018,10(5):4512-4518.
[53] XIAO M,QU X,LI L,et al.Ultrasensitive detection of metal ions with DNA nanostructure[J].Methods in Molecular Biology,2018,1811:137-149.
[54] QU X,LI M,ZHANG H,et al.Real-time continuous identification of greenhouse plant pathogens based on recyclable microfluidic bioassay system[J]. ACS Applied Materials&Interfaces,2017.9(37):31568-31575.
[55] QI L,XIAO M,WANG X,et al.DNA-encoded Raman-active anisotropic nanoparticles for microRNA detection[J] .Analytical Chemistry,2017,89(18):9850-9856.
[56] QU X,YANG F,CHEN H,et al.Bubble-mediated ultrasensitive multiplex detection of metal ions in three-dimensional DNAnanostructure-encoded microchannels[J] ACS Applied Materials&Interfaces , 2017,9(19):16026-16034.
[57] QU X.ZHANG H,CHEN H,et al.Convection-driven pull-down assays in nanoliter droplets using scaffolded aptamers[J]. Analytical Chemistry,2017,89(6):3468-3473.
[58] CHEN L,CHAO J,QU X,et al.Probing cellular molecules with polyA-based engineered aptamer nanobeacon[J].ACS Applied Materials&Interfaces , 2017 ,9(9):8014-8020.
[59] QI L,XIAO M,WANG F,et al.Poly-cytosine-mediated nanotags for SERS detection of Hg2+[J].Nanoscale,2017.9(37):14184-14191.
[60]闫璐李晓丹,肖明书,等.集成DNA分子机器的微针贴片用于细胞外三磷酸腺苷原位监测[J].分析化学,2022,50(4):516-524.
[61] QU X,ZHU D,YAO G,et al.An exonuclease I -powered,on-particle stochastic DNA walker[J] Angewandte Chemie-International Edition,2017 , 56(7):1855-1858.
[62] YANG H,XIAO M,L AI W,et al.Stochastic DNA dual-walkers for ultrafast colorimetric bacteria detection[J] Analytical chemistry,2020,92(7):4990-4995.
[63] XIAO M,MAN T,ZHU C,et al.MoS2 nanoprobe for microRNA quantification based on duplex-specific nuclease signal amplification[J].ACS Applied Materials&Interfaces ,2018, 10(9):7852-7858.
[64] XIAO M,CHANDRASEKARAN A R,JI W,et alAffinity-modulated molecular beacons on MoS2 nanosheets for microRNA detection[J] ACS Applied Materials&Interfaces,2018, 10(42):35794-35800.
[65] XIAO M,WANG X,LI L,et al.Stochastic RNA walkers for intracellular microRNA imaging[J] .Analytical Chemistry,2019,91(17):11253-11258.
[66] XIAO M,ZOU K,LI L,et al.Stochastic DNA walkers in droplets for super-multiplexed bacterial phenotype detection[J] .Angewandte Chemie International Edition,2019,58(43):15448-15454.
[67] WU Z.XIAO M,LAI W,et al.Nucleic acid-based cell surface engineering strategies and their applications[J].ACS Applied Bio Materials,2022, .5(5):1901-1915.
[68] XIAO M,LAI W,WANG X,et al.DNA mediated self-assembly of multicelllar microtissues[J].Microphysiological Systems ,2018,2(1).DOI:10.21037/mps.2017.12.01.
[69] QU X,WANG S,GE Z.et al.Programming cell adhesion for on-chip sequential boolean logic functions[J].Journal of the American Chemical Society,2017, 139(30):10176-10179.
[70] XIAO M,L AI W,YU H,et al.Assembly pathway selection with DNA reaction circuits for programming multiple cell-cell interactions[J].Journal of the American Chemical Society,2021, 143(9):3448-3454.
[71] XIAO M,LAI W,YAO X,et al.Programming receptor clustering with DNA probabilistic circuits for enhanced natural killer cell recognition[J] Angewandte Chemie-International Edition,2022 ,61(28):e202203800.
[72] LIN M,CHEN Y,ZHAO S,et al.A biomimetic approach for spatially controlled cell membrane engineering using fusogenic spherical nucleic acid[J]. Angewandte Chemie -International Edition,2022,61(1):e202111647.
[73] ZHONG R,TANG Q,WANG S,et al.Self-assembly of enzyme-like nanofibrous G-molecular hydrogel for printed flexible electrochemical sensors[J]. Advanced Materials ,2018,30(12)-:e1706887.
[74] MAN T,XU C,LIU X Y,et al.Hierarchically encapsulating enzymes with multi-shelled metal-organic frameworks for tandem biocatalytic reactions[J]. Nature Communcation,2022 13:305.
[75] ZHONG H,LO W S,MAN T,et al.Stabilizing DNAzymes through encapsulation in a metal-organic framework[J].Chemistry-AEuropean Journal,2020,26(57):12931-12935.
[76] ZHOU L,GAO M.FU W,et al.Three-dimensional DNA tweezers serve as modular DNA intelligent machines for detection and regulation 0f intracellular microRNA[J] Science Advances, 2020,6(22):eabb0695.
[77] CHAO J,LIU H,SU S,et al.Structural DNA nanotechnology for itelligent drug delivery[J] small,2014,10(22):4626-4635.





