摘 要: 生鲜肉的腐败是一个与多种特定微生物密切相关并受多种外界环境因素影响的生态现象。假单胞菌属是生鲜肉中的优势腐败菌之一。但是假单胞菌属在生鲜肉的致腐机制仍然缺乏较为系统的总结,尤其是对影响肉的腐败的相关基因调控及表达尚未有定论。因此本文首先概述了假单胞菌的特性、分类及种间差异,进而分析了假单胞菌的污染来源,最后从底物利用(糖代谢、氨基酸代谢、脂代谢等)角度分析了假单胞菌基因水平上的致腐原因,其中调控ED途径中相关酶类的基因、调控胞外蛋白酶的AprX基因和编码ABC转运蛋白的基因、编码脂肪酶lipA的基因在不同条件下和不同菌类中表达量的差异,可能是导致假单胞菌腐败的根本原因。通过本文的总结,以期为揭示肉品中假单胞菌的腐败机制并为控制腐败提供理论基础。
关键词: 肉; 腐败; 假单胞菌; 机制;
Abstract: Spoilage of raw meat is an ecological phenomenon closely related to many specific microorganisms and spoilage is affected by many external environmental factors. Pseudomonas spp. are one of is the dominant spoilage microorganism in raw meat. However, the mechanism of Pseudomonas spp.in the spoilage of raw meat is still lacking a systematic summary, especially the regulation and expression of genes involved in the spoilage of meat has not been determined. Therefore, this paper firstly summarizes the characteristics, classification and interspecific differences of Pseudomonas spp., and then overviews its contamination sources. It also elaborates the spoilage mechanisms from the perspective of substrate utilization (including carbohydrate metabolism, amino acid metabolism, lipid metabolism, etc.), and especially involved in the genes related to meat spoilage. Among them, the different expression levels of genes regulating enzymes in the ED pathway, and AprX gene that regulating extracellular protease and genes encoding ABC transporter, and lipA that encoding lipase may be the cause of the spoilage of Pseudomonas spp. The information gathered here will reveal the mechanism of Pseudomonas spp. on the spoilage of meat and provide a theoretical basis for controlling meat spoilage.
Keyword: meat; spoilage; Pseudomonas spp.; mechanism;
肉的腐败变质是指肉类在外界因素作用下,特别是微生物的污染,使其营养成分和感官性状发生变化,并可能产生对人体有害物质的过程[1]。全球消费者每年因腐败而拒绝购买的肉类总量为数百万吨[2]。因此,了解肉中腐败微生物的来源、种类及影响其生长繁殖的外界环境因素,并准确有效控制腐败微生物的生长,是肉类产业界和学术界共同的目标之一[3]。
假单胞菌(Pseudomonas spp.)是肉类腐败过程中最主要腐败菌之一。多种假单胞菌作为肉中的优势腐败菌被检出,如荧光假单胞菌(P. fluorescens)、莓实假单胞菌(P. fragi)、隆德假单胞菌(P. lundensis)、恶臭假单胞菌(P. putida)等。由于其耐冷特性,冷藏条件下易导致生鲜肉类的腐败变质;假单胞菌可以产生多种酶,包括蛋白酶和脂肪酶等,这些酶多是导致肉类变质的原因。目前很多研究探讨了假单胞菌的致腐机制,并取得一定研究进展,因此对假单胞菌对生鲜肉的致腐机制进行系统总结,对于控制肉的腐败进程十分必要。
因此,本文概述了假单胞菌的特性及其污染来源,并从底物利用(糖代谢、氨基酸代谢、脂代谢等)角度分析了假单胞菌基因水平上的致腐原因,以期为揭示肉品中假单胞菌的腐败机制并为控制腐败提供理论基础。

1、肉的腐败
肉的腐败是由微生物和内源酶的共同作用引起的[4],其主要原因是微生物随着贮藏时间的延长在肉的表面生长繁殖甚至利用较深层的营养物质产生代谢产物所导致。腐败微生物利用的营养物质主要分为三大类:碳水化合物(如糖酵解途径中的作用底物如葡萄糖原、葡萄糖等及代谢产物葡萄糖酸盐、葡萄糖-6-磷酸、丙酮酸盐、乳酸等),蛋白质及氨基酸(精氨酸、赖氨酸和鸟氨酸等氨基酸)、脂肪及脂肪酸等[5,7]。肉中的腐败微生物将碳水化合物如葡萄糖等物质分解为各种有机酸,同时产生醇和CO2等气体;产生的蛋白酶类使肉中的蛋白质被水解生成胺、氨、硫化氢、酚、粪臭素、硫化醇等[6];产生的脂肪酶类使脂肪被分解生成脂肪酸、甘油、醛、酮等化合物。也有研究显示肉品的腐败并不是全部的微生物菌群的作用,而是在初始菌群中占主导地位的少部分特定的微生物菌群(一般称为优势腐败菌)所导致。优势腐败菌群的种类、数量影响着肉的腐败进程[1,9]。
2、假单胞菌属
2.1、 假单胞菌属特性
肉中的腐败菌种类繁多,来源广泛。主要包括假单胞菌属、希瓦氏菌属、不动杆菌属等革兰氏阴性菌及乳酸杆菌属、明串珠菌属、环丝菌属等革兰氏阳性菌等[7]。其中假单胞菌属是肉类腐败过程中起主要作用的优势腐败菌之一,该菌为直或稍弯的革兰氏阴性杆菌,是严格好氧菌,但一些假单胞菌也可以在硝酸盐、富马酸盐或其他电子受体存在的缺氧条件下生长[8]。其生长营养条件较单一,大多数分泌精氨酸双水解酶,能利用不同的有机化合物(如乳酸盐、琥珀酸盐和葡萄糖等)作为碳源和氮源来提供能量,产生腐败代谢产物二甲硫醚,不产H2S[7]。最佳生长温度为25℃-35℃,其中大多数菌种为嗜冷菌,假单胞菌的冷适应能力与细胞膜内的高水平不饱和脂质和耐受冷诱导应激的多种机制有关[10]。
2.2、 假单胞菌种间差异品质
假单胞菌在不同环境如空气、土壤、水分、沉积物、动植物等甚至极端环境条件下均有检出,且菌种数量繁多。自1894年被发现以来,其分类方法逐步由宏观及微观形态学发展到将DNA碱基组成和DNA-DNA杂交技术应用于假单胞菌分类[11]。后来,根据RNA-DNA的测量结果,将假单胞菌分为5个rRNA亚群[12]。其中假单胞菌rRNA组I 类(P. sensu stricto)与肉类腐败密切相关,由铜绿假单胞菌(P. aeruginosa)、所有荧光假单胞菌和部分非荧光假单胞菌组成,如施氏假单胞菌(P. Stutzeri)、产碱假单胞菌(P. Alcaligenes)、类产碱假单胞菌(P. Pseudoalcaligenes)、门多萨假单胞菌(P. mendocina)等[13]。很多学者研究表明与肉类的腐败密切相关的莓实假单胞菌[14]、荧光假单胞菌[15],隆德假单胞菌[16]及恶臭假单胞菌[17]等多个菌种污染肉及肉制品后可引起肉表面腐败,并产生粘液和不良气味等现象。
莓实假单胞菌是有氧贮藏条件下冷却肉的优势腐败菌,在腐败肉中的检出率介于56.7%和79.0%之间[1]。在生成酯,特别是短链的乙基酯的过程中会产生果味异味,产生的挥发性化合物包括酮类、醇类、1-十一烯类和硫类化合物等[14]。Ercolini[14]等发现不同来源不同分型的莓实假单胞菌株在肉中都具有一定的腐败能力,但其蛋白水解活性及脂解活性有着较大差异,在冬季分离的菌株比夏季分离的菌株蛋白水解力更强。在气调包装的环境条件下,莓实假单胞菌无法分解肌酸和肌酐,因此无法成为优势腐败菌群,而荧光假单胞菌多被检出[9]。其次是隆德假单胞菌,在腐败肉中的检出率为40%[3],能产生大量的酮类化合物以及醇类、丁酸、1-十一烯和环己烷等。假单胞菌菌株可与其他腐败微生物群竞争性利用铁元素,如可以从乳铁蛋白中利用铁,因此莓实假单胞菌、隆德假单胞菌、荧光假单胞菌常在超高温瞬时灭菌的牛乳中检出[18]。恶臭假单胞菌除了作为低温有氧贮藏末期腐败肉的优势菌群,还在环境的治理中,起着降解与吸附污染物的重要作用[19]。
3、假单胞菌的污染来源
3.1、 屠宰过程
生鲜肉水分含量较高,营养丰富,在工厂生产加工过程中易受到多种微生物的污染。生鲜肉的腐败变质主要是由初始菌落占主导地位的微生物菌群决定的,控制其初始菌落的种类及数量可以有效减缓肉的腐败进程。许多学者研究发现假单胞菌属是猪肉、牛肉、羊肉、鸡肉等初始菌群中的优势腐败菌群之一[19,20,21,22]。
Korsak[23]研究发现清真屠宰技术与传统屠宰技术的差异不会影响消化道或呼吸道的特定微生物的生长模式,但其不同的卫生习惯会影响胴体表面微生物的污染情况,其中假单胞菌的数量在两种处理方式中未有显着性差异。堵舒桐[24]等研究发现在肉牛屠宰工序中,假单胞菌主要来源于操作台和分割工具。李虹敏[25]研究发现肉鸡加工厂的加工环境是肉鸡胴体细菌污染的潜在主要来源,黄羽肉鸡在屠宰加工的不同工序中微生物污染来源各不同,如空气、水质、操作人员、加工器具、内脏等,假单胞菌主要来源于预冷阶段。汤飞等[19]调查研究发现在操作工具和案板表面均检测出假单胞菌,张友华等[21]检测了冷鲜猪肉供应链的微生物污染情况,表明待宰圈、分割刀具、案板和工人的手是猪肉屠宰分割过程中最主要的微生物污染源,其中假单胞菌属主要来源于分割刀具、案板和工人的手。Drosinos 等[22]发现在羊肉生产加工阶段,去骨台、混合设备、传送带、托盘和箱体是最易污染荧光假单胞菌的地方。尹罗罗等[26]研究发现羊肉随着加工工序的进行,假单胞菌的数量逐渐上升,且夏季的细菌污染程度高于冬季。
综上,虽然肉的种类、屠宰环境、操作过程及宰前管理各有不同,但假单胞菌在各个环节均有不同程度的检出,其中屠宰环境与操作过程是假单胞菌的主要来源之一。故屠宰分割过程中应做好器具、工人的手等环境样品的定时清洗和消毒工作,规范操作过程、减少人员走动可以减少假单胞菌的污染。
3.2 、不同贮藏环境
假单胞菌的生长条件较简单,是严格好氧菌,常在低温有氧贮藏的肉中检出。顾春涛等[27]研究发现牛肉在0 ℃冷藏条件下,假单胞菌是致腐性较强的优势腐败菌,且在腐败后期占据主导地位。真空包装条件下,其他菌相的生长活动及缺氧环境会抑制假单胞菌的生长。但是有部分学者发现真空包装条件下假单胞菌在贮藏前期及中期腐败能力较强,在后期受到抑制。Chen等[28]将真空包装与冰温贮藏相结合探究了牛肉的腐败变化,发现假单胞菌数量在贮藏期间呈现先上升后下降的趋势。Blixt等[29]用电子鼻定量评定真空包装牛肉的变质程度,在少量初始假单胞菌存在的情况下,随着牛肉腐败程度的增加,假单胞菌的生长逐渐受到抑制。气调包装的气体成分比例不同,也会影响假单胞菌的生长状况。如杨啸吟等[30]等研究发现假单胞菌是高氧气调包装(80%O2/20%CO2)冷却牛肉的优势菌种,数量随贮藏时间的延长而显着增长,在一氧化碳气调包装(0.4%CO/30%CO2/69.6%N2)下假单胞菌的生长受到抑制但仍有检出。Fei等[31]将气调包装(75%O2/25%CO2和75%N2/25%CO2)与冰温贮藏相结合检测了假单胞菌在羊肉中的生长,发现假单胞菌数量随着贮藏时间的延长呈上升趋势。
不同贮藏与包装条件下在生鲜肉中的假单胞菌生长状况不同,不同菌种甚至菌株之间都有明显差异,此现象可能与不同假单胞菌间致腐基因的调控及表达机制有关。
4、 假单胞菌的腐败作用
4.1、 底物利用
在肉中,假单胞菌利用营养物质如糖类(葡萄糖、D-葡萄糖等)和氨基酸类,产生挥发性和非挥发性代谢物(酯类、酮类、醇类、醛类有机酸、硫化合物和胺等),引起肉的绿变、发粘及变味的现象。其中,葡萄糖尤其是D-葡萄糖是假单胞菌属优先利用的碳源及能量物质[3]。葡萄糖酸盐也常被研究者们用作肉类腐败标志性指示物质,生鲜肉中假单胞菌属多通过2-酮-3-脱氧-6-磷酸葡糖酸途径(Entner-Doudoroff,ED)代谢途径分解葡萄糖产生葡萄糖酸盐以供自身生长繁殖。
在葡萄糖酸盐消耗殆尽阶段时,假单胞菌将氨基酸作为新的能量来源[33]。大多数的假单胞菌能分泌蛋白酶,具有蛋白水解活性。并且当肉表面的营养物质消耗殆尽时,假单胞菌可以利用其蛋白水解活性渗透进肉的内部,使肉发生较深层次的腐败,加速肉腐败的进程。
此外,假单胞菌属分泌的脂肪酶可以在肉制品中催化脂肪酸甘油酯水解为甘油和脂肪酸,从而导致腐败[34]。
假单胞菌的致腐败能力往往与其代谢分解底物的能力及底物先后利用的顺序有一定关联,其代谢水平的差异除受到菌株的特异性、环境因素及群体感应等的影响外,还可能受到假单胞菌致腐基因的调控,因此,更深层次的探究其基因水平的表达差异可以更深一步揭示不同假单胞菌株在特定的环境下成为优势腐败菌的原因。本文从底物利用(糖代谢、氨基酸代谢、脂代谢等)角度概括分析了假单胞菌基因水平上的致腐原因,如图1所示。
图1 假单胞菌在生鲜肉中腐败机制示意图
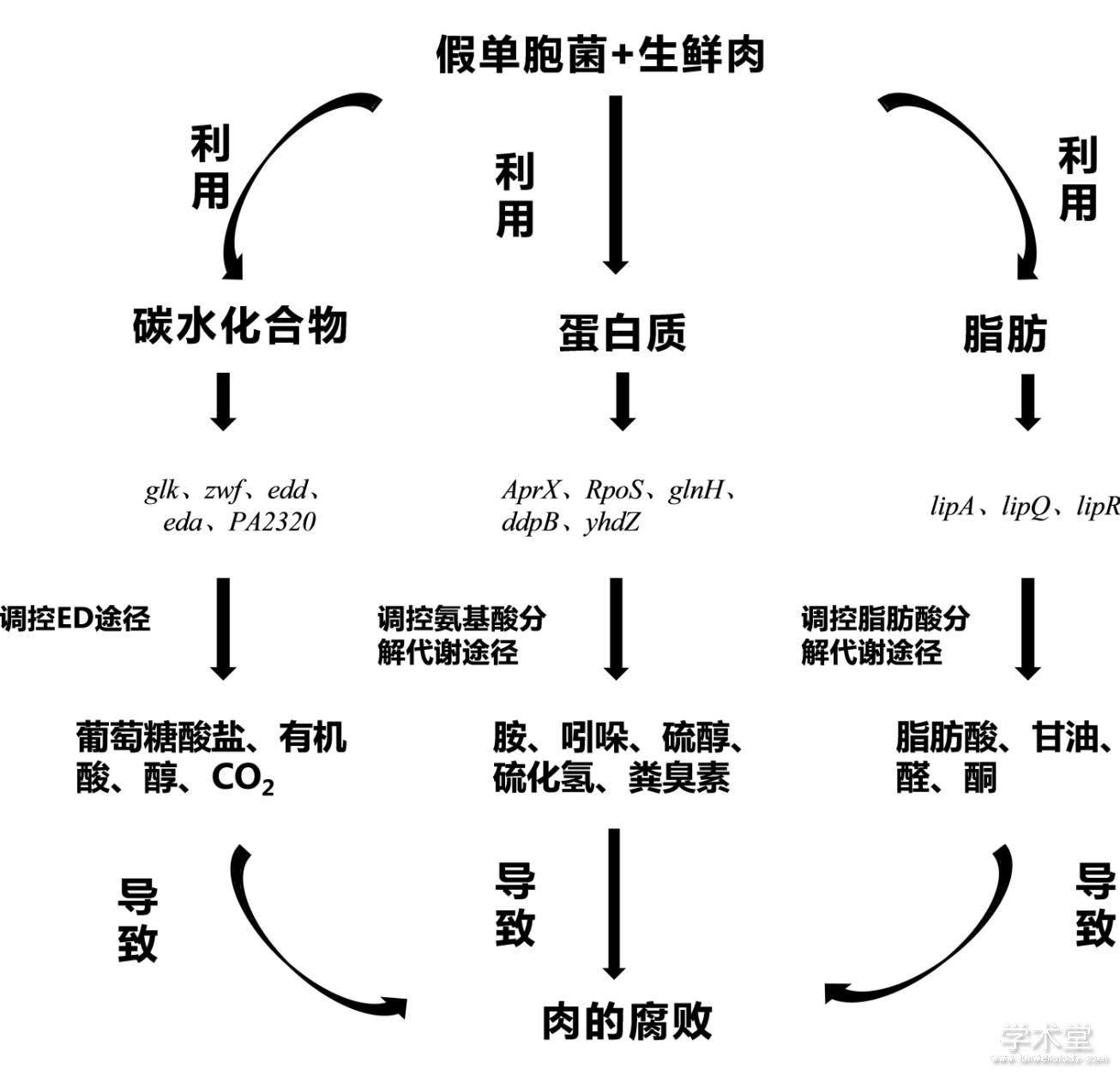
Figure 1. Schematic diagram of the spoilage mechanism of Pseudomonas spp. in raw meat
4.2、假单胞菌与腐败相关的基因研究
4.2.1、与碳水化合物代谢相关
一般来说,原核生物中ATP的合成主要通过糖酵解(Embden-Meyerhof-Parnas,EMP)和氧化磷酸化进行,许多学者研究表明假单胞菌属多通过ED代谢途径来代谢葡萄糖,产生葡萄糖酸盐。Pablo I[32]等研究发现恶臭假单胞菌是由于缺乏糖酵解(EMP)酶6-磷酸果糖-1-激酶(Pfk),从而几乎完全使用ED途径来降解己糖。Castill等[35]发现恶臭假单胞菌的葡萄糖分解代谢也可通过3条次级途径同时进行,分别是葡萄糖激酶途径、2-酮葡萄糖酸循环和葡萄糖酸磷酸化。通过这三种途径使葡萄糖转化成6-磷酸葡萄糖酸盐,而后进入中心代谢循环。其中编码葡萄糖激酶的glk基因位于edd基因的操纵子中,编码葡萄糖-6-磷酸脱氢酶的zwf1基因与eda基因形成操纵子。Sudarsan等[36]发现恶臭假单胞菌KT2440通过ED、EMP、戊糖磷酸途径和三羧酸(Tricarboxylic-Acid-Cycle,TCA)循环的葡萄糖代谢量远远高于任何一条次级途径,GltR基因控制葡萄糖激酶途径,PtxS和LacI通过编码酮葡萄糖酸盐和调节葡萄糖酸盐途径来控制葡萄糖的利用,ptxS基因和lacI基因的上调会诱导葡萄糖摄取基因(oprB1、oprB2、PP1015至PP1018)的表达以及参与葡萄糖酸盐和酮葡糖酸盐摄取的基因(gntP、kguT和kguK)的转化,并使负责形成中心ED途径中间体6-磷酸葡萄糖酸盐的基因(zwf1、zwf2和pgl)表达上调。从而使假单胞菌对葡萄糖的摄入增加。而Abdelali[37]等认为铜绿假单胞菌仅通过ED代谢途径来代谢葡萄糖,其代谢过程受到四种特定的调节系统(PtxS、HexR、GtrS / GltR与GntR)来调节葡萄糖的代谢与转运,其中PtxS调控因子控制其自身的表达以及操纵子gadCBA和kguEKTD的表达,后者编码参与碳水化合物运输和代谢的蛋白质,HexR抑制与糖代谢合成与转运相关的glk、zwf、edd和eda基因的表达[38]。在铜绿假单胞菌葡萄糖代谢相关基因的遗传图谱中识别了ORF PA2320基因片段,其对应蛋白为转录调控因子家族的GntR[39],GntR调控葡萄糖酸盐操纵子gntRKPZ、葡萄糖激酶(GntK)、葡萄糖酸盐渗透酶(GntP)和葡萄糖酸盐6-磷酸脱氢酶(GntZ)的表达。
Mohareb等[33]发现恶臭假单胞菌与腐败密切相关的基因表达受外界葡萄糖浓度与温度的调控。在不同温度下PP4444、PP4030、PP3088基因的差异表达是受葡萄糖浓度影响最大。其中PP4444基因对DNA转座酶的表达至关重要,PP4030基因参与顺式不饱和脂肪酸的降解,PP3088基因其负责调控的蛋白质尚未可知,对葡萄糖的利用机制值得进一步的研究。
Wang等[40]通过莓实假单胞菌在不同包装方式下的转录组分析,发现气调包装下电子传递链(nuoAB)基因的表达受到了抑制,糖酵解通路中编码二磷酸果糖醛酶II类的fbaA基因片段过量表达,从而可能导致丙酮酸的积累,抑制了假单胞菌对葡萄糖的摄入。
在肉品腐败初期,假单胞菌利用肉中的葡萄糖通过ED途径、EMP途径、戊糖磷酸途径、TCA循环及外周通路途径等来进行糖代谢。这其中涉及到的与糖代谢相关的基因较多,目前还不能确定调控其糖代谢的限速基因。同时,因与肉腐败的假单胞菌种类较多,每种假单胞菌糖代谢的速率、途径不尽相同。多种假单胞菌在糖代谢途径是否存在导致肉腐败的共同基因,有待于进一步研究。假单胞菌的基因片段通过编码或调控这些途径来参与糖代谢的过程,当葡萄糖消耗至不足以为假单胞菌的生长代谢提供碳源及能量时,便开始利用氨基酸作为新的能量来源。
4.2.2、与氨基酸代谢相关
许多研究表明有氧贮藏条件下,在生鲜肉的腐败初期假单胞菌与多个菌属均为优势腐败菌。但是随着贮藏时间的延长,假单胞菌的腐败优势更加明显[41],这可能是与假单胞菌有较强的蛋白水解活性,能够在表面营养物质利用完后深入到肉的内部,继续利用新的营养物质来完成自身的生长代谢有关。
大多数假单胞菌能分泌蛋白酶(如精氨酸酶、赖氨酸脱羧酶和鸟氨酸脱羧酶等)。假单胞菌在冷藏过程中分泌的蛋白酶是具有热稳定性的胞外蛋白酶,其主要代表是AprX家族,这是一个由AprX基因编码的碱性锌金属蛋白酶家族[42],在多数荧光假单胞菌株中,AprX家族蛋白酶已被确定为参与食品腐败的唯一蛋白酶。Wang等[40]发现在气调包装下,莓实假单胞菌NMC25涉及氨基酸生物合成的一系列基因如丝氨酸、缬氨酸、亮氨酸、异亮氨酸、半胱氨酸、天冬氨酸、丙氨酸、鸟氨酸等表达量较高,同时也影响了编码ABC转运蛋白的基因(如glnH、ddpB、yhdZ等)的表达,从而抑制肉类腐败。氨基酸的转录是通过RpoS基因表达的,RpoS基因与核心RNA聚合酶相关[43]。还有学者[44]认为CbrAB双组分系统是碳源或氮源的几种氨基酸(如组氨酸、脯氨酸或精氨酸)同化所必需的调控系统。
氨基酸的代谢较为复杂,并与葡萄糖和脂肪酸的代谢紧密相关。总体而言,鉴于AprX家族蛋白酶与腐败的确定性[42],可以从调控AprX蛋白酶的基因入手,探索假单胞菌与氨基酸代谢相关的腐败基因。而调控氨基酸合成的基因和编码ABC转运蛋白的基因的过量表达能够抑制莓实假单胞菌的腐败进程已经得到证实[40],因此相关基因在其它菌种,如荧光假单胞菌、恶臭假单胞菌中对肉腐败的调控作用可以作为未来的研究方向。
4.2.3 、与脂肪酸代谢相关
假单胞菌属在肉制品和蛋奶制品中分泌胞外脂肪酶,分解利用底物中的脂肪产生脂肪酸、甘油、醛、酮等物质,造成食物腐败并产生令人不悦的气味。脂肪酶是一类重要的酯解酶,属于α/β型水解酶超家族[45]。大多数假单胞菌属能分泌脂肪酶,但脂肪酶基因表达机制和途径至今仍未有所定论[46]。
在假单胞菌属中,通常由lipA基因编码脂肪酶的形成,Conor等[47]发现荧光假单胞菌B52的脂肪酶(lipA)基因和碱性金属蛋白酶(aprX)基因位于同一个操控子的两端,其表达受到温度以及渗透压双组分EnvZ/OmpR调控体系同源物的调控,Zha等[48]发现Gac/Rsm信号转导系统对假单胞菌的lipA基因的表达有调控作用,其中Gac-RsmE系统主要负责直接激活lipA翻译,而Gac-RsmA系统则间接激活了lipA的翻译。Krzeslak等[49]发现脂肪酶在假单胞菌中的表达受LipQR双组分体系的调控,其中lipQ和lipR两个基因的编码蛋白属于NtrBC家族的调控因子,通过与特定的上游激活因子序列(upstream activator sequence, UAS)结合来调控脂肪酶基因表达,双组分调节系统控制脂肪酶操纵子的转录。
基于酰基高丝氨酸内酯(acylhomoserine lactones, acyl-HSL)的群体感应(Quorum Sensing, QS)系统对脂肪酶基因的表达的影响显着[45,50]。假单胞菌通过I型分泌途径(由ATP-结合蛋白、内膜融合蛋白与外膜蛋白等组成)和II型分泌途径(由亚基、分泌子、酶、蛋白等构成)分泌胞外脂肪酶。Rosenau等[51]研究发现脂肪酶的分泌途径与细胞质前脂肪酶的26个氨基酸的N-末端信号序列有关,缺乏信号序列的荧光假单胞菌通过I型分泌途径分泌脂肪酶,拥有信号序列的铜绿假单胞菌脂肪酶是通过II型途径分泌的。而查代明等[46]认为假单胞菌胞外脂肪酶的分泌途径与是否拥有基于acyl-HSL的QS系统有关。
综上所述,编码脂肪酶lipA基因表达受到环境因素及多种双组分系统的调控,而且脂肪酶的分泌途径与表达机制也较为复杂,不同形成途径的脂肪酶其功能表达有无明显差异还需要进一步的研究。
4.2.4 、与生物膜黏附相关
肉中的腐败菌容易黏附在食品接触表面或食品表面以形成生物膜,这可能导致食品的连续或交叉污染,缩短保质期并影响产品的安全性。假单胞菌的致腐能力与生物膜的形成及黏附性有相关联系[52]。Amador等[44]发现恶臭假单胞菌CbrAB双组分系统中中CbrB的缺乏会影响生物膜的形成。王雨舟[53]确定了fleQ、fliA和rpoN是恶臭假单胞菌中合成鞭毛所必需的基因。细菌的运动、黏附、代谢转换、胞外多糖的产生、DNA和蛋白质的分泌等多种功能对生物膜的形成具有重要影响,编码这些细胞功能的基因在生物膜发育过程中受到很多因素的调控。Tribelli等[54]研究表明,在低氧条件下,anr基因调节细胞的聚集和运动来影响生物膜形成的第一阶段。荧光假单胞菌中的adnA基因是铜绿假单胞菌fleQ基因的同源基因,调节着细菌的运动和由鞭毛介导的在各种物品表面的黏附[55]。但是生鲜肉中主要假单胞菌如莓实假单胞菌、荧光假单胞菌及恶臭假单胞菌,其生物膜形成相关的基因与生鲜肉腐败之间的关系还远未探明。
5 、结语
肉的腐败往往不是肉中全部的微生物造成的,也不是单一微生物的作用,是特定优势腐败菌群的共同作用的结果。假单胞菌作为生鲜肉贮藏过程中的优势腐败菌,其致腐的机制十分复杂,它不仅受到多种环境因素影响,而且还受到在糖代谢、氨基酸代谢、脂代谢多个信号系统的调节。
目前对腐败菌研究逐渐深入到基因水平。假单胞菌属中的不同菌种在不同的生长环境下腐败能力差异显着,这可能与假单胞菌种间的腐败基因的表达差异有关。目前国内外对比探究不同假单胞菌种间、菌株间致腐能力差异的相关研究还较少,因此,未来的研究可以从腐败基因表达水平的差异来探究假单胞菌的致腐机制。利用新型基因技术来探究假单胞菌的与致腐相关的基因有助于揭示肉的腐败现象的深层原因,从而找到更好的抑制措施,来减缓肉的腐败,延长肉品货架期。
参考文献
[1] NYCHAS G J E, SKANDAMIS P N, TASSOU C C, et al. Meat spoilage during distribution[J].Meat Science, 2008, 78(1-2): 77-89. DOI:10.1016/j.meatsci.2007.06.020.
[2] Food and Agriculture Organization of the United States. Save Food:global initiative on food loss and waste reduction[EB/OL]. (2016-12-04) [2017-01-10]. http://211.85.197.33:80/rwt/02/http/P75YPLUGMFYT655TMH/save-food/resources/keyfindings/infographics/meat/en/.
[3]张一敏, 朱立贤, 张万刚,等.生鲜牛肉中的腐败微生物概述[J]. 食品科学, 2018, 39(13): 296-303. DOI:10.7506/spkx1002-6630-201813043.
[4] 刘朏. 不同贮藏温度对冷鲜鸡微生物和肉品品质的影响研究[D]. 杨凌: 西北农林科技大学, 2015: 1-2.
[5] 修艳辉. 鱼源腐败希瓦氏菌生长动力学及碳源代谢研究[D]. 上海:上海海洋大学, 2017:3-5.
[6]李雪, 蔡丹, 沈月, 等.微生物来源蛋白酶的研究进展[J]. 食品科技, 2019, 44(01): 37-41. DOI:10.13684/j.cnki.spkj.2019.01.006.
[7]尹德凤, 张莉, 张大文, 等.生鲜肉类产品中腐败细菌研究[J]. 农产品质量与安全, 2018, No.093(03): 23-31. DOI:CNKI:SUN:NYZL.0.2018-03-005.
[8] ANDREANI N A, FASOLATO L. Chapter 2 – pseudomonas, and related genera[J]. Microbiological Quality of Food, 2017:25-59. http://211.85.197.33:80/rwt/02/https/MSYXTLUQPJUB/10.1016/B978-0-08-100502-6.00005-4.
[9] DOULGERAKI A I, ERCOLINI D, VILLANI F, et al. Spoilage microbiota associated to the storage of raw meat in different conditions[J]. International Journal of Food Microbiology, 2012, 157(2). DOI:10.1016/j.ijfoodmicro.2012.05.020.
[10] MORENO R, ROJO F. Features of pseudomonads growing at low temperatures: another facet of their versatility[J]. Environmental Microbiology Reports, 2014, 6(5):417-426. DOI:10.1111/1758-2229.12150.
[11] PEIX A, MARTHA-HELENA RAM?REZ-BAHENA, ENCARNA VEL?ZQUEZ. Historical evolution and current status of the taxonomy of genus pseudomonas[J]. Infection Genetics & Evolution, 2009, 9(6):1132-1147. DOI:10.1016/j.meegid.2009.08.001.
[12] FRANZETTI L, SCARPELLINI M. Characterisation of pseudomonas spp. isolated from foods[J]. Annals of Microbiology, 2007, 57(1):39-47. DOI:10.1007/BF03175048.
[13] ALVAROPEIX, MARTHA-HELENARAM?REZ-BAHENA, ENCARNAVEL?ZQUEZ. The current status on the taxonomy of pseudomonas revisited: An update[J]. Infection, Genetics and Evolution, 2018, 57: 106-116. http://211.85.197.33:80/rwt/02/https/MSYXTLUQPJUB/10.1016/j.meegid.2017.10.026
[14] ERCOLINI D, CASABURI A, NASI A , et al. Different molecular types of pseudomonas fragi have the same overall behaviour as meat spoilers[J]. International Journal of Food Microbiology, 2010, 142(1-2):120-131. DOI:10.1016/j.ijfoodmicro.2010.06.012.
[15] MELLOR G E, BENTLEY J A, DYKES G A. Evidence for a role of biosurfactants produced by pseudomonas fluorescens in the spoilage of fresh aerobically stored chicken meat[J]. Food Microbiology, 2011, 28(5):1101-1104 DOI:10.1016/j.fm.2011.02.00.
[16] LIU Yongji, XIE Jing, ZHAO Lijun, et al. Biofilm formation characteristics of, pseudomonas lundensis, isolated from meat[J]. Journal of Food Science, 2015, 80(12):M2904-M2910. DOI:10.1111/1750-3841.13142.
[17]陈雨欣,苏粉良,鞠慧萍,等.进口肉类中恶臭假单胞菌的分离与鉴定[J].食品安全质量检测学报,2017,8(11):4436-4438.DOI:CNKI:SUN:SPAJ.0.2017-11-066 .
[18] MORALES P, FERN?NDEZ-GARC?A, ESTRELLA, NU?EZ M. Volatile compounds produced in cheese by, pseudomonas, strains of dairy origin belonging to six different species[J]. Journal of Agricultural and Food Chemistry, 2005, 53(17):6835-6843. DOI:10.1021/jf050717b.
[19] 汪晶, 柯凤乔. 一株降解雌二醇的恶臭假单胞菌的分离鉴定及降解活性[J]. 环境化学, 2015(4): 814-816.
[20] 汤飞. 冷鲜鸡肉生产过程中微生物污染分析及微冻保鲜技术研究[D]. 合肥: 合肥工业大学, 2017:10-21.
[21] 张友华. 冷鲜猪肉供应链微生物调查分析[D]. 成都: 西华大学, 2015:33-37.
[22] DROSINOS E H. Microbial and physicochemical attributes of minced lamb: sources of contamination with pseudomonads[J]. Food Microbiology, 1995, 12(3):189-19. DOI:10.1016/S0740-0020(95)80097-2.
[23] KORSAK N, TAMINIAU B, HUPPERTS C, et al. Assessment of bacterial superficial contamination in classical or ritually slaughtered cattle using metagenetics and microbiological analysis[J]. International Journal of Food Microbiology, 2017, 24(17): 79-86. DOI:10.1016/j.ijfoodmicro.2016.10.013.
[24] 堵舒桐. 新疆肉牛屠宰加工及销售过程中主要微生物生长情况的调查研究[D]. 乌鲁木齐: 新疆农业大学, 2014: 8-20.
[25] 李虹敏. 生鲜鸡肉产品微生物污染分析及其保鲜技术研究[D]. 南京: 南京农业大学, 2009:15-23.
[26]尹罗罗.塔城地区不同季节腐败微生物来源分析[J]. 安徽农业科学, 2015, 43(33): 112-114. DOI:10.3969/j.issn.0517-6611.2015.33.0395.
[27] 顾春涛,毕伟伟,朱军莉.冷鲜牛肉贮藏中菌群结构及优势菌致腐性的分析[J/OL].食品科学:1-10[2019-04-24].http://211.85.197.33:80/rwt/02/http/NNYHGLUDN3WXTLUPMW4A/kcms/detail/11.2206.TS.20181214.1404.064.html.
[28] CHEN Xue, ZHANG Yimin, YANG Xxiaoyin, et,al. Shelf-life and microbial community dynamics of super-chilled beef imported from Australia to China.[J]. Food research international (Ottawa, Ont.), 2019, 120. DOI:10.1016/j.foodres.2018.11.039.
[29] BLIXT Y, BORCH E. Using an electronic nose for determining the spoilage of vacuum-packaged beef[J]. International Journal of Food Microbiology, 1999, 46(2):123-134. DOI:10.1016/s0168-1605(98)00192-5.
[30] 杨啸吟. 高氧与一氧化碳气调包装对冷却牛排肉色稳定性影响的机理探究[D]. 泰安: 山东农业大学, 2018:56-58.
[31] ZHAO Fei, JING Hongpeng, WU Xinling, et al. Effect of modified atmosphere packaging in combination with ice-temperature on quality of mutton during storage[J]. Food Science, 2015, 45(9): 3772-3778. DOI:10.1021/ma300394u.
[32] NIKEL P I, MAX CHAVARR?A, FUHRER T, et al. Pseudomonas putida KT2440 strain metabolizes glucose through a cycle formed by enzymes of the entner-doudoroff, embden-meyerhof-parnas, and pentose phosphate pathways[J]. Journal of Biological Chemistry, 2015, 290. DOI:10.1074/jbc.M115.687749.
[33] MOHAREB F, IRIONDO M, DOULGERAKI A I, et al. Identification of meat spoilage gene biomarkers in pseudomonas putida using gene profiling[J]. Food Control, 2015, 57: 152-160. DOI:10.1016/j.foodcont.2015.04.007.
[34] 查代明, 闫云君. 细菌脂肪酶基因表达调控的研究进展[J]. 微生物学报, 2015, 55(11): 1378-1384. DOI:10.13343/j.cnki.wsxb.20150117.
[35] DEL CASTILLO T, RAMOS J L, RODRIGUEZ-HERVA J J, et al. Convergent peripheral pathways catalyze initial glucose catabolism in pseudomonas putida: genomic and flux analysis[J]. Journal of Bacteriology, 2007, 189(14): 5142-5152. DOI:10.1128/JB.00203-07.
[36] SUDARSAN S, DETHLEFSEN S, BLANK L M, et al. The functional structure of central carbon metabolism in pseudomonas putida KT2440[J]. Applied and Environmental Microbiology, 2014, 80(17): 5292-5303. DOI:10.1128/AEM.0164.
[37] DADDAOUA A, CORRAL-LUGO A, RAMOS J, et al. Identification of GntR as regulator of the glucose metabolism in Pseudomonas aeruginosa[J]. Environmental Microbiology, 2017, 19(9): 3721–3733. DOI:10.1111/1462-2920.13871.
[38] DADDAOUA A, KRELL T, RAMOS J L. Regulation of glucose metabolism in pseudomonas: the phosphorylative branch and entner-doudoroff enzymes are regulated by a repressor containing a sugar isomerase domain[J]. Journal of Biological Chemistry, 2009, 284(32): 21360-21368. DOI:10.1074/jbc.M109.014555.
[39] JAIN, DEEPTI. Allosteric control of transcription in GntR family of transcription regulators: A structural overview[J]. IUBMB Life, 2015, 67(7): 556-563. DOI:10.1002/iub.1401.
[40] WANG Guangyu, MA Fang, CHEN Xiaojing, et al. Transcriptome analysis of the global response of pseudomonas fragi NMC25 to modified atmosphere packaging stress[J]. Frontiers in Microbiology, 2018, 9(1277). DOI:10.3389/fmicb.2018.01277
[41] 赵晓策, 胡倩倩, 罗瑞明, 张赫宇. 冷鲜滩羊肉贮藏中菌体蛋白质与微生物群落演替的关联性研究[J/OL].食品科学:1-10[2019-03-07].http://211.85.197.33:80/rwt/02/http/NNYHGLUDN3WXTLUPMW4A/kcms/detail/11.2206.TS.20181213.1429.050.htm.
[42] ANDREANI N A, CARRARO L, FASOLATO L, et al. Characterisation of the thermostable protease AprX in strains of pseudomonas fluorescens and impact on the shelf-life of dairy products: preliminary results[J]. Italian Journal of Food Safety, 2016, 5(4): 6175. DOI:10.4081/ijfs.2016.6175.
[43] REPOILA F, GOTTESMAN S. Signal transduction cascade for regulation of RpoS: temperature regulation of DsrA[J]. Journal of Bacteriology, 2001, 183(13): 4012-4023. DOI:10.1128/JB.183.13.4012-4023.2001.
[44] AMADOR C I, IN?S CANOSA, GOVANTES F, et al. Lack of CbrB in pseudomonas putida affects not only amino acids metabolism but also different stress responses and biofilm development[J]. Environmental Microbiology, 2010, 12(6): 1748-1761. DOI:10.1111/j.1462-2920.2010.02254.x.
[45] ROSENAU F, JAEGER K E. Bacterial lipases from pseudomonas: regulation of gene expression and mechanisms of secretion[J]. Biochimie, 2000, 82(11): 1023-1032. DOI:10.1016/S0300-9084(00)01182-2.
[46] 查代明, 张炳火, 李汉全,等. 假单胞菌属脂肪酶的分子生物学研究进展[J]. 中国生物工程杂志, 2015, 35(9): 114-121. DOI:10.13523/j.cb.20150916.
[47] MCCARTHY C N, WOODS R G, BEACHAM I R. Regulation of the aprX–lipA operon of pseudomonas fluorescens B52: differential regulation of the proximal and distal genes, encoding protease and lipase, by ompR–envZ[J]. FEMS Microbiology Letters, 2004, 241(2): 243-8. DOI:10.1016/j.femsle.2004.10.027.
[48] ZHA D, XU L, ZHANG H, et al. The two-component GacS-GacA system activates lipA translation by RsmE but not RsmA in pseudomonas protegens Pf-5[J]. Applied and Environmental Microbiology, 2014, 80(21): 6627-6637. DOI:10.1128/aem.02184-14.
[49] KRZESLAK J, GERRITSE G, MERKERK R V, et al. Lipase expression in pseudomonas alcaligenes is under the control of a two-component regulatory system[J]. Applied and Environmental Microbiology, 2008, 74(5): 1402-1411. DOI:10.1128/AEM.01632-07.
[50] REIMMANN C, BEYELER M, LATIFI A, et al. The global activator GacA of pseudomonas aeruginosa PAO positively controls the production of the autoinducer N-butyryl-homoserine lactone and the formation of the virulence factors pyocyanin, cyanide, and lipase.[J]. Molecular Microbiology, 2010, 24(2): 309-319. DOI:10.1046/j.1365-2958.1997.3291701.x.
[51] ROSENAU F, JAEGER K E. Bacterial lipases from pseudomonas: regulation of gene expression and mechanisms of secretion[J]. Biochimie, 2000, 82(11): 1023-1032. DOI:10.1016/S0300-9084(00)01182-2.
[52] 邓旗. 对虾特定腐败菌生物被膜的形成特性及抗菌脂肽的控制作用[D]. 湛江: 广东海洋大学, 2013: 14-16.
[53] 王雨舟. 恶臭假单胞菌KT2442鞭毛关键调控基因对菌株生物学特性的影响[D]. 无锡: 江南大学,2018: 8-27. DOI:CNKI:CDMD:2.1018.063656.
[54] TRIBELLI P M, HAY A G, NANCY I L?PEZ. The global anaerobic regulator Anr, is involved in cell attachment and aggregation influencing the first stages of biofilm development in pseudomonas extremaustralis[J]. PLOS ONE, 2013, 8(10): e76685. DOI:10.1371/journal.pone.0076685.
[55] WILLIAMSON K S, RICHARDS L A, PEREZOSORIO A C, et al. Heterogeneity in pseudomonas aeruginosa biofilms includes expression of ribosome hibernation factors in the antibiotic-tolerant subpopulation and hypoxia-induced stress response in the metabolically active population.[J]. Journal of Bacteriology, 2012, 194(8): 2062-73. DOI:10.1128/JB.00022-12.





