摘 要: 环状RNA (circular RNA, circRNA) 是由真核生物中的前体mRNA反向剪接产生的环状内源性RNA分子, 分布广泛、结构稳定、序列高度保守, 具有细胞和组织特异性。circRNA以多种方式参与调控生物学过程以及疾病的发生发展。目前发现的功能模式包含竞争性内源性RNA作用、与蛋白质互作、翻译成蛋白质以及顺式调控亲本基因的转录等。随着circRNA的基本信息、功能分析和与人类疾病的关系等研究内容不断更新增加, 急需对海量的信息进行整合分析, 以及对相应细胞或组织来源的circRNA进行分类。本文从基本信息整合及分析、相关功能分析和与人类疾病的相关性等方面, 对目前常用的circRNA数据库进行综述, 以期为circRNA研究提供参考。
关键词: 环状RNA; 数据库; 信息整合; 功能预测; 人类疾病;
Abstract: Circular RNA (circRNA) is a circular endogenous RNA molecules generated by reverse splicing of precursor mRNA in eukaryotes. It is widely distributed with stable structure, highly conservative sequences, and cell-and tissue-specificity. CircRNA is involved in the occurrence and development of human diseases by regulating multiple biological processes, such as competitively interacting with proteins, translating into proteins, or cis-regulating the transcription of parental gene. For better understanding the roles of circRNA, it is urgent to integrate and analyze the massive information and classify the circRNA molecules from different sources. Here, we summarized the circRNA databases in terms of basic information integration, function prediction and the association with human disease, which would provide integrated information on circRNA databases for the relevant scientific research.
Keyword: circular RNA (circRNA) ; database; information integration; function prediction; human disease;
环状RNA (circular RNA, circRNA) 是由前体mRNA通过反向剪接产生, 在真核细胞中表达的内源性共价闭合环状RNA分子[1]。按照产生来源不同, circRNA可分为以下几类[2,3]:外显子circRNA (exonic circRNA) 、内含子circRNA (intronic circRNA, ciRNA) 、外显子-内含子杂合circRNA (exonic-intronic circRNA, EIciRNA) 、反义circRNA (antisense circRNA) 、基因内circRNAs (intragenic circRNA) 、基因间circRNAs (intergenic circRNA) 等。其中, 外显子来源的circRNA占的比例最大, 主要分布在细胞质中, 而ciRNA、EIciRNA主要分布在细胞核中[4,5]。circRNA以多种方式参与调控生物学过程并且与人类疾病相关, 包括通过竞争性内源性RNA (competitive endogenous RNA, ceRNA) 途径竞争性结合miRNA调控下游分子[6];与蛋白质互作介导转录、复合体形成等生物进程[7,8];参与蛋白质的翻译[9];通过内含子间竞争性互补配对等机制顺势调控亲本基因的转录[10]。基于大量发表的circRNA研究, 出现了众多的数据整合分析平台。但是, 由于不同数据库采用的资料收录条件、计算方法和侧重点等的不同, 如何根据研究目的去正确选择以及联合应用, 要求我们对目前的数据库进行一定的分类和对比描述。本文据常用circRNA 数据库, 从以下方面分别进行阐述: (1) circRNA基本信息整合及分析数据库; (2) circRNA相关功能分析数据库; (3) 与人类疾病相关circRNA数据库。通过对circRNA数据库收录内容、使用方法和 (或) 预测分析功能等几个方面的介绍, 帮助相关研究者快捷准确地查找和有效利用有价值的网络资源。
1 circRNA基本信息整合及分析数据库
1.1 circBase
circBase于2014年首发[11], 最新版本更新于2017年6月。目前收集6个物种包括人、小鼠、秀丽线虫、黑腹果蝇、矛尾鱼和腔棘鱼的circRNA信息。circBase提供了基因组位置、细胞系或组织来源和参考文献等信息, 并支持circRNA序列查询和相关信息下载。该平台可对拟选取研究的circRNA有具体的认识, 以及下载相关信息以供进一步的研究。另外值得一提的是, circBase虽在circRNA命名系统做出了一定的贡献, 但依旧没有做到精确的归类和定位, 有待进一步研究以建立统一规范化的circRNA命名方式。
1.2 CIRCpedia
circRNA的剪接形式存在细胞定位、组织类型等多方面的特异性特征[12], 表明circRNA的剪接形式很可能隐含着重要的调控功能, 值得深入研究。因此, 建立对circRNA可变反向剪接和可变剪接的分析和归类的数据库, 无疑能推动对circRNA生成加工的复杂性和潜在功能多样性的理解。2016年6月, 上海计算生物学研究所Yang研究组, 利用建立的高效circRNA分析流程CIRCexplorer2[13], 对多种组织和细胞系样品中的circRNA进行计算生物学预测分析。通过比较circRNA和对应线性RNA的表达, 绘制了circRNA可变反向剪接和可变剪接图谱, 揭示了circRNA的可变反向剪接受到其上下游内含子互补配对序列的竞争配对调控机制, 并进一步构建了CIRCpedia数据库[13]。对circRNA分子中的可变反向剪接和可变剪接进行归类。该数据库同时也包含细胞组织来源、GEO (gene expression omnibus) 编号、样品基本信息, 并且可直接观察circRNA及可变剪接对应基因组中的位置。用户还可据可变剪接类型和细胞类型等下载数据。截至到2016年12月的更新, 目前该数据库包含31个人类、26个小鼠、30个苍蝇和12个蠕虫样本的circRNA可变反向剪接和可变剪接信息, 为系统阐述circRNA的来源、加工及其在体内的潜在功能奠定了坚实的数据理论基础。
1.3 CircView
可视化是探索和分析基因组/蛋白质组数据的一个重要方面, 交互式可视化有助于研究人员理解研究对象潜在的生物学意义。鉴于circRNA基本信息数据库和检测工具数量的增多, Han课题组和He课题组共同开发了环状RNA可视化程序CircView[14], 能够准确和有效地识别和量化由不同技术从各种样本中鉴定的circRNA, 包括人、小鼠、斑马鱼、蝇以及蠕虫。通过导入circRNA数据, CircView能够根据选定的物种、计算方法以及基因名字和 (或) 位置等注释文件信息, 自动将circRNA信息映射到物种基因上, 并以直观的形式展现circRNA在基因上的位置, 也可分析不同样本或不同circRNA预测算法获得的数据样本特异性和预测算法分布。此外, CircView也能通过导入相关数据, 以直观形式显示circRNA上的miRNA识别位点 (miRNA recognition element, MRE) 和RNA结合蛋白 (RNA binding protein, RBP) 的位点数据。CircView界面操作简单, 同时还可直接获取多种常用数据库的链接, 对circRNA的分析研究十分有利。
1.4 AtCircDB
circRNA 同样广泛分布于植物中, 参与光合作用等重要过程的调控。收录多种植物circRNA资料的数据库虽然已经建立[15,16], 但是鉴于缺乏全面的描述植物组织特异性的circRNA数据库, 2017年, Zhao等分析了87个NCBI SRA (sequence read archive) 独立研究, 包括622个拟南芥RNA测序数据集, 开发了AtCircDB数据库[17], 可供识别、储存和检索拟南芥circRNA。通过分析反向剪接位点, 注释了84 685个circRNA, 30 648个组织特异性circRNA和3 486个miRNA-circRNA互作, 涵盖9种植物组织, 鉴定了313个叶绿体相关的circRNA以及82个植物线粒体相关的circRNA。用户通过查询circID、TAIR ID (The Arabidopsis Information Resource ID) 、基因组位点、miRNA、富集区域等, 获取相关circRNA的信息, 如染色体位置、母本链信息、反向剪接位点、潜在相互作用的miRNA、实验证据、检测分数 (detection score) 和组织特异性等。并且该数据库支持可视化, 为研究人员寻找circRNA具体研究思路提供帮助。此外, 该平台界定了超circRNA区域 (super circular RNA regions) 范围, 并且提供了相应的检测模块, 表明其与反向剪接和叶绿体功能等有一定相关性。该平台的建立弥补了circRNA植物组织特异性注释领域的空白, 对植物组织功能的研究有重要的意义。
2 CircRNA相关功能分析数据库
circRNA作为ncRNA, 大量研究表明其身份并不单一, 除了在前言中叙述的功能模式, Ying 等提出了microRNA新的调控机制, 提示ncRNA也有可能通过外泌体进行旁分泌或内分泌调节发挥功能[18]。可见, 众多功能模式的神秘面纱还未被揭开, 而支持功能分析的数据库的建立必将加速该领域研究进程。
2.1 deepBase 2.0
deepBase是可供分析预测19个不同物种的ncRNA的演变、表达模式和功能的平台[19]。2016年, 中山大学RNA信息中心研究人员将deepBase升级为v2.0[20]。deepBase v2.0从NCBI GEO和SRA数据库中检索了558个小RNA数据集和478个转录物组测序 (RNA-Seq) 数据集。从中识别出所有的ncRNA, 包括长非编码RNA (lncRNA) 以及circRNA, 并将其数据分析规整, 显示在集成交互式可视化的web页面中, 以显示多维数据, 促进转录物研究和发现新的ncRNA。
2.2 Circlnteractome
Gorospe等人于2016年1月构建了CircInteractome数据库[21], 预测了109个RBP与circRNA的结合位点, 同时利用Targetscan软件预测了miRNA与circRNA的潜在结合位点并进行整合分析。除了基本信息, 相关研究者可以 (1) 预测circRNA上的RBP结合位点; (2) 预测circRNA上的miRNA结合位点; (3) 识别潜在的内部核糖体进入位点 (internal ribosome entry site, IRES) ; (4) 设计用于沉默circRNA的siRNA; (5) 进行特定circRNA的引物设计及序列获取。一些研究发现, circRNA分子可直接与蛋白质互作。这在另一层面揭示了circRNA的调控途径:一方面circRNA可以动态调控蛋白质, 另一方面蛋白质也可反调控于circRNA[22]。该数据库对circRNA的RBP结合位点的预测, 在一定程度上推动了该领域的发展, 有助于进一步发现和研究目前还有着众多未解之谜的circRNA功能模型。该数据库对miRNA结合位点的预测以及circRNA引物设计可为研究提供极大的便利。
2.3 CircNet
circRNA、miRNA 和基因之间的互作调控是目前研究热点之一。2016年1月, 我国台湾国立交通大学研究人员利用464个RNA-seq测序数据, 整合挖掘并建立了CircNet数据库[23]。这是第1个提供组织特异性的circRNA表达谱以及circRNA-miRNA-mRNA调控网络的公共信息平台。该数据可进行新circRNA预测及基因组的注释, 并对circRNA亚型的序列以及表达水平进行分析, 构建circRNA-miRNA-mRNA调控网络。该数据库支持通过预测circRNA的基因来源或miRNA结合位点进而对circRNA进行功能预测, 这对于circRNA功能和调控机制的研究意义重大。CircNet数据库的一大特点在于运用了该作者发布的新的circRNA命名系统[24]。该系统对circRNA进行了编目, 提供有关源基因的信息, 使得来源于同一基因的不同circRNA亚型得以通过名字被区分开, 标准化的命名系统势必利于circRNA的生物信息学分析和实验研究。
2.4 circRNADb
研究数据显示, circRNA上也存在开放阅读框 (open reading frame, ORF) 、内部核糖体进入位点, 以及m6A等修饰[25], 表明其可能参与多肽或蛋白质的翻译。circRNADb数据库[26]共收集了32 914条人类外显子circRNA资料, 包括基因组位置信息、RNA编辑情况、所对应的基因组序列、IRES序列元件、预测的ORF以及相关的参考文献。该收录数据的分析表明, 有16 328条circRNA包含了编码超过100个氨基酸的ORF, 其中7 170种circRNA存在IRES序列元件, 基本符合翻译蛋白质的特征。研究者可以根据基因名称, PubMed ID及细胞或组织类型等多种检索条件进行检索, 从而获取可能存在的具备蛋白质翻译潜能的circRNA, 同时也提供相关质谱信息供参考。该数据库的资料收录相对严苛, 因此数据有一定的局限性。另外, 根据预测可能翻译的蛋白质, 该数据库还提供了结构域、N-糖基化位点、O-糖基化位点、磷酸化位点等翻译后修饰位点和半衰期等蛋白质信息, 有助于研究人员识别和预测其潜在的生物学功能。
2.5 PlantcircBase
PlantcircBase是首个用于植物circRNA研究的数据库[15], 发表于2017年, 收录了近几年实验证据以及生物信息学预测的77 595个circRNA, 包含了亚洲栽培稻、拟南芥、玉米、番茄以及大麦等5个物种。该平台支持circRNA-miRNA-mRNA互作分析预测, 同时提供基因上的反向剪接位点和可能产生的circRNA的预测, 并且可实现数据可视化。该数据库试图涵盖多种研究所得以及预测的植物相关circRNA, 对植物circRNA功能研究领域有极大的推动作用, 填补了领域空缺。
2.6 PlantCircNet
相比于PlantcircBase, 发表于2017年的数据库PlantCircNet[16]收录了更多的植物circRNA以及circRNA-miRNA-mRNA互作网络信息。该数据库涵盖信息分析和鉴定预测的circRNA来源, 为拟南芥、二穗短柄草、大豆、大麦、亚洲栽培稻、番茄、小麦和玉米等8个物种。同时, 还提供了circRNA的类型、反向剪接位点、亲本基因、相关亚型以及表达水平等基本信息, 进一步加速了该领域信息的完善和研究进度。
2.7 TSCD与CSCD
目前, 虽然已有大量具有细胞特异性和疾病相关的circRNA被发现, 表明了circRNA的表达以及功能发挥与组织特异性和疾病发生发展相关[27], 但关注组织特异性和疾病特异性的circRNA数据库较少, 该类数据库的完善将有利于对组织器官的发育和分化以及疾病相关的circRNA的深入研究。He和Han团队针对该方面的功能挖掘, 于2017年11月和2018年1月先后发表了全新的组织特异性分析circRNA数据库TSCD[28]和肿瘤特异性circRNA数据库CSCD[29]。其中, TSCD是检索人和小鼠中组织特异性circRNA信息的数据库, 包括成人和胚胎等26种不同组织, 共收录了302 853个circRNA, 同时也发现了大脑组织中存在着大量的组织特异性circRNA。该数据库可供检索不同组织中特异性表达的circRNA的信息, 包括circRNA ID、基因组位置、基因组跨度长度和组织类别等, 提供了组织特异性circRNA的全局视图。此外, 该数据库还能显示单独的比较页面, 方便不同组织之间的circRNA比较。CSCD利用了4种常用的反向拼接算法:CIRI2[30]、find_circ[1]、circRNA_finder[31]和CIRCexplorer[32], 收录条件相对宽松, 从ENCODE中收集了19种肿瘤类型的87种细胞系以及141种正常细胞的circRNA数据, 分析汇总了272 152种肿瘤特异性的circRNA, 950 962种正常细胞特异的circRNA, 还有170 909种为肿瘤和正常样本共有的circRNA。一共1 394 023种circRNA分子, 其中外显子来源的有759 039种, 相应的, 内含子来源436 668种、基因间序列来源122 806种, 这与其他数据库相比, 极大提高了circRNA记录的数量。检索结果显示基因和circRNA两种界面, 可以对肿瘤相关的circRNA进行亚细胞定位分析、MRE预测、RBP结合位点预测、ORF预测和相关基因可变剪接分析等, 以帮助肿瘤相关circRNA的功能研究。CSCD也收集正常组织的circRNA信息, 它允许用户进行其它的功能预测研究。这两个数据库的问世将加速组织特异性和疾病特异性相关的circRNA的功能研究, 有助于circRNA功能数据的进一步丰富。
2.8 circlncRNAnet
众多研究表明, ncRNA在基因调控中发挥重要的作用, 但是由于其靶基因的广泛性和作用方式复杂性, 其具体调控网络成员以及机制研究存在重重困难, 因此, 建立更加整合的数据基础显得尤为重要。2018年1月, 我国台湾科学家发表了整合lncRNA和circRNA功能网络的在线分析数据库circlncRNAnet[33], 收录了TCGA数据库20多个肿瘤类型数据, 支持在线可视化分析。用户可提交自己的lncRNA和circRNA芯片或测序所得的数据, 并且可以和TCGA数据库实现共享。该数据库操作简易, 分析功能庞大, 分析模块包括lncRNA组织表达热图、箱线图、基因功能富集GO分析和KEGG图、RBP-RNA结合蛋白质网络图、miRNA网络图等。此外, 还可在线制作ceRNA网络图等, 是研究circRNA分子互作及其功能的良好信息平台。
3 与人类疾病相关的CircRNA数据库
circRNA与疾病的相关性一直是研究的热点, 其中以肿瘤学为主。2017年以来, 循环系统包括血管重塑性疾病、心血管疾病等研究也呈现上升趋势。该方面数据库的收集和建立, 将有利于对疾病的发生发展以及诊断和转归有更进一步的了解。
3.1 Circ2Traits
Ghosal等于2013年构建了第1个circRNA与人类疾病潜在关联的数据库[34]。通过分析circRNA与疾病相关的miRNA的相互作用和与疾病相关SNP的关联程度, 观察circRNA与人类疾病的潜在联系, 构建了相关分子互作网络, 并对miRNA-circRNA相互作用组中的蛋白质编码基因进行了GO富集分析。可从该数据库中查看与疾病相关的circRNA, 以及与Ago相互作用位点等信息。该数据库提供了一个circRNA在各种人类疾病中与miRNA互作的信息平台, 但预测功能方面稍显薄弱, 有待进一步收集数据、完善和优化。
3.2 exoRBase
外泌体是细胞衍生的囊泡结构, 直径约为30~150 nm, 存在于许多真核生物液体中, 包括血液、尿液、唾液和细胞培养基[35,36,37,38]。外泌体携带有蛋白质、DNA以及RNA, 其中也包括circRNA等ncRNA[39]。越来越多的证据表明, 通过转运这些细胞内分子, 外泌体可能在癌细胞生存[40]、凝血[41]、细胞间信号传递[42]和废物处理[39]等过程中发挥关键作用。而外泌体的分子成分则和分泌细胞关系密切, 后者可在不同的生理和病理过程中分泌不同的分子[43]。因此, 外泌体有作为健康和疾病的生物标志物, 具有用于疾病治疗的前景。而circRNA分子是封闭环形结构, 耐受核酸外切酶降解, 比线性RNA更加稳定, 更适合作为诊断标志物, 对各种人类疾病进程的分子表现更为准确。2017年, 复旦大学医学院上海肿瘤研究所黄胜林教授发表了专门收录外泌体中各种长链RNA的数据库:exoRBase[44], 收录了来自92份血清外泌体RNA-seq测序样本的15 501种lncRNA, 18 333种mRNA, 58 330种circRNA。组织来源包括正常人、冠心病、结肠直肠癌、肝癌、胰腺癌和乳腺癌等。同时, 该数据库还支持其他研究人员上传新的数据。利用exoRBase可获得正常组织和所研究疾病的RNA表达谱, 通过限定不同的规则和选项提取感兴趣的RNA分子。exoRBase数据库作为一个外泌体以及疾病相关的信息平台, 将帮助人们识别血液外泌体中的分子特征, 并将促进新的疾病相关循环生物标志物的发现, 以及提示其在人类疾病中发挥的功能。
3.3 circRNA disease
2018年4月, Zhao 等[45]发表了收集人类疾病相关circRNA的数据库。该数据库的资料主要是收录通过PubMed检索circRNA和disease关键词后, 分析汇总的相关研究结果。系统回顾了800多篇研究论文, 共收集了354项研究中共330种circRNA以及其相关的48类疾病, 其中癌症占了54%, 心血管疾病占了27%。该平台支持通过circRNA的名称或者疾病名称来进行搜索, 得到相应的基本信息, 并且提供circRNA在circBase的链接, 有助于理解circRNA在人类疾病中扮演的角色。
3.4 CircR2Disease
CircR2Disease[46]提供了大量和人类疾病相关的circRNA调控紊乱的资料以供研究, 包含了661个circRNA和100种人类疾病之间的725例信息, 每一个条目都包含circRNA – 人类疾病关系的详细信息, 整合了多种生物功能, 并且保持着每2个月更新的速度。在数据定位和分析过程中, 作者引入了疾病谱宽度 (disease spectrum width, DSW) 以及疾病的circRNA谱宽度 (circRNA spectrum width, CSW) 作为衡量circRNA和疾病之间关系的指标之一, 使得相关的预测更加准确。
4 问题与展望
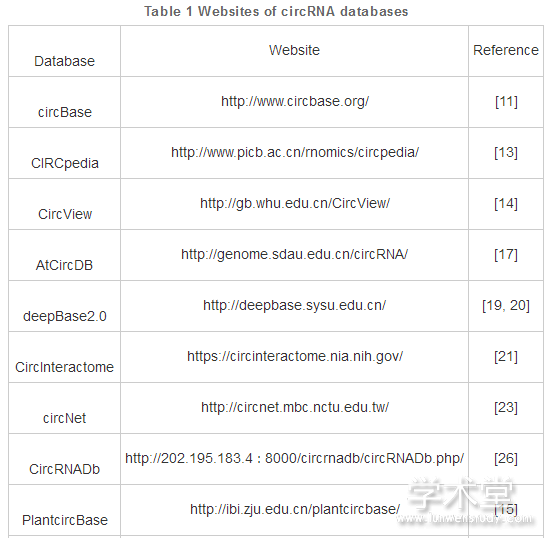
本文所描述的数据库对应的网站见Table 1。现有的数据库虽然涵盖了大量的circRNA基本信息以及元件和功能预测, 为研究者提供了极大便利。但是, 物种覆盖面若能更广, 组织分类以及细胞分类更加完善, 则更有利于多种样品的探索。目前, 植物中的circRNA研究依旧存在着较多的空白。其次, circRNA发挥功能的机制模型已经有多种, 但是现有数据库普遍关注蛋白质翻译潜能以及ceRNA功能预测, 而针对其它功能模型, 例如circRNA-蛋白质互作, 外泌体分泌等其余机制的数据库还较为匮乏;而且实际应用这些数据库时需结合特定的研究方向和内容, 通常会选取相应的一个或多个数据库, 这就暴露出不同数据库之间的数据存在的形式以及数据本身的差异存在的问题。另外, circRNA的数据库主要针对于一级结构, 而对于二级结构的预测、功能域、功能分析还有待进一步开发。值得一提的是, circRNA目前未能有如同miRNA规范统一的命名系统, 这给数据分析也带来了一定的阻碍。目前, 数据库建立所依赖的计算方法已有数十种, 如CIRCexplorer2[13], DCC[47], CircPro[48], sail?sh-cir[49]等。但若能进一步优化, 能够针对更多的数据类型综合计算、兼容不断发展的新技术、开拓更多的功能分析以及更准确的定量分析, 将会更进一步推动该领域的发展[50]。

参考文献:
[1] Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495 (7441) : 333-338
[2] Qu S, Zhong Y, Shang R, et al. The emerging landscape of circular RNA in life processes[J]. RNA Biol, 2017, 14 (8) : 992-999
[3] Holdt LM, Kohlmaier A, Teupser D. Molecular roles and function of circular RNAs in eukaryotic cells[J]. Cell Mol Life Sci, 2018, 75 (6) : 1071-1098
[4] Zhang Y, Zhang XO, Chen T, et al. Circular intronic long noncoding RNAs[J]. Mol Cell, 2013, 51 (6) : 792-806
[5] Li Z, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nat Struct Mol Biol, 2015, 22 (3) : 256-264
[6] Li P, Yang X, Yuan W, et al. CircRNA-Cdr1as exerts anti-oncogenic functions in bladder cancer by sponging microRNA-135a[J]. Cell Physiol Biochem, 2018, 46 (4) : 1606-1616
[7] Ashwal-Fluss R, Meyer M, Pamudurti NR, et al. circRNA biogenesis competes with pre- mRNA splicing[J]. Mol Cell, 2014, 56 (1) : 55-66
[8] Holdt LM, Stahringer A, Sass K, et al. Circular non-coding RNA ANRIL modulates ribosomal RNA maturation and atherosclerosis in humans[J]. Nat Commun, 2016, 7: 12429
[9] Yang Y, Fan X, Mao M, et al. Extensive translation of circular RNAs driven by N6- methyladenosine [J]. Cell Res, 2017, 27 (5) : 626–641
[10] Qu S, Liu Z, Yang X, et al. The emerging functions and roles of circular RNAs in cancer[J]. Cancer Lett, 2018, 414: 301-309
[11] Gla?ar P, Papavasileiou P, Rajewsky N. circBase: a database for circular RNAs[J]. RNA, 2014, 20 (11) : 1666-1670
[12] Gao Y, Wang J, Zheng Y, et al. Comprehensive identification of internal structure and alternative splicing events in circular RNAs[J]. Nat Commun, 2016, 7: 12060
[13] Zhang XO, Dong R, Zhang Y, et al. Diverse alternative back-splicing and alternative splicing landscape of circular RNAs[J]. Genome Res, 2016, 26 (9) : 1277-1287
[14] Feng J, Xiang Y, Xia S, et al. CircView: a visualization and exploration tool for circular RNAs[J]. Brief Bioinform, 2017Jun 30. doi: 10.1093/bib/bbx070
[15] Chu Q, Zhang X, Zhu X, et al. PlantcircBase: A database for plant circular RNAs[J]. Mol Plant, 2017, 10 (8) : 1126-1128
[16] Zhang PJ, Meng XW, Chen HJ, et al. PlantCircNet: a database for plant circRNA- miRNA- mRNA regulatory networks[J]. Database, 2017, doi: 10.1093/database/bax089
[17] Ye J, Wang L, Li S, et al. AtCircDB: a tissue-specific database for Arabidopsis circular RNAs[J]. Brief Bioinform, 2017, 1-11.doi: 10.1093/bib/bbx089
[18] Ying W, Riopel M, Bandyopadhyay G, et al. Adipose tissue macrophage-derived exosomal miRNAs can modulate in vivo and in vitro insulin sensitivity[J]. Cell, 2017, 171 (2) : 372-384
[19] Yang JH, Shao P, Zhou H, et al. deepBase: a database for deeply annotating and mining deep sequencing data[J]. Nucleic Acids Res, 2010, 38 (Database issue) : D123- D130
[20] Zheng LL, Li JH, Wu J, et al. deepBase v2.0: identification, expression, evolution and function of small RNAs, LncRNAs and circular RNAs from deep-sequencing data[J]. Nucleic Acids Res, 2016, 44 (D1) : D196- 202
[21] Dudekula DB, Panda AC, Grammatikakis I, et al. CircInteractome: A web tool for exploring circular RNAs and their interacting proteins and microRNAs[J]. RNA Biol, 2016, 13 (1) : 34-42
[22] Du WW, Yang W, Liu E, et al. Foxo3 circular RNA retards cell cycle progression via forming ternary complexes with p21 and CDK2[J]. Nucleic Acids Res, 2016, 44 (6) : 2846-2858
[23] Liu YC, Li JR, Sun CH, et al. CircNet: a database of circular RNAs derived from transcriptome sequencing data[J]. Nucleic Acids Res, 2016, 44 (D1) : D209- D215
[24] Jeck WR, Sharpless NE. Detecting and characterizing circular RNAs[J]. Nat Biotechnol, 2014, 32 (5) : 453-461
[25] Zhou C, Molinie B, Daneshvar K, et al. Genome-wide maps of m6A circRNAs identify widespread and cell-type-specific methylation patterns that are distinct from mRNAs[J]. Cell Rep, 2017, 20 (9) : 2262-2276
[26] Chen X, Han P, Zhou T, et al. circRNADb: A comprehensive database for human circular RNAs with protein-coding annotations[J]. Sci Rep, 2016, 6: 34985
[27] Xu T, Wu J, Han P, et al. Circular RNA expression profiles and features in human tissues: a study using RNA-seq data[J]. BMC Genomics, 2017, 18 (Suppl 6) : 680
[28] Xia S, Feng J, Lei L, et al. Comprehensive characterization of tissue-specific circular RNAs in the human and mouse genomes[J]. Brief Bioinform, 2017, 18 (6) : 984-992
[29] Xia S, Feng J, Chen K, et al. CSCD: a database for cancer-specific circular RNAs[J]. Nucleic Acids Res, 2018, 46 (D1) : D925-D929
[30] Zheng Y, Zhao F. Detection and reconstruction of circular RNAs from transcriptomic data[J]. Methods Mol Biol, 2018, 1724: 1-8
[31] Westholm JO, Miura P, Olson S, et al. Genome-wide analysis of drosophila circular RNAs reveals their structural and sequenceproperties and age-dependent neural accumulation[J]. Cell Rep, 2014, 9 (5) : 1966-1980
[32] Zhang XO, Wang HB, Zhang Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159 (1) : 134-147
[33] Wu SM, Liu H, Huang PJ, et al. circlncRNAnet: an integrated web-based resource for mapping functional networks of long or circular forms of noncoding RNAs[J]. Gigascience, 2018, 7 (1) : 1-10
[34] Ghosal S, Das S, Sen R, et al. Circ2Traits: a comprehensive database for circular RNA potentially associated with disease and traits[J]. Front Genet, 2013, 4: 283
[35] Keller S, Sanderson MP, Stoeck A, et al. Exosomes: from biogenesis and secretion to biological function[J]. Immunol Lett, 2006, 107 (2) : 102-108
[36] van der Pol E, B?ing AN, Harrison P, et al. Classification, functions, and clinical relevance of extracellular vesicles[J]. Pharmacol Rev, 2012, 64 (3) :676-705
[37] Bryzgunova OE, Zaripov MM, Skvortsova TE, et al. Comparative study of extracellular vesicles from the urine of healthy individuals and prostate cancer patients[J]. PLoS One, 2016, 11 (6) : e0157566
[38] Ogawa Y, Kanai-Azuma M, Akimoto Y, et al. Exosome-like vesicles with dipeptidyl peptidase IV in human saliva[J]. Biol Pharm Bull, 2008, 31 (6) : 1059-1062
[39] Lasda E, Parker R. Circular RNAs Co-precipitate with extracellular vesicles: a possible mechanism for circRNA clearance[J]. PLoS One, 2016, 11 (2) : e0148407
[40] Syn N, Wang L, Sethi G, et al. Exosome-mediated metastasis: from epithelial-mesenchymal transition to escape from immunosurveillance[J]. Trends Pharmacol Sci, 2016, 37 (7) : 606-617
[41] Kapustin AN, Shanahan CM. Emerging roles for vascular smooth muscle cell exosomes in calcification and coagulation[J]. J Physiol, 2016, 594 (11) : 2905-2914
[42] Teng Y, Ren Y, Hu X, et al. MVP-mediated exosomal sorting of miR-193a promotes colon cancer progression[J]. Nat Commun, 2017, 8: 14448
[43] Fujita Y, Yoshioka Y, Ochiya T. Extracellular vesicle transfer of cancer pathogenic components[J]. Cancer Sci, 2016, 107 (4) : 385-390
[44] Li S, Li Y, Chen B, et al. exoRBase: a database of circRNA, lncRNA and mRNA in human blood exosomes[J]. Nucleic Acids Res, 2018, 46 (D1) : D106-D112
[45] Zhao Z, Wang K, Wu F, et al. circRNA disease: a manually curated database of experimentally supported circRNA-disease associations[J]. Cell Death Dis, 2018, 9 (5) : 475
[46] Fan C, Lei X, Fang Z, et al. CircR2Disease: a manually curated database for experimentally supported circular RNAs associated with various diseases[J]. Database (Oxford) , 2018, doi: 10.1093/database/bay044
[47] Cheng J, Metge F, Dieterich C. Specific identification and quantification of circular RNAs from sequencing data[J]. Bioinformatics, 2016, 32 (7) : 1094-1096
[48] Meng X, Chen Q, Zhang P, et al. CircPro: an integrated tool for the identification of circRNAs with protein-coding potential[J]. Bioinformatics, 2017, 33 (20) : 3314-3316
[49] Li M, Xie X, Zhou J, et al. Quantifying circular RNA expression from RNA-seq data using model-based framework[J]. Bioinformatics, 2017, 33 (14) : 2131-2139
[50] Gao Y, Zhao F. Computational strategies for exploring circular RNAs[J]. Trends Genet, 2018, 34 (5) : 389-400





