植物生物技术论文第八篇:基因分型技术的原理、流程及其在植物领域的应用
摘要:随着高通量测序技术的发展, 一种高效而经济的SNP (single nucleotide polymorphism) 发掘和基因分型技术GBS (genotyping by sequencing) 被开发出来, 避免了传统基因型鉴别方法的不足, 现已被广泛应用于植物遗传图谱构建、全基因组关联分析、基因多样性研究以及种质鉴定和品种识别等多个领域。从GBS技术的原理、流程、特点以及应用等方面对近年来植物应用GBS技术所进行的研究进展进行综述, 并提出了今后GBS技术的研究重点, 以期为GBS技术在植物的应用研究提供参考。
关键词:GBS技术; 植物; 应用领域; 研究进展;
Research Progress of GBS Technology in Plant
QIAO Yan SUN Jiaqing WEI Ruqian LIU Yue
Key Laboratory of Ethnomedicine, Ministry of Education, Minzu University of China College of Life and Enviironmental Sciences, Minzu University of China National Resource Center for Chinese Materia Medica, China Academy of Traditional Chinese Medicine
Abstract:With the development of high throughput sequencing technology, GBS (genotyping by sequencing) technique, an economic technique to discover SNP and genotype myriad of crop germpasms in an effective way, has been developed, which avoids the deficiency of traditional genotypic identification methods. It has been widely used in many fields including construction of genetic map, genome-wide association study, genetic diversity and germplasm dentification and variety identification. In this paper, the research progress of GBS technology in plants was reviewed from the principle, process, characteristics and application. And the research emphasis on GBS technology was putting forward in the future, which was expected to provide reference for the future research of GBS technology on plants.
伴随着新一代测序技术的逐渐成熟,高通量测序技术,又称为下一代测序技术,逐渐被广泛认可和利用,并为植物基因组学研究提供充分信息[1]。下一代测序技术提供的数据信息为遗传信息的发掘和基因表达调控等基础性研究做出了很大的贡献。简化基因组测序技术 (reducedrepresentation sequencing) 是目前比较高效的适用于群体分析的手段,已有的方法主要有限制性片段长度多态性、扩增片段长度多态性等[2]。而新型的基因分型方法基于测序基因分型技术 (genotyping by sequencing, GBS) ,由于其快速、简便、低成本等优点逐渐被应用于多种植物的研究中[3,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18]。为了更好地了解和应用GBS技术,本文从原理、技术流程、特点以及应用领域4个方面对近年来GBS技术研究进展进行综述,以期为以后GBS技术的改良以及在植物领域更好地应用提供参考。
1 GBS测序技术的原理
GBS技术是目前较为常见的一种简化基因组测序技术[3]。该技术通过测序进行基因分型,其命名是为了与传统的通过芯片进行基因分型的方法相区别,原理是将基因组DNA进行酶切,然后对酶切片段进行高通量测序,通过分析获得SNP信息并进行基因分型的分子标记的方法[4]。
为确定样本的基因分型,GBS技术将样本的基因组利用物理手段 (如超声波等) 或者选用内切酶对DNA进行酶切,将其打断成一些小的片段,然后对带有标签序列的小片段进行富集,对利用高通量测序技术测得的小片段两端的序列进行数据分析得到被测基因组大量的SNP信息,原理如图1所示。该技术可以根据研究的灵活性来调整捕获酶切位点所需的片段 (tags) 的数量,从而控制捕获序列的范围,极大地降低了遗传多样性较高物种的研究挑战性。
图1 GBS测序技术原理[3]
2 GBS技术关键流程
2.1 限制性内切酶选择
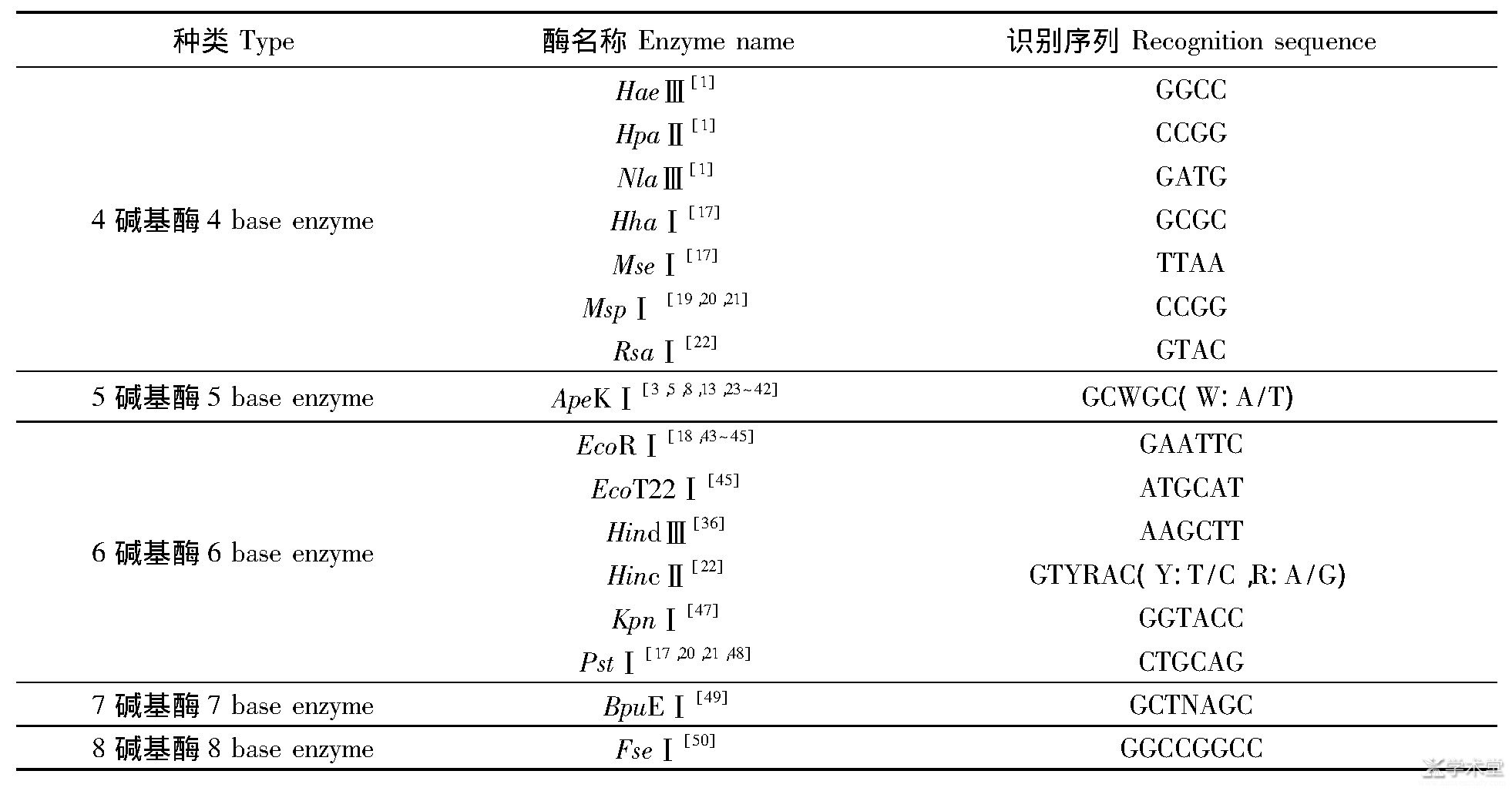
提取所研究物种DNA后,应用限制性内切酶进行酶切。根据所研究植物的DNA序列特点,选择合适的限制性内切酶对DNA进行切割。Ape KⅠ酶在所研究物种中的应用频率是最高的,该酶是一种Ⅱ型限制性内切酶,在所研究物种DNA的转座子区几乎没有识别位点,且部分甲基化敏感,如果两条链上识别序列3'-端的碱基是5'-甲基胞嘧啶则不会被限制性内切酶切断,从而避免了DNA片段长度过短的问题[3]。Ape KⅠ酶广泛应用于玉米、大麦、小麦、大豆等作物。对目前应用GBS技术在植物中使用的限制性内切酶进行总结归类,由于四碱基酶在DNA样品中的切割相对频繁,得到的片段最小,有益于下一步的处理,因此已有的研究中四碱基酶的利用相对较多。GBS技术限制性内切酶种类如表1所示。
在开发GBS技术的时候,通常会遵守以下三个原则: (1) 尽量避开重复序列; (2) 酶切位点在基因组上均匀分布; (3) 根据研究的目的,灵活地调整所需要的酶切位点的片段数量 (tags) [1]。
表1 GBS技术限制性内切酶种类
2.2 测序文库的构建
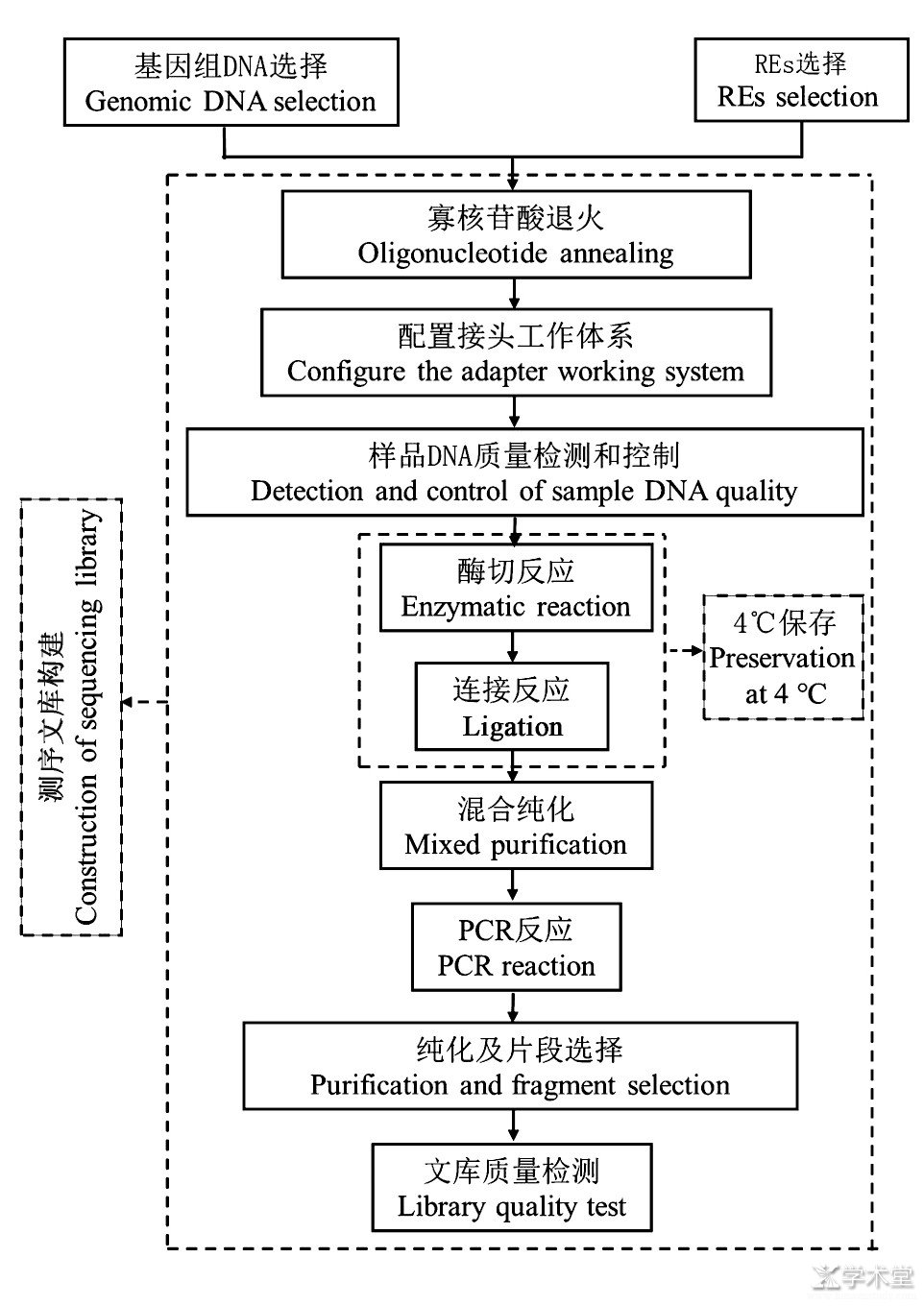
GBS文库构建过程中,在应用限制性内切酶进行酶切后,在酶切片段两端加上带Barcode接头,对每个样品进行PCR扩增,然后对样品进行混合,选择需要的片段构建文库,利用Illumina Hiseq测序平台进行末端测序。GBS文库构建过程如图2所示。
2.3 数据分析
GBS技术的应用可以通过有无参考基因组分为两类,对于有参基因组的物种可以将测序结果与基因组对比进行多态性鉴别[42]。对得到的Raw Data进行质量控制得到Clean Data,将其与参考基因组进行比对,根据比对结果对SNP进行检测和注释。对于没有参考基因组的物种来说,想要获得大批量SNP数据存在着一定的限制,GBS方法从技术上解决了这一问题,原理为利用合适的限制性内切酶对完整的基因组DNA进行酶切,之后利用序列信息标识来源不同的基因型个体,利用测序得到的信息将不同个体短序列进行聚类,通过序列分析得到SNP位点在群体中的变异位点[23]。对SNP可以进行群体遗传结构分析,主要包括系统进化树的构建、主成分分析和群体结构分析。以及对群体遗传多样性进行分析,包括Ho (观测杂合度) 、He (预期杂合度) 和核苷酸多样性分析[13]。
图2 GBS文库构建流程
2.4 GBS技术的特点
RFLP、RAPD、AFLP、SSR等传统鉴别基因型的方法已广泛应用于许多方面,但需要耗费巨大的人力财力,所需实验时间长,并且在基因组中的丰富性较差。GBS是一种简化基因组测序技术,避免了传统方法的一些不足。虽然不足以覆盖全基因组深度,但是由于其省时、价廉、有效的特点而得到普及[1]。GBS技术在发展之初就是为了降低较大基因组测序的复杂程度,其设计原理上的优势主要体现在: (1) 测序前无需了解基因组的信息; (2) Barcode的长度为4~8碱基,允许连接成百上千的DNA样品进行测序,减少了基因型鉴定的时间; (3) 限制性内切酶的选择,可以控制酶切片段的长度,简化了基因组的富足性,便于进行下一代测序; (4) 从成本方面进行考虑,实验所需的酶体系可以自己配置,与购买试剂盒相比,便宜10倍之多,更有利于高密度SNP鉴定[16]。
如今GBS技术已广泛应用于大豆、大麦、小麦等基因组较大、重复度较高的生物上,并在诸多方面进行了改良。Sonah等[14]对豆科植物进行研究,降低基因复杂度,对GBS技术进行改进,在8个遗传多样性的大豆中开发10 120个高质量SNP标记,且其分布接近基因丰富的区间。其中位于编码区和基因区间的分别占52.5%和39.5%,之后用SANGER法得到验证。在GBS测序流程中使用特异性引物,使SNP数量和覆盖度均得到提升。Poland等[4]对GBS单酶切方法进行改进,对大麦和小麦的基因组DNA进行PstⅠ和MspⅠ双酶切,对大麦、小麦双亲杂交群体构建不依赖参考基因组的遗传图谱,为基因组关联的植物育种奠定基础。
3 GBS技术在植物领域的应用
对应用GBS技术得到的SNP标记可以进行遗传图谱构建、遗传图谱加密、全基因组关联分析 (GWAS) 等相关研究 (表2) 。这些研究对于GBS技术未来在植物育种,保护,全球物种或种群的调查中起到至关重要的作用。
3.1 遗传图谱构建
遗传图谱是指基因或遗传标记在染色体上以一定顺序排列的图谱,根据基因连锁和基因交换值重组来得到基因和遗传标记的排列顺序[10]。
2012年,Poland等[4]在传统GBS技术方法上进行改进,开发了一种双酶切GBS流程,应用于诸如小麦、大麦这种拥有大型复杂基因组的物种,以开发一个基因锚定的参考图谱。在Oregon Wolfe大麦图谱上得到了34 000个SNP和240 000个tags,与此同时在W97846Opata85 (SynOpDH) 小麦参考图谱上进行作图得到20 000个SNP和367 000个标签,用GBS技术处理得到的SNP标记构建了小麦的遗传图谱。Crossa等[10]利用GBS技术对覆盆子进行遗传图谱构建,通过SSR与已有的R.idaeus图谱相比较,利用GBS构建的连锁图谱具有较强的可靠性,识别畸变的基因组区域,有助于鉴别该物种由于近亲繁殖而产生的有害等位基因。秦伟伟等[12]以玉米自交系黄早四 (HZS) 和Mo17为亲本,构建130个重组自交系的RIL群体,利用GBS测序技术得到的高密度多态性SNP位点,采用完备区间作图法,对不同环境中生长的玉米的不同性状分别进行QTL定位,利用JoinMap4.0软件对重组区块的标记构建其遗传图谱,并且采用完备区间作图法对5个环境下的4个形状分别进行QTL定位,该研究不仅为分子标记辅助选择籽粒性状提供了实用标记,而且为主效基因的进一步精细定位和候选基因挖掘奠定了基础。
通过这些研究表明,GBS技术对基因定位起到了非常重要的作用,具有很高的可靠性,对于分子育种提供了更多依据,利用GBS技术对植物遗传图谱的构建可以使人类对植物的研究愈加深化。
3.2 全基因组关联分析
GBS技术也可以应用于植物的全基因组关联分析 (genome-wide association study, GWAS) ,优势是使被测目标物种基因组的复杂程度大幅度降低,以及测定成本也会大幅减少,非常适合基因组较大较复杂的物种。
基因组较大的物种,如玉米、豇豆等,GBS技术所测得的大量SNP,广泛应用于全基因组关联分析。Romay等[48]应用GBS技术对美国国家种质库中2 815份玉米自交系的基因型进行鉴定,获得将近70万个分布在整个基因组中的SNP,通过GBS技术对遗传距离测定,得到的数据用于全基因组关联分析,其结果有利于更好的探索植物复杂性状的遗传结构。熊海铮等[56]通过USDA (美国农业部) 收集到502份核心种质和52份美国商业种共716份豇豆资源,分别从两方面对得到的豇豆资源进行研究,利用GBS技术从供体豇豆中获取SNP标记,对其表现型和基因型进行多方面的研究,主要包括对豇豆群体遗传多样性、群体结构及系统聚类进行分析,与此同时对8个农艺性状如结荚数、结荚位置、荚长、株高等方面进行全基因组关联作图,有利于对豇豆的关联分析研究,并为基因辅助育种提供参考。
表2 应用GBS技术进行研究的植物
GBS技术还可以应用于植物疾病相关的研究。Yu等[33]用GBS技术对紫花苜蓿抗黄萎病进行简化全基因组关联分析。通过人工接种病原体对健康幼苗进行表型分型,并对其抗病性进行评分,连锁不平衡关联分析确定了10个SNP与VW抗性显著相关,发现的位于第7号和第8号染色体上抗性位点标记与前人报道的QTL定位结果相同。使用抗性基因座侧翼序列对基因组两两比对 (BLASTN) 鉴定出具有假定抗病功能的潜在候选基因。进一步研究表明,这些标记可通过标记辅助选择实现育种计划,最终导致苜蓿VW抗性的提高。
由此可见,GBS测序可以测得SNP,结合全基因组关联分析,有助于发现探索植物新性状相关的变异来源,对今后品种选育、驯化改良都有一定的指导意义。
3.3 基因多样性研究
GBS技术还广泛用于基因多样性研究。Lin等[58]用GBS技术对一个PHS抗性亲本和一个PHS易感亲本杂交进行处理所获得的SNP,对155个重组自交系 (recombinant inbred lines, RIL) 进行评估,鉴定小麦的穗发芽抗性与种子休眠相关的数量性状位点,然后对GBS109947和GBS212431进行作图,对小麦的基因型多样性进行研究。GBS技术在不断发展,Lu等[57]对柳枝稷样本个体进行基因分型,开发了一种称为UNEAK的SNP发掘方法,可以避免SNP发现的复杂性。
3.4 种质鉴定及品种识别等
在农业栽培过程中,植物物种的表型和基因型都会在漫长的驯化改良过程中发生变化,利用GBS技术对这些植物进行研究,可以鉴定出其不同品种的性状及对环境的适应所发生的变化。
Sim等[54]采用GBS技术对番茄基因组变异的选择进行了研究,超过97%的SNP呈现多态性。对其进行PCA主成分分析,近交系被分为7个亚群。在当代改良种亚群中可以进一步划分,区分了三个有代表性的栽培番茄 (加工、新鲜市场和年份) 亚群。所观察到的SNP变化和LD衰减表明栽培番茄的遗传变异的不同模式是由于野生物种的渗入和市场专业化的选择。在品种识别方面,王小柯等[42]采用GBS技术对具有广泛遗传多样性的240份宽皮柑橘进行研究,用限制酶进行酶切后对基因组DNA进行测序,经过一定条件的筛选过滤,最后共获得了114 200个高质量SNP位点,之后通过构建进化树以及PCA分析,结果显示240份宽皮柑橘被分成四大类,通过GBS技术完成了宽皮柑橘的品种识别。
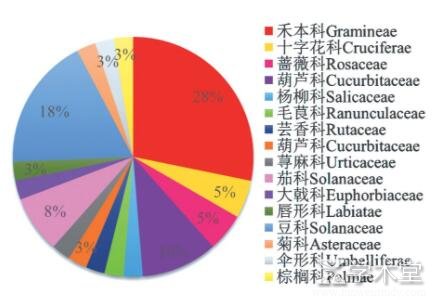
根据在数据库中所检索的文献,列出了在2011-2018年之间应用GBS技术所研究植物的种类,共39种,禾本科11种 (28.21%) 、豆科7种 (17.95%) 、葫芦科4种 (10.26%) ,其他17种 (43.59%) ,如图4所示。
GBS技术主要应用的植物主要是禾本科植物,如玉米、大麦、小麦、高粱、南瓜等农作物,近年来也逐渐应用于牡丹等观赏植物以及柑橘等经济作物。对洋甘菊、山羊草和羽扇豆等药用植物的研究也对以后利用GBS技术对药用植物进行处理分析具有很好的指导意义。
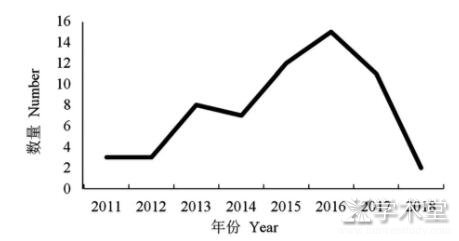
图3 各年份文献数量
图4 各科植物在研究中所占比重
4 展望
下一代技术的进步推动了DNA测序的成本下降,有助于推动高多样性、大基因组物种的研究。GBS技术现已应用在很多方面,对植物的准确鉴定、遗传多样性研究以及可持续发展都具有重要的意义。今后应当加大对GBS技术的研究力度,对其进行改进使其能得到更好地应用,在技术层面: (1) 选定一种限制内切酶对基因组DNA进行酶切,只能识别特定的核苷酸序列,在一定水平上限制了测序的覆盖范畴,今后要对限制酶的选择以及内切酶的使用进行深入研究; (2) PCR反应次数过多,可能会引发非特异性扩增,以及接头二聚体可能过滤不完全,会在测序中造成一定损失,今后要对实验流程进行改进,减少实验中不必要的损失,影响实验结果。而在应用对象中,目前GBS应用的研究主要集中于禾本植物,如柳枝稷、燕麦等农作物均有涉猎,对药用植物也有研究,但相对较少,今后不能仅仅局限于农作物,也应当把GBS技术应用于药用植物,对药用植物进行深入研究。
参考文献
[1] 张琳.基于GBS技术构建牡丹高密度遗传连锁图谱[D].河南洛阳:河南科技大学, 硕士学位论文, 2016.Zhang L. Construction of high-density genetic linkage map based on GBS technique in tree peony[D]. Henan Luoyang:Henan University of Science and Technology, Master Dissertation, 2016.
[2] Harismendy O, Ng P C, Strausberg R L, et al.. Evaluation of next generation sequencing platforms for population targeted sequencing studies[J]. Genome Biol., 2009, 10 (3) :1-13.
[3] Elshire R J, Glaubitz J C, Sun Q, et al.. A robust, simple genotyping-by-sequencing (GBS) approach for high diversity species[J]. PLoS ONE, 2011, 6 (5) :e19379.
[4] Poland J A, Brown P J, Sorrells M E, et al.. Development of high-density genetic maps for barley and wheat using a novel two-enzyme genotyping-by-sequencing approach[J]. PLoS ONE, 2012, 7 (2) :e32253.
[5] Sonah H, Bastien M, Iquira E, et al.. An improved genotyping by sequencing (GBS) approach offering increased versatility and efficiency of SNP discovery and genotyping[J]. PLoS ONE, 2013, 8 (1) :e54603.
[6] Narum S R, Buerkle C A, Davey J W, et al.. Genotyping-bysequencing in ecological and conservation genomics[J]. Mol.Ecol., 2013, 22 (11) :2841-2847.
[7] Beissinger T M, Hirsch C N, Sekhon R S, et al.. Marker density and read depth for genotyping populations using genotyping-by-sequencing[J]. Genetics, 2013, 193 (4) :1073-1081.
[8] Spindel J, Wright M, Chen C, et al.. Bridging the genotyping gap:Using genotyping by sequencing (GBS) to add highdensity SNP markers and new value to traditional bi-parental mapping and breeding populations[J]. Theor. Appl. Genetics, 2013, 126 (11) :2699-2716.
[9] De M D, Peters S O, Mitchell S E, et al.. Genotyping-bysequencing (GBS) :A novel, efficient and cost-effective genotyping method for cattle using next-generation sequencing[J]. PLoS ONE, 2013, 8 (5) :e62137.
[10] Crossa J, Beyene Y, Kassa S, et al.. Genomic prediction in maize breeding populations with genotyping-by-sequencing[J].G3, 2013, 3 (11) :1903-1926.
[11]李翔, 范成明, 卢龙斗, 等.甘蓝型油菜遗传图谱构建及重要性状遗传解析的研究进展[J].分子植物育种, 2016, 14 (1) :98-116.Li X, Fan C M, Lu L D, et al.. Research on construction of genetic maps and dissection of important traits in Brassica napus[J]. Mol. Plant Breed., 2016, 14 (1) :98-116.
[12]秦伟伟, 李永祥, 李春辉, 等.基于高密度遗传图谱的玉米籽粒性状QTL定位[J].作物学报, 2015, 41 (10) :1510-1518.Qin W W, Lin Y X, Lin C H, et al.. QTL mapping for kernel related traits based on a high-density genetic map[J]. Acta Agron. Sin., 2015, 41 (10) :1510-1518.
[13]罗昕.基于下一代测序的玉米高通量SNP开发及关联分析[D].武汉:华中农业大学, 硕士学位论文, 2015.Luo X. Development and correlation analysis of maize high throughput SNP based on next-generation sequencing[D].Wuhan:Huazhong Agricultural University, Master Dissertation, 2015.
[14] Sonah H, Bastien M, Iquira E, et al.. An improved genotyping by sequencing (GBS) approach offering increased versatility and efficiency of SNP discovery and genotyping[J]. PLoS ONE, 2013, 8 (1) :e54603.
[15] Gardner K M, Brown P, Cooke T F, et al.. Fast and costeffective genetic mapping in apple using next-generation sequencing.[J]. G3-Genes Genomes Genetics, 2014, 4 (9) :1681-1687.
[16]李春辉.玉米高密度重组图谱构建及耐旱相关性状的遗传解析[D].北京中国农业科学院, 博士学位论文, 2015.Lin C H. Construction of high density recombination map and genetic analysis of drought tolerance traits in maize[D].Beijing:Chinese Academy of Agricultural Sciences, Doctor Dissertation, 2015.
[17] Li H, Vikram P, Singh R P, et al.. A high density GBS map of bread wheat and its application for dissecting complex disease resistance traits[J]. BMC Genomics, 16, 1 (2015-03-19) , 2015, 16 (1) :216.
[18] Liu C, Zhu S, Tang S, et al.. QTL analysis of four main stem bark traits using a GBS-SNP-based high-density genetic map in ramie[J]. Sci. Rep., 2017, 7:13458.
[19] Edae E A, Olivera P D, Jin Y, et al.. Genotype-by-sequencing facilitates genetic mapping of a stem rust resistance locus in Aegilops umbellulata, a wild relative of cultivated wheat[J].BMC Genomics, 2016, 17 (1) :1039
[20] Heffelfinger C, Fragoso C A, Moreno M A, et al.. Flexible and scalable genotyping-by-sequencing strategies for population studies[J]. BMC Genomics, 2014, 15 (1) :979.
[21] Huang Y F, Poland J A, Wight C P, et al.. Using genotypingby-sequencing (GBS) for genomic discovery in cultivated oat[J]. PLoS ONE, 2014, 9 (7) :e102448.
[22] Wong M M L, Gujariaverma N, Ramsay L, et al..Classification and characterization of species within the genus Lens using genotyping-by-sequencing (GBS) [J]. PLoS ONE, 2015, 10 (3) :e0122025.
[23]沈登锋.不同海拔藏川杨遗传多样性评估及苗期表型关联分析[D].北京:北京林业大学, 博士学位论文, 2014.Shen D F. Genetic diversity analysis of Tibetan poplar (Populus szechunanica var. Tibetica) population collected from different altitudes and phenotype of seedlings association analysis[D].Beijing:Beijing Forestry University, Doctor Dissertation, 2014.
[24]黄之涛.基于Bin map高密度连锁图谱定位玉米容重及相关性状的QTL[D].四川雅安:四川农业大学, 硕士学位论文, 2014.Huang Z T. QTL analysis for maize test weight related traits using the high density linkage map Bin map[D]. Sichuan Yaan:Sichuan Agricultural University, Master Dissertation, 2014.
[25]赖国荣, 张静, 刘函, 等.基于GBS构建玉米高密度遗传图谱及营养品质性状QTL定位[J].农业生物技术学报, 2017, 25 (9) :1400-1410.Lai G R, Zhang J, Liu H, et al.. Construction of high density genetic map via GBS technology and QTL mapping for nutritional auality traits in maize (Zea mays) [J]. J. Agric.Biotechnol., 2017, 25 (9) :1400-1410.
[26]牛璐瑶.一个人工合成玉米群体的基因组变异分析[D].武汉:华中农业大学, 硕士学位论文, 2016.Niu L Y. Genomic variation of a diverse synthetic population in maize[D]. Wuhan:Huazhong Agricultural University, Master Dissertation, 2016.
[27]王明.玉米抗丝黑穗病的全基因组关联分析[D].武汉:华中农业大学, 博士学位论文, 2012.Wang M. Genome-wide association study (GWAS) of resistance to head smut in maize (Zea mays L.) [D]. Wuhan:Huazhong Agricultural University, Doctor Dissertation, 2012.
[28] Torkamaneh D, Laroche J, Belzile F. Genome-wide SNP calling from genotyping by sequencing (GBS) data:A comparison of seven pipelines and two sequencing technologies[J]. PLoS ONE, 2016, 11 (8) :e0161333.
[29] Faville M J, Ganesh S, Cao M, et al.. Predictive ability of genomic selection models in a multi-population perennial ryegrass training set using genotyping-by-sequencing[J].Theor. Appl. Genetics, 2018, 131 (3) :703-720.
[30] Ward J A, Jasbir B, Felicidad F F, et al.. Saturated linkage map construction inRubus idaeususing genotyping by sequencing and genome-independent imputation[J]. BMC Genomics, 2013, 14 (1) :2.
[31] Rocher S, Jean M, Castonguay Y, et al.. Validation of genotyping-by-sequencing analysis in populations of tetraploid alfalfa by 454 sequencing[J]. PLoS ONE, 2015, 10 (6) :e0131918.
[32] Liu H, Bayer M, Druka A, et al.. An evaluation of genotyping by sequencing (GBS) to map the Breviaristatum-e (ari-e) locus in cultivated barley[J]. BMC Genomics, 2014, 15 (1) :104-104.
[33] Yu L X, Zheng P, Zhang T, et al.. Genotyping-by-sequencing based genome-wide association studies on Verticillium wilt resistance in autotetraploid alfalfa (Medicago sativa L.) [J].Mol.Plant Pathol., 2016, 18 (2) :187-194.
[34] Reasor E H, Brosnan J T, Staton M E, et al.. Genotypic and phenotypic evaluation of off-type grasses in hybrid Bermudagrass[Cynodon dactylon (L.) Pers.×C.transvaalensis Burtt-Davy] putting greens using genotyping-bysequencing and morphological characterization:[J]. Hereditas, 2018, 155 (1) :8.
[35] Arbizu C I, Ellison S L, Senalik D, et al.. Genotyping-bysequencing provides the discriminating power to investigate the subspecies of Daucus carota (Apiaceae) [J]. BMC Evol.Biol., 2016, 16 (1) :234.
[36] Taranto F, D'Agostino N, Greco B, et al.. Genome-wide SNP discovery and population structure analysis in pepper (Capsicum annuum) using genotyping by sequencing[J].BMC Genomics, 2016, 17 (1) :943.
[37] Schafleitner R, Huang S M, Chu S H, et al.. Identification of single nucleotide polymorphism markers associated with resistance to bruchids (Callosobruchus spp.) in wild mungbean (Vigna radiata var. sublobata) and cultivated V. radiata, through genotyping by sequencing and quantitative trait lo cus analysis[J]. BMC Plant Biol., 2016, 16 (1) :159.
[38] Pavan S, Marcotrigiano A R, Ciani E, et al.. Genotyping-bysequencing of a melon (Cucumis melo L.) germplasm collection from a secondary center of diversity highlights patterns of genetic variation and genomic features of different gene pools[J]. BMC Genomics, 2017, 18 (1) :59.
[39] Montero-Pau J, Blanca J, Esteras C, et al.. An SNP-based saturated genetic map and QTL analysis of fruit-related traits in Zucchini using genotyping-by-sequencing[J]. BMC Genomics, 2017, 18 (1) :94.
[40] Otto L G, Mondal P, Brassac J, et al.. Use of genotyping-bysequencing to determine the genetic structure in the medicinal plant chamomile, and to identify flowering time and alphabisabolol associated SNP-loci by genome-wide association mapping[J]. BMC Genomics, 2017, 18 (1) :599.
[41] Pucher A, Hash C T, Wallace J G, et al.. Mapping a malefertility restoration locus for the A4 cytoplasmic-genic malesterility system in pearl millet using a genotyping-bysequencing-based linkage map[J]. BMC Plant Biol., 2018, 18 (1) :65.
[42]王小柯, 江东, 孙珍珠.利用GBS技术研究240份宽皮柑橘的系统演化[J].中国农业科学, 2017, 50 (9) :1666-1673.Wang X K, Jiang D, Sun Z Z. Study on phylogeny of 240mandarin accessions with genotyping-by-sequencing technology[J]. Sci. Agric. Sin., 2017, 50 (9) :1666-1673.
[43] Fu Y B, Cheng B, Peterson G W. Genetic diversity analysis of yellow mustard (Sinapis alba L.) germplasm based on genotyping by sequencing[J]. Genetic Resour. Crop Evol., 2014, 61 (3) :579-594.
[44] Yang H, Ye T, Zheng Z, et al.. Application of next-generation sequencing for rapid marker development in molecular plant breeding:A case study on anthracnose disease resistance in Lupinus angustifolius L.[J]. BMC Genomics, 2012, 13 (1) :318.
[45] Giovanny C P, Luis D G, Brandon S, et al.. Exploiting genotyping by sequencing to characterize the genomic structure of the American cranberry through high-density linkage mapping[J]. BMC Genomics, 2016, 17 (1) :451.
[46] Sch?nhals E M, Ding J, Ritter E, et al.. Physical mapping of QTL for tuber yield, starch content and starch yield in tetraploid potato (Solanum tuberosum L.) by means of genome wide genotyping by sequencing and the 8. 3 K Sol CAP SNP array[J]. BMC Genomics, 2017, 18 (1) :642.
[47] Pootakham W, Sonthirod C, Naktang C, et al.. Effects of methylation-sensitive enzymes on the enrichment of genic SNPs and the degree of genome complexity reduction in a two-enzyme genotyping-by-sequencing (GBS) approach:A case study in oil palm (Elaeis guineensis) [J]. Mol. Breed., 2016, 36 (11) :154.
[48] Romay M C, Millard M J, Glaubitz J C, et al.. Comprehensive genotyping of the USA national maize inbred seed bank[J].Genome Biol., 2013, 14 (6) :R55.
[49] Cooke T F, Yee M C, Muzzio M, et al.. GBStools:A statistical method for estimating allelic dropout in reduced representation sequencing data[J]. PLoS Genetics, 2016, 12 (2) :e1005631.
[50] Wang Y H, Upadhyaya H D, Burrell A M, et al.. Genetic structure and linkage disequilibrium in a diverse, representative collection of the C4 model plant, Sorghum bicolor[J]. G3-Gene Genomes Genetics, 2013, 3 (5) :783-793.
[51] Edae E A, Olivera P D, Jin Y, et al.. Genotype-by-sequencing facilitates genetic mapping of a stem rust resistance locus in Aegilops umbellulata, a wild relative of cultivated wheat[J].BMC Genomics, 2016, 17 (1) :1039.
[52]毛晓微.印度南瓜主要园艺性状的QTL定位[D].河北邯郸:河北工程大学, 硕士学位论文, 2016.Mao X W. QTL mapping of main agronomic traits in pumpkin[D]. Hebei Handan:Hebei University of Engineering, Master Dissertion, 2016.
[53] Nimmakayala P, Levi A, Abburi L, et al.. Single nucleotide polymorphisms generated by genotyping by sequencing to characterize genome-wide diversity, linkage disequilibrium, and selective sweeps in cultivated watermelon[J]. BMC Genomics, 2014, 15 (1) :767.
[54] Sim S C, Van A D, Stoffel K, et al.. High-density SNP genotyping of tomato (Solanum lycopersicum L.) reveals patterns of genetic variation due to breeding[J]. PLoS ONE, 2012, 7 (9) :e45520.
[55] Boutet G, Alves C S, Falque M, et al.. SNP discovery and genetic mapping using genotyping by sequencing of whole genome genomic DNA from a pea RIL population[J]. BMC Genomics, 2016, 17 (1) :121.
[56]熊海铮.豇豆遗传多样性及若干农艺性状关联分析[D].杭州:浙江大学, 博士学位论文, 2016.Xiong H Z. Genetic diversity and association analysis of agronomic traits in Cowpea[D]. Hangzhou:Zhejiang University, Doctor Dissertation, 2016.
[57] Lu F, Lipka A E, Glaubitz J, et al.. Switchgrass genomic diversity, ploidy, and evolution:Novel insights from a network-based SNP discovery protocol[J]. PLoS Genetics, 2013, 9 (1) :e1003215.
[58] Lin M, Cai S, Wang S, et al.. Genotypingbysequencing (GBS) identifed SNP tightly linked to QTL for preharvest sprouting resistance[J]. Theor. Appl. Genet., 2015, 128 (7) :1385-1395.
[59] Prochnik S, Marri P R, Desany B, et al.. The cassava genome:Current progress, future directions[J]. Tropical Plant Biol., 2012, 5 (1) :88-94.
[60] Anja B, Jochen H, Bruno H, et al.. High-throughput polymorphism detection and genotyping in Brassica napususing next-generation RAD sequencing[J]. BMC Genomics, 2012, 13 (1) :281.
点击查看>>植物生物技术论文(精选范文8篇)其他文章