摘要:采用结合转录组、代谢通路、蛋白结构的呼出气体检测生物信息学分析方法来确定肺癌气体标志物,用于肺癌的筛选诊断.采用标准仪器(GCMS)检测肺癌病人和正常人的呼吸气体样本;经统计分析,筛选出10种特异性挥发性有机物(VOC).采用转录组分析得到肺癌和健康人的差异表达基因,其富集的代谢通路与人体内产生VOC的代谢通路一致,证明所筛选的VOC标志物与肺癌病人代谢具有相关性.基于此VOC建立的肺癌诊断模型的灵敏度、特异性和整体正确率分别为86.2%,91.2%和89.6%,说明所提方法能简便、有效区分正常人和肺癌病人,为早期肺癌筛查提供方便、可靠的检测方法.
关键词:呼出气体检测; 肺癌标志物; 生物信息学; 转录组分析; 蛋白结构分析; 肺癌早期筛查;
Screening and bioinformatics analysis of lung cancer exhale breath biomarkers
WU Qian WANG Ping
Key Laboratory for Biomedical Engineering of Education Ministry, Zhejiang University
Abstract:The exhale breath detection combined bioinformatics analysis method, including transcriptome, metabolic pathway and protein structure, was proposed to identify gas markers for screening and diagnosis of lung cancer.Lung cancer patients and healthy controls' samples were collected to performe GC-MS and ROC curve analysis which obtained ten specific VOCs. Differentially expressed genes were obtained by transcriptome analysis. The differentially expressed genes and relative metabolic pathways were consistent with in vivo biological process,which meant that these VOCs come from the metabolism of lung cancer patient. The sensitivity, specificity and overall accuracy of lung cancer diagnosis model established based on VOCs were 86.2%, 91.2% and 89.6%,respectively. Thus, the proposed method can distinguish normal people and lung cancer patients simply and effectively, providing convenient approach for early screening of lung cancer.
在世界范围内,肺癌的发病率和死亡率都在各类癌症中居首位,但医学界一直缺少一种简便的早期肺癌筛查方法.人类的呼出气体中包含大量人体健康状态的信息,其中挥发性有机物(volatile organic compound,VOC)被认为是一种可用于无创肺癌筛查的理想标志物[1].在过去的几十年里,关于肺癌病人呼出气中VOC的研究已有大量的报道[1,2].基于不同的统计方法,不同的研究提出了各自的肺癌特异性VOC标志物[3].由于关于呼气中的VOC与肺癌在生理病理学上的联系鲜有报道,肺癌病人代谢的特异性VOC一直难以确定.
癌症组织中发生基因突变或是基因表达量的变化已是科学界内普遍的共识,因此,从基因层面入手研究呼出气体中的VOC与肺癌之间的关系很有前景.目前,很多研究在美国癌症基因组数据库(TCGA)上进行数据挖掘,已取得丰硕成果[4].特别是转录组数据分析发现了大量癌症标志性基因变异位点[5,6],这些基因靶点对相关癌症的靶向治疗具有指导意义.由于癌症是多基因指导的复杂疾病,基因表达下游的通路分析也是必不可少的,这在认识癌症的发生、发展上起着至关重要的作用[7].
将呼出气体检测与转录组、代谢通路、蛋白结构分析这些生物信息学方法相结合,可以筛选出可靠的肺癌特异性VOC.这有利于确定肺癌病人呼出气体标志物以及通过检测呼出气体实现肺癌病人的大规模早期筛查.
1 材料和方法
1.1 呼出气体标本的采集和标准仪器分析
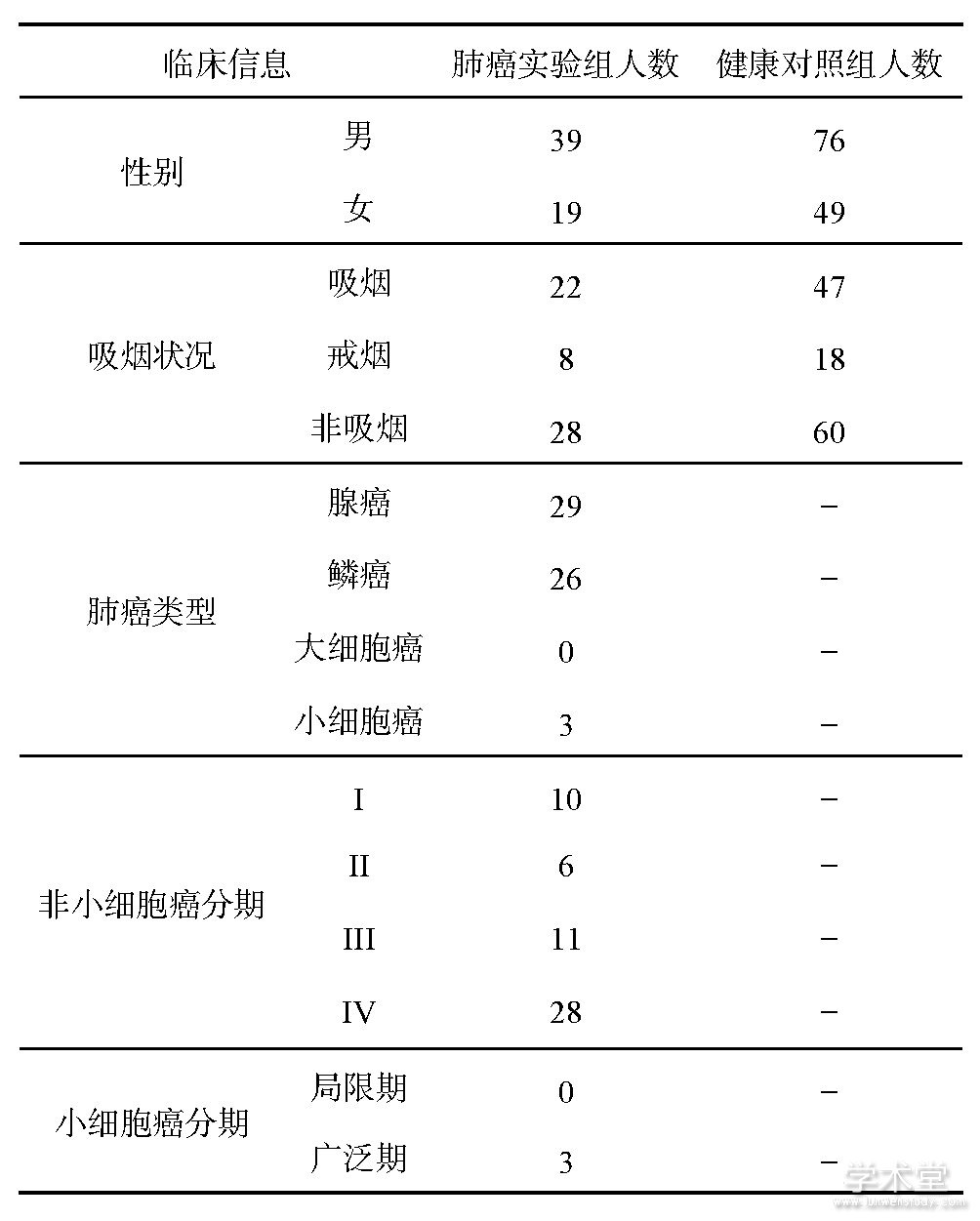
本课题组长期与浙江大学医学院附属邵逸夫医院的呼吸科合作,收集肺癌病人呼出气体,前期研究已经积累了大量样本;健康人的呼出气体收集于浙江大学生仪学院和邵逸夫医院,所有受试者的统计数据见表1,其中,肺癌实验组58人(平均年龄为59.0),健康对照组125人(平均年龄为56.1).采集呼出气体前,每个受试者都签署了知情同意书(邵逸夫医院伦理委员会,No.20070525、ChiCTR-DCD-15007106).人体呼出气体样本由自主研制的采气仪收集,样本采集过程和分析参见之前的研究[8].呼气样本使用气相色谱-质谱联用仪(GC-MS QP2010 Plus,购于日本Shimadzu公司)进行成分分离和定性检测(见图1).
1.2 肺癌特异性VOC筛选
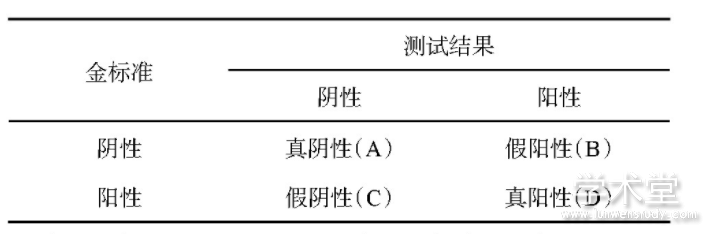
受试者工作特征(receiver operator characteristic,ROC)曲线是以灵敏度为Y轴,1-特异度为X轴的曲线图,是一种评估诊断模型性能的有效方法,特别适用于诊断结果非0即1的二分类问题.其中二分类逻辑回归问题的特异度与灵敏度计算方式如表2所示.通过绘制ROC曲线,可以获得任意阈值对疾病的识别能力,从中选出最优的诊断阈值.一般而言,ROC曲线越靠近左上角,阈值点诊断效果越好.因为ROC曲线的横轴是1-特异度,纵轴是灵敏度,据此可知在该点处的二分类法的特异度和灵敏度最高,也就是说出错率最低.ROC曲线的曲线下面积(area under curve,AUC)是评价模型诊断能力最常用的指标,AUC取值在0~1.0,越接近1.0,诊断模型的整体性能越好[9].
表1 受试者基本临床信息统计
图1 受试者呼吸气体的采集和分析
表2 二分类逻辑回归计算方法
注:假阳性率=B/(A+B),特异度=1-假阳性率;真阳性率=D/(C+D),灵敏度=真阳性率
1.3 转录组数据分析
肺癌病人和健康人的转录组数据从美国癌症基因组数据库TCGA下载(更新时间为2018年4月).采用R语言中的edgeR包和DESeq包来筛选差异表达基因.R语言是用于统计分析、绘图的优秀工具和操作环境,其中的edgeR包[10]是基于负二项分布来分析不同生物样本中RNA测序谱的经典方法,用到包括经验贝叶斯估计和广义线性模型等算法.而DESeq包[11]根据动态数据的范围,对差异表达基因的选择更平衡,对只有较少重复的样本实验有更好的表现.两者最重要的差别就是:edgeR根据分散平均关系,将特征水平的分散估计趋向于一个趋势平均值,而DESeq则取个体分散估计和分散平均趋势的最大值[12].差异表达基因的评判用到了错误发现率(false discovery rate,FDR)和差异倍数(fold-change,FD)两个指标.本研究以正常人转录组数据为对照组,将肺癌转录组中符合FDR<0.001且FD>4以上的基因定义为差异表达基因.
1.4 代谢通路分析
为了阐明差异表达基因在人体内参与的信号通路和发挥的功能,根据得到的差异表达基因,使用KOBAS 3.0[13]进行分析,对靶基因进行KEGG(Kyoto encyclopedia of genes and genomes)通路分析和基因本体论(gene ontology,GO)注释分析.由此,得到这些基因的功能和相关的代谢通路;查询文献以及人类代谢数据库(HMDB)、KEGG等数据库中产生肺癌标志性VOC的代谢通路;比较差异表达基因与人体内代谢通路的匹配情况.
1.5 蛋白结构分析
在蛋白结构数据库Protein Data Bank中下载蛋白质三维结构文件并使用Pymol软件以棍棒或是卡通风格实现三维结构的图形化.使用UCSF Chimera软件分析突变蛋白质中氨基酸残基的空间结构变化.在基因突变后,氨基酸残基的不同旋转异构体(rotamer)都可能产生原子碰撞,本文选择最有可能的5种rotamers并作能量最小化处理来稳定结构.
2 结果与讨论
2.1 筛选和验证肺癌特异性挥发性有机物
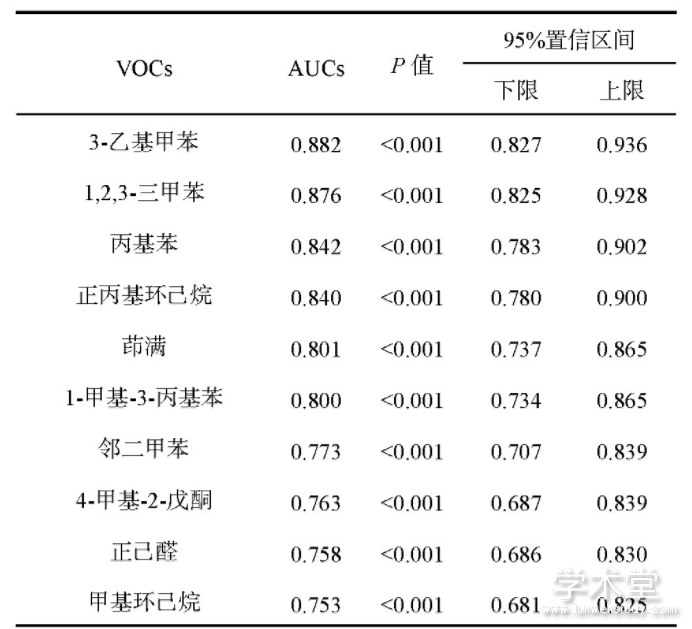
从2008年4月—2017年12月采集的样本中,选取183个已经完成GC-MS分析并且结果较好的呼吸样本.按照临床组织病理学诊断结果,将所有呼气样本分为肺癌组58例样本和健康组125例样本.利用GC-MS分析,得到174种样本中普遍存在的VOC.使用二分类逻辑回归算法绘制这174种VOC的ROC曲线,并将曲线下面积(AUC)最大的10个VOC列于表3.这些AUC都大于0.75,表明所提方法对肺癌组和健康组有很好的区分能力,且在区分肺癌组和健康组的差异上具有显着性(P<0.001).表3还展示了95%置信区间(confidence interval,CI)的上限和下限数值,结果显示这些曲线下面积都偏移不大.
表3 肺癌特异性挥发性有机物(VOC)的分析结果
为了验证这10个VOC对肺癌的判别能力,使用二元逻辑回归分析建立一个肺癌诊断模型,得到的灵敏度、特异性和整体正确率分别为86.2%,91.2%和89.6%.这表明,选取的这10个VOC可以很好地区分肺癌病人和正常人,具有显着的肺癌特异性.但已有报道称,VOC对于肺癌早期病人和晚起病人的区分还不明显[2];有研究表明对于不同类型的肺癌,8-己基十五烷、3,7-二甲基十五烷和2,6,10,14-四甲基十五烷可以作为区分腺癌和鳞癌的标志物,但是其AUC都只有0.65左右,因此区分度不是很好[14].
2.2 筛选差异表达基因
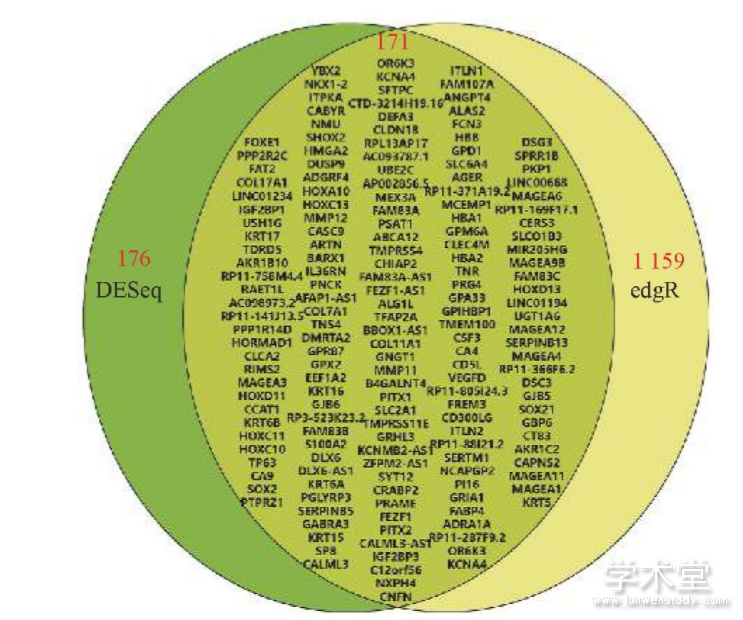
从TCGA数据库中下载经标准化处理的347个肺癌转录组样本和22个健康人转录组样本,使用edgeR计算得到1 159个差异基因,其中在肺癌样本在表达量上调的基因(1 106个)大致是下调基因(53个)的4倍;在同样的阈值下(FDR<0.001且FD>4),使用DESeq计算得到176个差异基因,其中表达量上调的基因为128个,下调的基因为48个.采用2种不同的计算方法得到的目标基因差了6倍之多,但使用DESeq计算得到的差异基因在用edgeR计算时都能找到,说明DESeq用了更为严苛的筛选条件.一般情况下,研究者为了能准确地预测,结合2种方法的优势,综合2种方法的结果,并将同时出现的差异表达基因作为最终候选靶基因用于后续分析,使用韦恩图的形式展示这171个基因,如图2所示.这些基因中表达量下调最低的是ITLN1基因,只有正常水平的0.003 4,上调最多的是MAGEA1基因,表达量是正常水平的1 241倍.这说明在癌症组织中存在着大量表达异常的基因,而且这些基因经转录翻译后产生的蛋白质在癌症的发生、发展中可能起到重要作用.不同于前面的呼出气体样本,TCGA的转录组数据来源于世界各地,而且样本量大,据此分析得到的差异表达基因能够代表人类的普遍情况.
图2 由edgeR和DESeq分析得到共有差异表达基因的韦恩图
2.3 差异表达基因的富集分析
GO通过对基因及其产物在细胞中参与的生物学过程、组成成分以及分子功能对基因及产物进行描述.对这些基因分类和功能注释的结果显示,171个差异表达基因主要发挥30项生物过程的功能.前5个分别为糖脂化作用、脂肪酸代谢、基因沉默、端粒形成和黄酮类的合成.另外还有与癌症密切相关的DNA修复、磷酸化作用和免疫反应等.在这些基因中,与核小体相关的基因最多,达30个,其次是RNA干扰(13个)和组蛋白结合(13个).在代谢通路方面,差异表达基因参与了酒精代谢通路、自身免疫反应、甾类激素合成、尼古丁代谢、细胞色素P450(Cytochrome p450,CYP)介导的外源物质代谢等,其中有与吸烟肺癌病人吸烟密切相关的尼古丁代谢.
2.4 人体内VOC产生通路
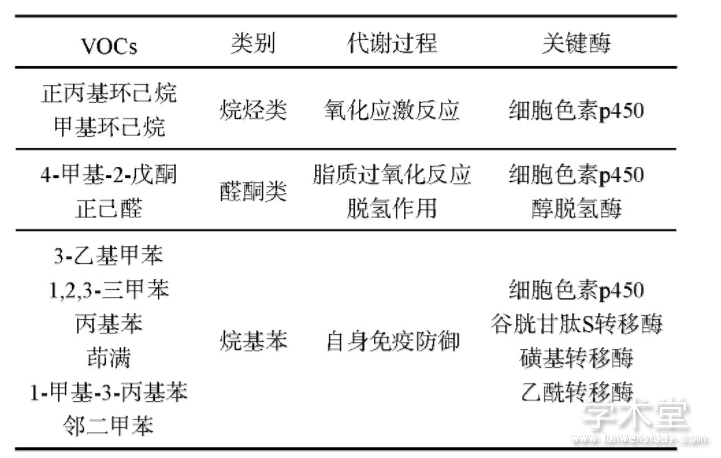
从肺癌病人呼出气体中检测到的VOC是来自于人体内的新陈代谢.由于与肺癌相关的基因发生突变或是表达量改变,且挥发性有机物具有不溶于血液的特点,痕量的VOC经过肺泡的气体交换,可在呼出气体中被检测出来[15].前文中10个经过ROC曲线筛选出来的VOC按官能团的差别可以分为3类(见表4),在人体内的生物过程大致如下文所述.
表4 人体内产生VOC的代谢通路和关键酶
2.4.1 烷烃类
这类VOC主要由多不饱和脂肪酸的氧化应激反应产生.脂质的氧化降解通过自由基链式反应机制,会对细胞造成伤害,其中的活性氧(reactive oxygen species,ROS)与体内生物大分子(如磷脂、酶和核酸等)发生脂质过氧化反应,形成丙二醛等代谢产物,从而改变细胞膜的流动性和通透性.戊烷和乙烷作为脂质过氧化作用的指标,由于其灵敏性和非侵入性已被广泛应用.在肺癌病人中,催化有机物氧化的CYP蛋白会通过羟基化产生一些烷烃类VOC[16].
2.4.2 醛酮类
这类VOC主要由CYP介导的脂质β氧化产生[17],经过活化的脂酰CoA在线粒体基质中经过脱氢、加水、再脱氢和硫解4步反应,生成一分子乙酰CoA和一个少2个碳的新的脂酰CoA,长链脂酰CoA可再次进行β氧化,也可进入其他代谢通路;另外,醇类的脱氢反应也会通过消耗NAD+生成NADH而产生醛酮类VOC.细胞摄取的烷烃进入微粒体,在细胞色素P245和NADPH2细胞色素P245还原酶催化作用下生成脂肪醇,然后由长链脂肪醇脱氢酶(或醇氧化酶)和醛脱氢酶催化依次生成脂肪醛和脂肪酸.2.4.3烷基苯这类VOC主要由外源物质的代谢造成,如:烟草、酒精、其他污染物等.这些分子渗透过细胞膜对蛋白、脂肪酸合DNA造成过氧化损伤.大部分肺癌病人有吸烟史,有研究表明,吸烟者的呼气中甲苯的含量明显高于一般人[18].另外人体内的自身免疫防御机制也会通过谷胱甘肽S转移酶、磺基转移酶和乙酰转移酶消除外源化合物[19].
2.5 验证VOC产生通路
表4中代谢通路的关键酶都是由一个或多个基因编码的蛋白质.在肺癌病人的转录组分析中,这些基因的表达量都有不同程度的改变.例如,ADH7是编码醇脱氢酶的基因,在醇代谢的第一步中发挥重要作用.同时,在转录组分析中发现,ADH7的表达量是明显上调的(log CPM=4.4),这样肺癌病人体内将会表达更多醇脱氢酶,导致发生更多脱氢作用,产生烷烃类的VOC,并且VOC通过呼吸气体排放出来.同样地,编码谷胱甘肽S转移酶、磺基转移酶和乙酰转移酶的基因(SULT4A1、GSTA8P、GSTA9P、NAA11和NAALADL2-AS2)也都显着性上调,其log CPM分别为4.6、6.0、5.6、6.0和5.2.这意味着肺癌病人的呼出气中烷苯类的VOC也会比正常人更多.之前筛选出的10个VOC中有6个属于烷苯类,说明这是一类较为常见的肺癌特异性VOC.
CYP是由一个基因家族(CYP1A2、CYP24A1、CYP26A1、CYP4F11、CYP4F3和CYP2AB1P)编码的蛋白超家族,具有多种蛋白质亚型.根据转录组分析结果可知,除了CYP1A2基因是下调的(log CPM=-4.4),而其他5个基因都是上调的.CYP参与催化多种物质的氧化代谢反应,像甾体、脂肪酸和外源性物质等[20].例如,CYP26A1参与视黄酸的代谢;CYP4F11通过omega羟基化在维生素K的分解过程中发挥重要作用[21];CYP4F3是催化白三烯B4的关键酶[22].烷烃类、醛酮类和烷基苯的VOC都可以通过这些反应产生.下调的CYP1A2基因在催化黄曲霉毒素B1和乙酰胺酚的分解代谢中发挥重要作用.由于其表达量的下调,代谢底物会在体内累积或是转到其他可能产生VOC的代谢通路.
2.6 基因突变对VOC的影响
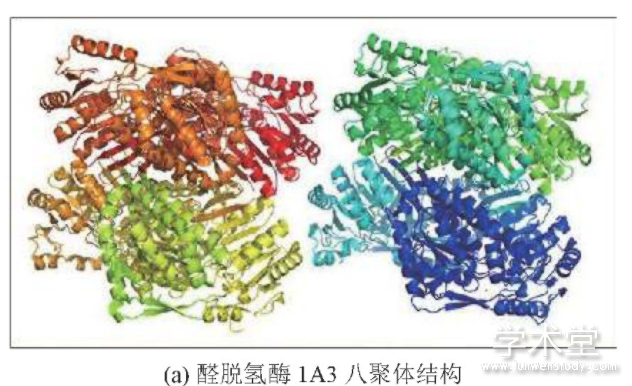
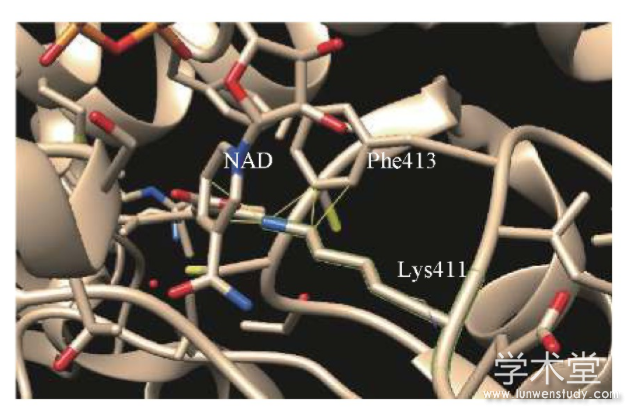
除了基因表达量上的差异,通路上关键酶的基因突变也是影响代谢产物变化的关键因素.醛脱氢酶(aldehyde dehydrogenase,ALDH)可以催化不可逆氧化反应,将醛生成羧酸盐.许多研究发现,ALDH基因的改变是肺癌的一个特异性标志物[23].除了基因表达调节失常,ALDH的基因突变会导致醛脱氢酶活性下降,从而减少羧酸的产出.以一个在ALDH1A3蛋白上的自然突变(p.E411K[24])为例,分析其在蛋白结构层面对酶活性的影响.本文使用的蛋白结构文件ID为5fhz,为人类醛脱氢酶1A3蛋白结合NAD和视黄酸的复合体,分辨率为2.9?.使用Pymol软件对ALDH1A3蛋白以卡通和棍棒模式图形化展示.该酶在人体内是以八聚体的形态发挥作用(见图3(a)),其活性中心主要由NAD结合411位的谷氨酸构成(见图3(b)).
图3 醛脱氢酶1A3的蛋白结构
PolyPhen-2是一款最常用于预测基因突变所造成蛋白结构和功能影响的软件,给出数值0~1.0来表示突变的严重程度,分数越高则表示影响越大[25].软件对突变p.E411K进行预测,结果给出0.956的得分,表示该突变会对蛋白造成比较严重的影响.突变p.E411K对ALDH1A3蛋白空间结构的影响使用UCSF Chimera软件分析,当411位的谷氨酸突变成赖氨酸时,由于赖氨酸的残基相对较长,侵入了原本NAD和413位苯丙氨酸的空间.Chimera软件预测结果显示,突变的赖氨酸会与周围产生5个原子碰撞(见图4).当出现这类结构变化,蛋白本身结构就会变得不稳定;而且411位的谷氨酸是NAD的结合位点[26],这会影响酶蛋白与底物的结合能力,最终表现为酶活力下降.因此,ALDH1A基因突变的肺癌病人对醛类物质的代谢能力就会下降,从而导致底物在体内积聚.经过一定时间的积累后,醛类VOC就可能在呼出气体中被检测到.
图4 突变后的醛脱氢酶1A3在411位赖氨酸与NAD和413位苯丙氨酸发生空间碰撞
3 结语
本文采用呼出气体检测仪器分析方法筛选出了10种在人群中区分肺癌病人和正常人的特异性挥发气体标志物.运用生物信息学统计方法分析了美国癌症基因组数据库的转录组数据,筛选出肺癌病人的差异基因,并通过富集分析算法确定了这些差异基因的代谢通路.进一步通过与人体内产生VOC的国际标准数据库比较,阐明了肺癌病人呼出气体中特异性VOC来源的可能机制,初步证明了VOC标志物与肺癌病人的关联性.这项创新性的研究为确定呼出气体中的肺癌标志物提供了生物学支持,也为通过呼出气体特异性标志物检测的早期筛查肺癌病人提供了有利的生理学证据.本文未对不同类型的肺癌以及不同分期的病人进行转录组数据上的区分,若要精准了解人体内VOC的代谢途径,还须进一步研究.
参考文献
[1]CHEN X, XU F, WANG Y, et al. A study of the volatile organic compounds exhaled by lung cancer cells in vitro for breath diagnosis[J]. Cancer, 2007,110(4):835–844.
[2]WANG Y, HU Y, WANG D, et al. The analysis of volatile organic compounds biomarkers for lung cancer in exhaled breath, tissues and cell lines[J].Cancer Biomarkers, 2012, 11(4):129–137.
[3]HAKIM M, BROZA Y Y, BARASH O, et al. Volatile organic compounds of lung cancer and possible biochemical pathways[J]. Chemical Reviews, 2012,112(11):5949–5966.
[4]TOMCZAK K, CZERWINSKA P, WIZNEROWICZ M. The cancer genome atlas(TCGA):an immeasurablesourceofknowledge[J].Contemporary Oncology, 2015, 19(1A):A68–77.
[5]LI T, KUNG H J, MACK P C, et al. Genotyping and genomic profiling of non-small-cell lung cancer:implications for current and future therapies[J].International Journal of Clinical Oncology, 2013,31(8):1039–1049.
[6]NETWORK CGAR. Comprehensive genomic characterization of squamous cell lung cancers[J].Nature, 2012, 489(7417):519–525.
[7]VOGELSTEIN B, PAPADOPOULOS N,VELCULESCU V E, et al. Cancer genome landscapes[J]. Science, 2013, 339(6127):1546–1558.
[8]ZOU Y, ZHANG X, CHEN X, et al. Optimization of volatile markers of lung cancer to exclude interferences of non-malignant disease[J]. Cancer Biomarkers, 2014, 14(5):371–379.
[9]PARK S H, GOO J M, JO C H. Receiver operating characteristic(ROC)curve:practical review for radiologists[J]. Korean Journal of Radiology,2004, 5(1):11–18.
[10]MCCARTHY D J, CHEN Y, SMYTH G K.Differential expression analysis of multifactor RNASeq experiments with respect to biological variation[J]. Nucleic Acids Research, 2012, 40(10):4288–4297.
[11]ANDERS S, HUBER W. Differential expression analysis for sequence count data[J]. Genome Biology, 2010, 11(10):R106–106.
[12]ANDERS S, MCCARTHY D J, CHEN Y, et al.Count-based differential expression analysis of RNA sequencing data using R and Bioconductor[J].Nature Protocols, 2013, 8(9):1765–1786.
[13]XIE C, MAO X, HUANG J, et al. KOBAS 2.0:a web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Research,2011, 39(2):W316–322.
[14]王怡珊.肺癌呼出气体标志物确定及电子鼻临床诊断方法研究[D].杭州:浙江大学, 2012.WANG Yi-shang. The research on lung cancer biomarkers in exhaled breath and clinical diagnostic method of e-nose[D]. Hangzhou:Zhejiang University, 2012.
[15]LOUREN?O C, TURNER C. Breath analysis in disease diagnosis:methodological considerations and applications[J]. Metabolites, 2014, 4(2):465–498.
[16]DE MONTELLANO PRO. Cytochrome P450:structure, mechanism, and biochemistry[M]. 3th ed. Berlin/Heidelberg:Springer Science and Business Medical, 2005.
[17]VAZ A, COON M J. Hydrocarbon formation in the reductive cleavage of hydroperoxides by cytochrome P-450[J]. Proceedings of the National Academy of Sciences, 1987, 84(5):1172–1176.
[18]KISCHKEL S, MIEKISCH W, SAWACKI A, et al.Breath biomarkers for lung cancer detection and assessment of smoking related effects-confounding variables, influence of normalization and statistical algorithms[J]. Clinica Chimica Acta, 2010,411(21/22):1637–1644.
[19]GUENGERICH F P, SHIMADA T. Oxidation of toxic and carcinogenic chemicals by human cytochrome P450 enzymes[J]. Chemical Research in Toxicology, 1991, 4(4):391–407.
[20]WHITE J A, BECKETT-JONES B, GUO Y D, et al.cDNA cloning of human retinoic acid-metabolizing enzyme(hP450RAI)identifies a novel family of cytochromes P450(CYP26)[J]. Journal of Biological Chemistry, 1997, 272(30):18538–18541.
[21]EDSON K Z, PRASAD B, UNADKAT J D, et al.Cytochrome P450-dependent catabolism of vitamin K:ω-hydroxylation catalyzed by human CYP4F2 and CYP4F11[J]. Biochemistry, 2013, 52(46):8276–8285.
[22]CHRISTMAS P, JONES J P, PATTEN C J, et al.Alternative splicing determines the function of CYP4F3 by switching substrate specificity[J].Journal of Biological Chemistry, 2001, 276(41):38166–381672.
[23]COROMINAS-FAJA B, OLIVERAS-FERRAROS C,CUY?S E, et al. Stem cell-like ALDHbright cellular states in EGFR-mutant non-small cell lung cancer:a novel mechanism of acquired resistance to erlotinib targetable with the natural polyphenol silibinin[J].Cell Cycle, 2013, 12(21):3390–3404.
[24]ABOUZEID H, FAVEZ T, SCHMID A, et al.Mutations in ALDH1A3 represent a frequent cause of microphthalmia/anophthalmia in consanguineous families[J]. Human Mutation, 2014, 35(8):949–953.
[25]ADZHUBEI I A, SCHMIDT S, PESHKIN L, et al. A method and server for predicting damaging missense mutations[J]. Nature Methods, 2010, 7(4):248–249.
[26]MORETTI A, LI J, DONINI S, et al. Crystal structure of human aldehyde dehydrogenase 1A3complexed with NAD(+)and retinoic acid[J].Scientific Reports, 2016, 6:35710.