摘 要: 杨絮是引起过敏性疾病的重要气传变应原之一。为了分析杨絮的蛋白组分并预测其T、B细胞表位,本研究采用有机溶媒法提取杨絮总蛋白,二喹啉甲酸(bicinchoninic acid,BCA)试剂盒检测蛋白浓度。胰酶酶解总蛋白,经高效液相色谱法(high performance liquid chromatography,HPLC)分级、液相色谱-质谱进行组分分析以及质谱质控鉴定;生物信息学方法在线预测不同组分中的T细胞和线性B细胞表位。结果显示,杨絮蛋白总浓度为5.53μg/μL,经酶解及液相色谱-质谱进行组分分析共获得22 023个肽段,质谱质控显示所获肽段的质量偏移(mass error)<10 ppm,肽段长度主要分布在8~20个氨基酸残基之间;生物信息学在线预测结果表明,所得肽段含16个高亲和力T细胞表位、2 300个中等亲和力的T细胞表位以及10个线性B细胞表位。该研究结果表明,杨絮的蛋白组分含有具有潜在诱发Ⅰ型过敏性疾病的T和B细胞表位。
关键词: 杨絮; 表位; 变应原; 过敏性疾病;
Abstract: Mature pollen from Populus deltoides is one of the important aeroallengens that can cause allergic diseases. To analyze the protein components of mature pollen and predict potential T and B cell epitopes,total protein in mature pollen samples from Populus deltoides were extracted by organic solvent method and protein concentration was determined using bicinchoninic acid(BCA) protein quantification assay. Total protein was digested with trypsin and fractionated by high performance liquid chromatography(HPLC) before analyzed using liquid chromatography-mass spectrometry. T cell and linear B cell epitopes in the different components were predicted by bioinformatics. The results showed that the total concentration of poplar protein was 5.53 μg/μL. A total of 22 023 peptides were obtained by enzymatic hydrolysis and liquid chromatography-mass spectrometry. Mass spectrometry showed that the mass error of the obtained peptide was lower than 10 ppm. The peptide lengths were mainly distributed between 8 and 20 amino acid residues. Bioinformatics analysis indicated that these peptides contained 16 high-affinity T cell epitopes, 2 300 medium-affinity T cell epitopes, and 10 linear B cell epitopes. In summary, the protein components of Populus deltoides contains T and B cell epitopes that could potentially induce type I allergic diseases.
Keyword: Populus deltoides mature pollen; epitope; allergen; allergic diseases;
杨树是在我国华北及华东地区广泛种植的树种,具有生长速度快、成活率高等特点,被广泛应用于城市绿化和防风(沙)护堤。随着种植面积的增加,由此带来的环境问题受到越来越多的关注,尤其是每年4至5月份,漫天飞舞的杨絮给人们的生活带来诸多困扰[1,2]。杨絮引起过敏性疾病的新闻报道屡见不鲜,成为了人们备受关注的公共问题,但相关报道仅限于临床表现。之前关于杨絮弱抗原性的报道也缺少系统的实验依据[3,4]。

抗原表位是抗原分子中决定抗原特异性的特殊化学基团,是与TCR、BCR或抗体特异性结合的结构单位,通常由5~17个氨基酸残基组成;抗原表位的性质、数目、位置和空间构象决定抗原表位的特异性;不同抗原之间含有相同或相似的抗原表位,可引起Ⅰ型过敏性疾病的交叉反应[5]。了解变应原的T细胞表位和B细胞表位无论对掌握变应原本身的生物活性,还是对由其引起的过敏交叉反应均具有重要意义。本研究提取杨絮总蛋白并酶解,鉴定到相关肽段22 023个,并通过生物信息学软件预测所得肽段中含有大量T细胞表位和B细胞表位,现将实验结果报告如下,旨在为杨絮引起的过敏性疾病的防治提供参考。
1、 材料与方法
1.1 、样本采集
本研究的杨絮样本采集于安徽省芜湖市长江大堤鲁港段,采集时间为2018年4月20日至2018年5月20日,采集树种为美洲黑杨南林895树种,树上采集杨絮放置于采样袋,带回实验室,-80 ℃冰箱保存备用。
1.2、 蛋白提取及浓度测定
样本从-80 ℃冰箱取出,称取20 g至液氮预冷的研钵中,加液氮充分研磨至粉末后加入4倍粉末体积酚抽提缓冲液(含10 mmol/L二硫苏糖醇,1%蛋白酶抑制剂,2 mmol/L EDTA),超声裂解后加入等体积的Tris平衡酚,4℃、5 500×g离心10 min,取上清并加入5倍体积的0.1 mol乙酸铵/甲醇沉淀过夜,蛋白沉淀分别用甲醇和丙酮进行洗涤,最后沉淀用8 mol/L尿素复溶,利用二喹啉甲酸(bicinchoninic acid,BCA)试剂盒进行蛋白浓度测定,操作步骤按试剂盒说明书进行。
1.3、 胰酶酶解
蛋白溶液中加入二硫苏糖醇使其终浓度为5 mmol/L,56 ℃还原30 min。之后加入碘代乙酰胺使其终浓度为11 mmol/L,室温避光孵育15 min。最后将样本的尿素浓度稀释至低于2 mol/L。以1∶50的质量比例(胰酶∶蛋白)加入胰酶,37 ℃酶解过夜。再以1∶100的质量比例(胰酶∶蛋白)加入胰酶,继续酶解4 h。
1.4、 高效液相色谱法(high performance liquid chromatography,HPLC)分级
肽段用高pH值反相HPLC分级,色谱柱为Agilent 300Extend C18(粒径5 μm,内径4.6 mm,长250 mm)。操作如下:肽段分级梯度为8%~32%乙腈、pH 9,60 min时间分离60个组分,随后肽段合并为6个组分,合并后的组分经真空冷冻干燥后进行后续操作。
1.5、 液相色谱-质谱联用分析
肽段用液相色谱流动相A相溶解后使用EASY-nLC 1000超高效液相系统进行分离。流动相A为含0.1%甲酸和2%乙腈的水溶液;流动相B为含0.1%甲酸和90%乙腈的水溶液。液相梯度设置:0~40 min,7%~25% B;40~52 min,25%~35% B;52~56 min,35%~80% B;56~60 min,80% B,流速维持在700 nL/min。肽段经由超高效液相系统分离后注入NSI离子源中电离,然后进Q Exactive质谱进行分析。
1.6、 T细胞表位和B细胞表位预测
选取与人类过敏性疾病相关的等位基因HLA-DRB1*03:01和HLA-DRB1*04:01[6],利用免疫表位数据库和分析资源(Immune Epitope Database and Analysis Resource, IEDB)对鉴定到的肽段进行表位预测,网址为http://www.iedb.org。其中T细胞表位选择CD4+T细胞MHC Ⅱ类表位,线性B细胞表位选择Bepipred Linear Epitope Prediction 2.0预测方法,阈值设置为0.500。
2、 结果
2.1 、蛋白电泳及浓度测定
对提取的杨絮总蛋白行SDS-PAGE分析,杨絮总蛋白在10 000~35 000之间出现多条清晰蛋白条带,35 000以上出现多条隐约条带(图1);BCA试剂盒检测蛋白总浓度为5.53 μg/μL。
2.2、 质谱质控检测
肽段的质量偏移(mass error)可见,质量误差以0为中轴并且集中在<10 ppm的范围内(图2A),且绝大部分的肽段长度分布在8~20个氨基酸残基之间(图2B)。
图1 20 μg杨絮总蛋白行12% SDS-PAGE分析结果
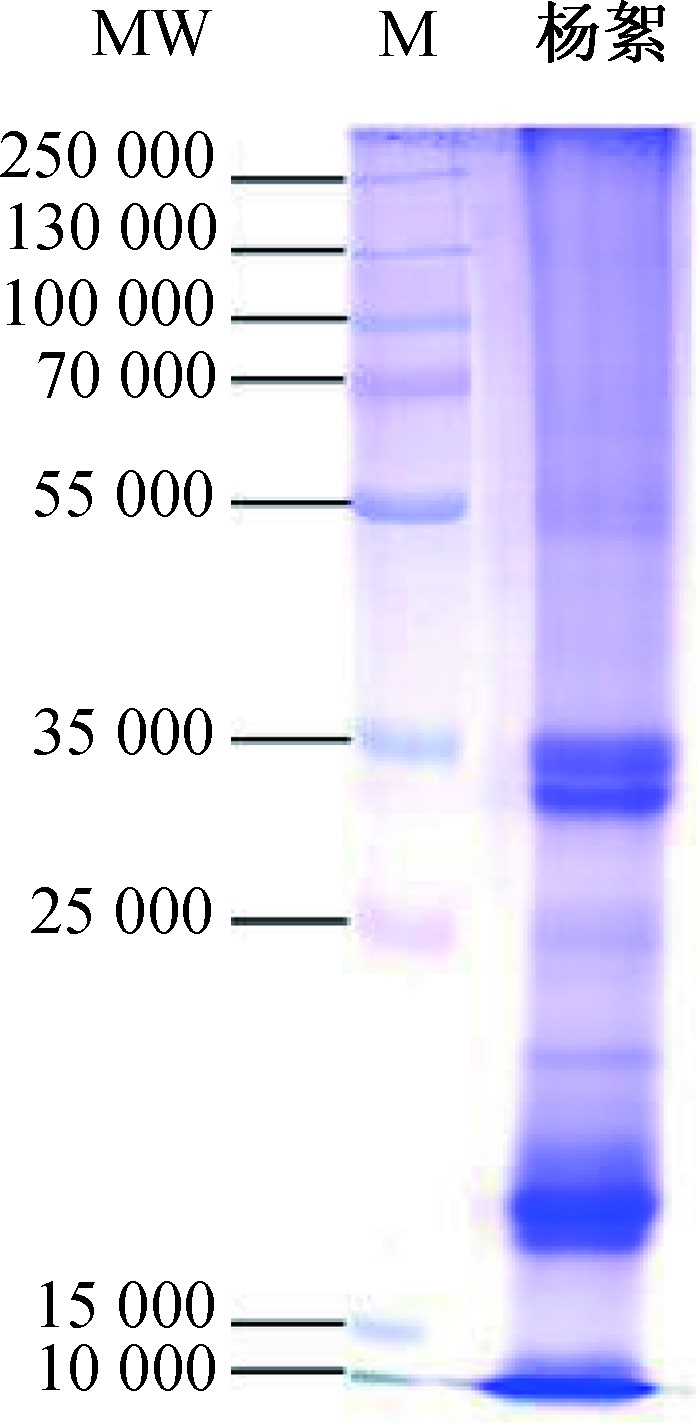
注:MW为蛋白相对分子质量;M为蛋白标记物
2.3、 T细胞表位预测
经在线预测,共筛选出具有高亲和力的CD4+T细胞表位16个(表1),中等亲和力的T细胞表位约2 300个(数据未列出)。
2.4、 B细胞表位预测
经在线预测,共筛选出线性B细胞表位10个,其预测分值在0.501~0.608之间。(表2)
3、 讨论
变应原的T、B细胞表位是诱发过敏性疾病的功能单位,同时也是发生交叉反应的结构基础。由于植物源性变应原具有广泛的植物种、属,甚至科的共同结构属性和生物活性,不同种属的变应原之间发生交叉过敏反应的现象较为常见[7,8]。随着生物信息学技术的发展,越来越多的T、B细胞表位预测软件和数据库被广泛的应用于疫苗设计及疾病诊断中[9,10]。利用IEDB,通过等位基因的设定,可提高HLA Ⅱ类限制性T细胞表位的准确率[11],NN法是基于此背景下派生出的新型CD4+ T细胞MHC Ⅱ类表位预测方法,进一步提升了表位预测精度[12]。B细胞有线性表位和构象表位2种类型,受多肽链的亲水性、柔韧性、螺旋构象、暴露表面及极性等多种因素影响;BepiPred 2.0是一种运用随机森林算法,基于抗体-抗原间互相作用进行表位注释的预测方法[13],Zhao等[14]通过实验验证了该B细胞表位预测方法具有较高精度。
图2 质谱数据的质控检测结果
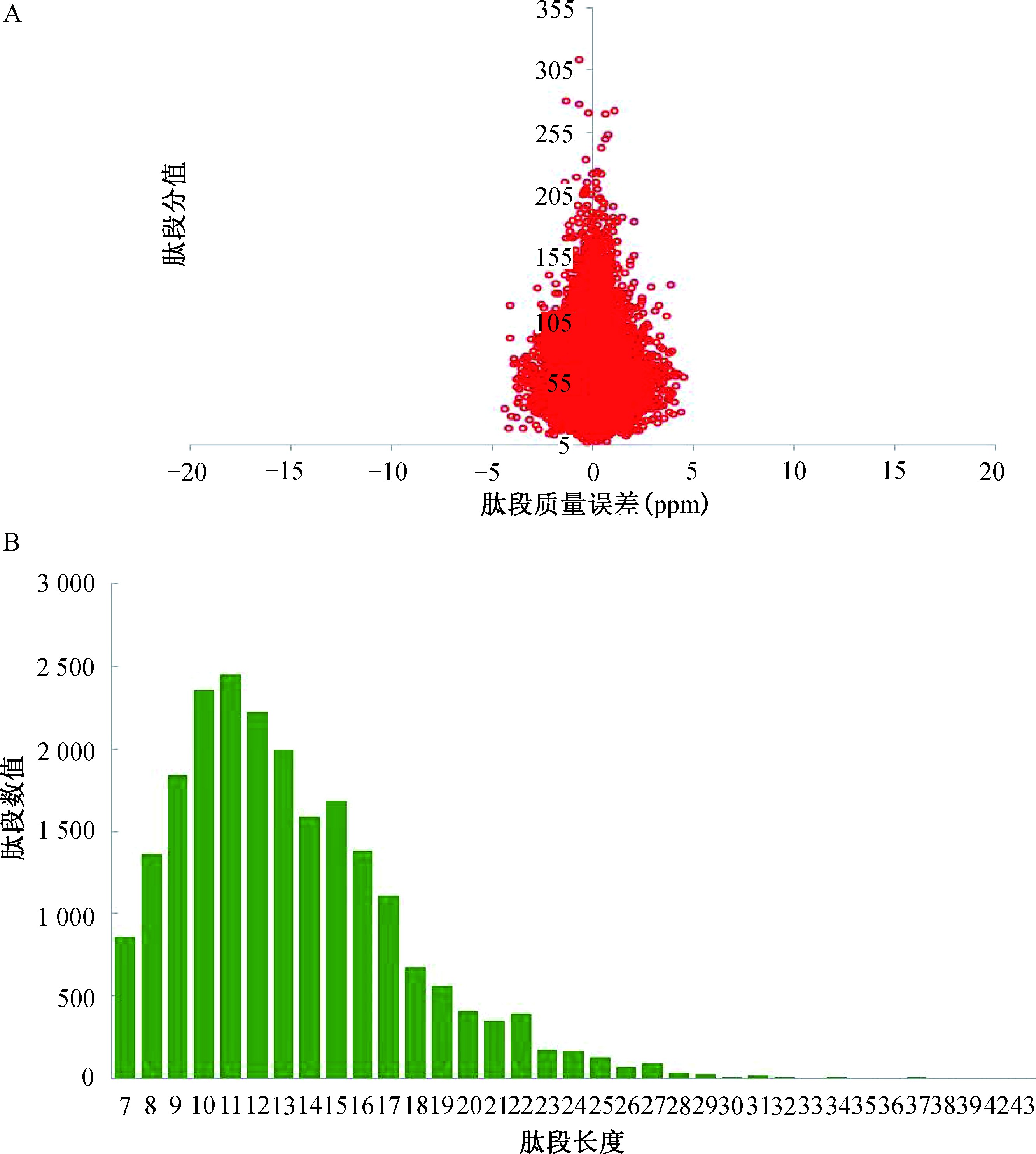
注:A.鉴定肽段的质量偏移分布;B.鉴定肽段的长度分布
表1 CD4+T细胞表位预测结果
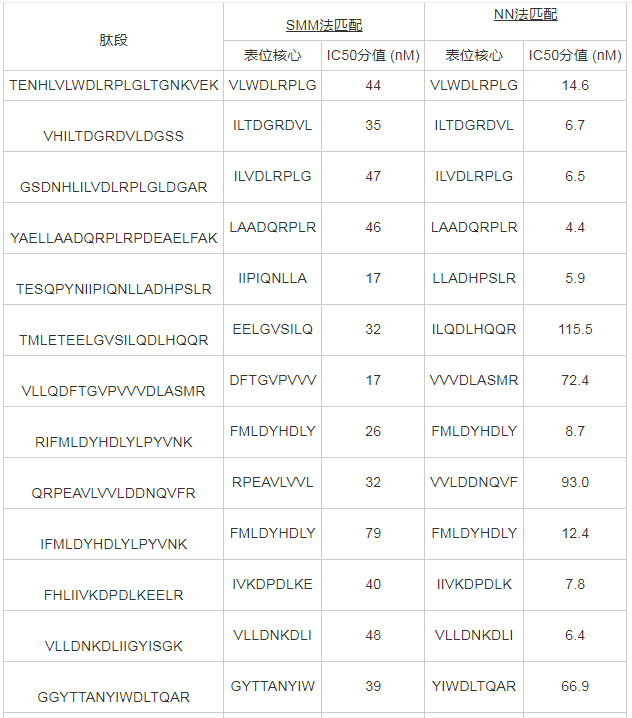
注:SMM法和NN 法为2种不同的预测方法;IC50分值为亲和力值,IC50分值 < 50 nM被认为具有高亲和力,< 500 nM被认为具有中等亲和力,IC50分值越小亲和力越高
表2 B细胞表位预测结果

注:分值高于阈值(0.500)的残基被认为是表位的一部分
本研究提取了杨絮总蛋白,SDS-PAGE显示其中含有丰富的蛋白组分,总蛋白经胰酶酶解,HPLC分级,液相色谱-质谱联用进一步剔除污染肽段,并通过质谱质控检测出所得肽段质量误差符合实验要求,且长度符合胰酶消化肽段规律,从而保证了后续实验的准确性。通过预测,共筛选出16个高亲和力及大量中等亲和力的T细胞表位,同时筛选出10个线性B细胞表位,该结果提示杨絮总蛋白可能具有引起过敏性疾病的变应原成分。Zhang等[15]通过在线软件预测出美洲黑杨花粉的28个IgE表位并得到验证;Wang等[16]通过变应原结构数据库(structural database of allergenic proteins,SDAP)预测出毛白杨花粉中27个候选变应原,并通过qRT-PCR验证了候选变应原基因在花粉中高表达。本研究结果同上述研究相符。本研究后期将通过动物实验对上述预测的T、B细胞表位进行验证,同时也对该预测方法的准确性进行评估。
综上所述,本研究通过预测,从杨絮总蛋白中筛选出了T、B细胞表位,提示杨絮中可能含有引起过敏性疾病的变应原成分,该结果丰富了花粉变应原的理论研究,同时也为杨絮等花粉变应原引起的过敏性疾病的防治提供了理论依据。
参考文献
[1] 叶艳涛,刘中山,化振奎,等.打造周口市“有杨树无杨絮城市”的设想[J].河南林业科技,2018,38(3):43-44.
[2] 张美英.蒙城县杨树飞絮的危害及治理对策[J].安徽农学通报,2018,24(15):120-147.
[3] Lin RY,Clauss AE,Bennett ES.Hypersensitivity to common tree pollens in New York City patients[J].Allergy Asthma Proc,2002,23(4):253-258.
[4] Kadocsa E,Bittera I,Juhász M.Aeropollinologic and allergologic studies for the clarification of "poplar tree hay fever"[J].Orv Hetil,1993,134(38):2081-2083.
[5] 金伯泉,熊思东.医学免疫学[M].第5版.北京:人民卫生出版社,2009:25-27.
[6] Horne C,Quintana PJ,Keown PA,et al.Distribution of DRB1 and DQB1 HLA class Ⅱ alleles in occupational asthma due to western red cedar[J].Eur Respir J,2000,15(5):911-914.
[7] Pauli G,Metz-Favre C.Cross reactions between pollens and vegetable food allergens[J].Rev Mal Respir,2013,30(4):328-337.
[8] Vieths S,Scheurer S,Ballmer-Weber B.Current understanding of cross-reactivity of food allergens and pollen[J].Ann N Y Acad Sci,2002,964:47-68.
[9] Raza S,Siddique K,Rabbani M,et al.In silico analysis of four structural proteins of aphthovirus serotypes revealed significant B and T cell epitopes[J].Microb Pathog,2019,128:254-262.
[10] Hua R,Jiang X,Qi L,et al.Screening HCV genotype-specific epitope peptides based on conserved sequence analysis and B cell epitope prediction in HCV E2 region[J].Immunol Res,2018,66(1):67-73.
[11] Paul S,Lindestam Arlehamn CS,Scriba TJ,et al.Development and validation of a broad scheme for prediction of HLA class II restricted T cell epitopes[J].J Immunol Methods,2015,422:28-34.
[12] Nielsen M,Lund O.NN-align.An artificial neural network-based alignment algorithm for MHC classⅡ peptide binding prediction[J].BMC Bioinformatics,2009,10:296-305.
[13] Jespersen MC,Peters B,Nielsen M.BepiPred-2.0:Improving sequence-based B-cell epitope prediction using conformational epitopes[J].Nucleic Acids Res,2017,45(W1):W24-W29.
[14] Zhao L,Li J.Mining for the antibody-antigen interacting associations that predict the B cell epitopes[J].BMC Struct Biol,2010(Suppl 1):S6.
[15] Zhang J,Wu LS,Fan W,et al.Proteomic analysis and candidate allergenic proteins in populus deltoides CL.“2KEN8” mature pollen[J].Front Plant Sci,2015,6:548-570.
[16] Wang L,Zhang X,Zhang J,et al.Proteomic analysis and identification of possible allergenic proteins in mature pollen of populus tomentosa[J].Int J Mol Sci,2018,19(1):E250.





