摘 要: 从古代原始材料中提取古DNA的方法多种多样,但是古DNA的研究受限于降解严重,内源性古DNA含量低,微生物和现生人群DNA污染严重等因素的影响。能否从古代人类遗骸中成功获取可靠且足量的内源性古DNA,一直是古DNA研究领域面临的实际困难和挑战。控制污染最直接且简便的策略就是在古DNA提取阶段的有效排除,本文整理了古DNA提取常用的去除污染的方法,对比分析了每种方法表现出来的优缺点。介绍了通常使用的骨粉裂解时间,并研究了在常温环境下,不同的裂解时间对古DNA回收效率的影响,提出了常温裂解过程中最佳孵育时间。同时对常用的古DNA纯化方法及其原理和在实际应用中的表现进行了概述与讨论。本文对古DNA提取技术的概述和实践经验,为古DNA相关领域的研究提供借鉴与参考。
关键词: 古DNA; 提取技术; 污染防控;
Abstract: There are many methods to extract ancient DNA from ancient raw materials. Generally, the research of ancient DNA is subject to factors such as serious degradation, while the low content of endogenous ancient DNA, the high content of microbial and modern human DNA. Whether we can successfully obtain reliable and sufficient endogenous ancient DNA has always been a practical difficulty and challenge in the field of ancient DNA research. The most direct and convenient strategy to eliminate the pollution effectively is in the procedure of ancient DNA extraction. This paper summarized the common methods of ancient DNA extraction to remove exogenous pollutants. We compared and analyzed the advantages and disadvantages of each method. This paper also introduced the time commonly used in the bone lysis step and suggested the best incubation time was 4 days at room temperature by exploring the effect of different lysis time on the recovery efficiency of ancient DNA. At the same time, we surveyed the representative methods of ancient DNA purification and the performance in the application. Our summary and experience could provide reference information for researchers in the field of ancient DNA research.
Keyword: Ancient DNA; Extraction technology; Pollution prevention and control;
1 、引言
古DNA是指古代生物遗体或遗迹中残存的DNA片段,包括古代人类DNA、古代动植物、微生物DNA[1,2,3,4]。古DNA研究为构建和复原古代人群遗传结构和种族属性[1,2,3,4,5,6,7]、群体间的交流及迁移模式[8,9],以及灭绝物种的系统发育学等研究打开了通道[10,11,12]。古DNA的研究同时暗示我们,从原始时期以来,人类的迁徙和族群间的同化一直伴随着冲突、战争和疾病,近些年来古DNA的研究领域开始渗透于传染病和瘟疫历史等领域的研究[13]。当前,在抗击新冠肺炎疫情的战斗中,复旦大学的古DNA研究团队成功的将古DNA研究技术和经验应用于新冠病毒的诊断,并致力于构建新冠病毒基因序列档案库,有利于加深对新冠病毒的认识,防范潜在的各类风险[14]。得益于考古基因组学这一新兴学科的出现,我们才可以利用古老的DNA重现人类祖先的历史,通过这种方式,我们获得了全新的视角,对人类认识过去、把握现在和选择将来都会产生重大影响。

古DNA的保存状况不仅仅取决于样本材料的年代,更重要的是受遗址所处环境的影响[15,16]。目前认为古DNA的保存机制可能与牙齿和骨骼中的主要成分羟基磷灰石和胶原质相互作用有关[15,17,18]。古DNA与羟基磷灰石相互作用的机制是通过带正电的钙离子与DNA分子中带负电的磷酸基团之间相互吸附产生的。羟基磷灰石结合的DNA与游离的DNA相比,降低了脱嘌呤的作用[19]和DNA被核酸酶降解[20]的风险。目前,DNA与胶原质的相互作用及其在古代组织中长期保存的机制还不清楚,但有人推测,在体内水溶液中自发形成的DNA-胶原质复合物可能对古DNA起到保护作用[17,21]。
虽然古DNA研究技术逐步走向成熟,但是古DNA的污染问题一直面临着巨大考验。遗骸中的骨和牙基质中不仅有内源性DNA(即死亡前或死亡时存在于生物体内的DNA),而且还有外源DNA,例如微生物在分解过程中进入骨或牙基质中的DNA。除了少数的例外[22,23,24],微生物DNA通常占古DNA提取液的95%以上,影响了高通量测序技术的经济高效性。虽然通过对部分DNA片段[25,26,27]或者全基因组杂交捕获的方法[28]可以缓解外源DNA污染带来的影响,提高古DNA测序效率,但是这一策略耗时耗力,并容易导致基因组单核苷酸突变(SNP)对比和识别的偏向性[29,30]。然而,更复杂的是古人类遗骸或古DNA通常被现代人类DNA污染,在这种情况下利用古DNA中的尿嘧啶(U),从现代DNA污染中把内源性古DNA分离出来。尿嘧啶(U)是由胞嘧啶(C)脱氨作用诱导并随时间积累的,优先在DNA分子[31]末端的单链悬链中积累。DNA聚合酶将尿嘧啶解读为胸腺嘧啶(T),从而与参考基因组产生C到T的差异,并使古老的DNA分子得以鉴定。携带尿嘧啶的DNA分子也可以在文库制备过程中被物理分离[32]。然而,这两种方法丢弃了大多数不含去氨胞嘧啶残基的古DNA片段,导致内源性古DNA大量丢失。
控制污染最直接的办法就是在古DNA提取阶段,对古代遗骸中微生物和现代人类DNA进行去除。然而,大多数DNA提取方法都是针对含有完整细胞和高分子量DNA的新鲜组织而设计的。古代遗骸中的DNA,通常没有细胞结构,而且由于没有特征性的化学修饰,DNA甚至可能难以溶于水溶液中[33]。古DNA在长期的环境中已经遭受了严重损伤和降解[34,35,36],因此在提取过程中应避免再次受损,特别是过度腐蚀性处理,例如高温或使用强力表面活性剂[37]。尽管这些处理可能会增加DNA的释放,但它们会对古DNA分子造成进一步的损伤,从而降低DNA的总产量。古代骨骼和牙齿通常含有大量的PCR抑制剂[38,39],它们干扰DNA扩增,并与古DNA一起被纯化。现在越来越多的研究除了使用线粒体序列外,还使用核DNA序列,与线粒体DNA相比,核DNA在活细胞中的拷贝数要低得多,因此很难从低质量的样本中获得。因此,获得高质量的内源性的DNA一直是古DNA提取方法不断改进的方向。为了提高古DNA的回收率,近年来出现了多种优化的古DNA提取技术。本文综述了一些具有代表性的古DNA提取方法,并比较了各种方法的优缺点,对古DNA的提取提供参考和借鉴。
2、 骨粉样品的预处理
尽管近些年在古DNA提取方法的改进方面已经取得了很大进展,但由于微生物或现代人类DNA的大量污染,也经常会出现一些样本仍然无法进行分析。通过化学或酶处理是避免不同来源的现代人类或微生物DNA污染的有效方法,适当的预处理可以提高内源性古DNA的获得。骨粉样品的预处理已经报道了很多方法,包括用磷酸盐缓冲液、次氯酸钠缓冲液[18]、蛋白酶[40]、EDTA或酸处理骨骼或骨粉[41]。以下概述了三种典型的骨粉预处理方法和其在古DNA研究中的应用。
2.1、 次氯酸钠预处理
次氯酸钠处理骨头或骨粉[41,42]是法医学研究中最常用的去除外源DNA污染的方法,也被用于古代DNA的研究[43]。在不同的研究中,关于次氯酸钠去污染效率的报道各不相同,从使用≥3%的次氯酸钠溶液[41]完全去除人体污染物到不考虑次氯酸钠浓度的不完全去除污染物。然而,据报道,次氯酸钠处理骨粉后,大约四分之三的内源性DNA丢失[44]。Korlevi?[18]对不同浓度下,次氯酸钠溶液处理后的内源性古DNA的丢失和损伤程度进行了对比研究。他将250mg骨粉分成五等分,除了设置的一个空白对照,其余四份分别加入0.1%、0.5%、2%和6%浓度的次氯酸钠溶液。然后用EDTA/蛋白酶K裂解液进行孵育完成古DNA的释放,最后用二氧化硅方法进行纯化[45]。通过检测古DNA文库分子量和shotgun测序之后的古DNA片段长度的对比分析,发现使用0.5%次氯酸钠溶液处理的骨粉样品,虽然古DNA有一定程度的丢失,但是不会对古DNA造成损伤[18]。
在重建3.9万至4.7万年前尼安德特人的遗传历史研究中,大量尼安德特人内源性古DNA保存不善且受到大量微生物和现代人类DNA的污染。当骨粉或牙粉进行次氯酸盐处理后再进行古DNA的提取,有效的去除了外源污染物,增加了内源性古DNA的释放率和提取效率,使得五个尼安德特人的基因组覆盖率提高了1到2.7倍,且获得有效的基因组序列的尼安德特人样本数量增加了一倍[46]。
2.2 、基于EDTA/蛋白酶的预消化
骨粉在裂解过程中,骨结构降解,增加了骨的孔隙和总表面积[17]。与外源污染物相比,内源性古DNA位于更受保护的骨晶体内[47]。在骨材料的消化过程中,表面污染物将首先释放到溶液中[48]。因此,在短时间内用预消化缓冲液处理磨碎的骨材料会去除一部分外源性非靶向DNA,从而提高内源性DNA的富集效率。
预消化是一种简单有效的方法,可以从古代样本中去除一定比例的外源DNA。研究表明,基于EDTA酶(EDTA和蛋白酶)预消化骨骼粉末可将内源性DNA的比例提高数倍。使用较短的预消化时间(15-30分钟),在50°C下孵育,获得的内源性DNA是未使用预消化的14倍。但随着长时间的预消化,会影响内源性古DNA的回收率[48]。
对西伯利亚东北部晚更新世的人类历史研究中,在发现的距今3.1万至600年前的34个古人类DNA进行提取之前,骨粉中加入0.5M EDTA,蛋白酶K,100×TE和10%N-Laurylsarcosyl,在40°C环境中预消化45 min,尽可能多得排除了外源DNA的污染,并获得了有效和可靠的全基因组序列[49]。该项研究进一步说明了EDTA酶的预消化作用同样适用于从3.1万~600年前这样大跨度时间范围内的古DNA提取。
2.3 、磷酸盐缓冲液预处理
古DNA与羟基磷灰石是通过带正电的钙离子与DNA分子中带负电的磷酸基团吸附而相互作用的。而游离的磷酸盐离子与DNA主链上的磷酸基团相互竞争结合到羟基磷灰石上[50,51],使得DNA游离于溶液中,这一原理通常在液相色谱中用于分离核酸[50]。同时磷酸盐缓冲液预处理也被用于古DNA的提取中的外源DNA的去除[52,53]。
Korlevi?[18]对磷酸钠缓冲液预处理条件进行了研究,使用欧亚大陆晚更新世骨骼和牙齿样本,加入0.5 M磷酸钠缓冲液,p H 7.0,常温振荡孵育,并预处理三次,研究发现大多数污染物DNA在第一次磷酸盐孵育过程中被释放出来,当缩短第一次孵育培养时间时,可能会获得更有利的结果。所以建议每次孵育时间使用15 min,可有效的将外源DNA先释放到溶液中得以去除。
3、 裂解时间的研究
骨粉的裂解时间直接影响古DNA的释放和回收率,裂解时间如果不够,古DNA不能完全从骨骼晶体内释放出来。如果裂解时间过长和孵育温度过高,又会导致古DNA进一步损伤。骨粉裂解常用的孵育温度为37℃,因为此温度下蛋白酶K的活性最高[54,55],裂解时间通常为16~24小时[56]。对于骨粉颗粒比较大的样品来说,裂解完成后在56℃温度下,再次孵育1~3小时,可以增加更多古DNA的释放[57]。也有研究在使用预处理过程时,首先加入0.5M EDTA(p H 8.0)去除样本表面污染物和保持DNA的稳定性,常温孵育24小时,离心收集沉淀物,加入蛋白酶K和DTT,50~55℃温度下孵育24小时[2],使DNA完全释放到溶液中。
为了防止温度的升高对古DNA的进一步损伤,我们研究了在常温下,不同的孵育时间对DNA提取效率的影响。研究样本来自于仰韶文化晚期的五庄果墚遗址,该遗址位于陕西省靖边县黄蒿界乡小界村。选取了其中一具遗骸中的六颗牙齿并研磨成粉末,将同一骨粉样品分装为200 mg、500 mg和1 g各五等份,分别加入裂解缓冲液(0.45 M EDTA和0.25 mg/ml的蛋白酶K,p H 8.0),恒温裂解摇床转速设为250r/min振荡孵育。同一骨粉量的5个等分样品孵育时间分别设为24、48、72、96和120小时,孵育时间超过48小时添加一次0.25 mg/ml的蛋白酶K。孵育完成后用二氧化硅层析柱进行纯化(Qiagen,28004),再加入30μl的TE缓冲液(10 m M Tris,1 m M EDTA,p H 8.0)进行洗脱,得到古DNA提取液。用微量紫外分光光度计对古DNA提取液进行浓度的初步检测,检测到的为总的DNA浓度,其中除了内源性古DNA还包括了微生物DNA。然后用现代人的基因组,扩增预先设计好的一段序列并确定其浓度,用已知浓度的现代人DNA样品制备出具有稳定扩增的DNA溶液作为参照(标准品),借助实时荧光定量PCR(Polymerase Chain Reaction,PCR)对古DNA提取液进行扩增,参照已知浓度的标准品梯度范围,计算排除微生物DNA污染的古DNA起始浓度。制备标准品的过程是:首先,根据需要设计扩增引物(F15977-R16509:5’-CCACCATTAGCACCCAAA-3’;-5’-AGGAACCAGATGTCGGATA-3’),以现代人基因组为模版扩增目标片段,用琼脂糖凝胶电泳进行目标片段的分离和纯化,将目标片段克隆进质粒载体,构建含有目标片段的重组质粒,以重组质粒作为模版进行扩增,并选择适合检测古DNA起始浓度的标准品浓度梯度,将制备的标准品与古DNA提取液同时进行实时荧光定量PCR,以标准品作为参照,计算古DNA的起始模板量。实时荧光定量PCR结果如图1所示,结合PCR实时监测的扩增曲线(左侧)和标准曲线(右侧),对比200mg、500mg和1g骨粉在不同裂解时间下的浓度变化,研究结果发现常温孵育96小时(4天)获得的DNA浓度最大,而孵育120小时后浓度稍有降低,表明孵育时间超过96小时后,DNA可能开始出现明显的降解。所以我们建议如果是在常温条件下孵育进行骨粉裂解过程,孵育时间使用96小时为宜。
图1 常温条件下不同的孵育时间对DNA提取效率的影响
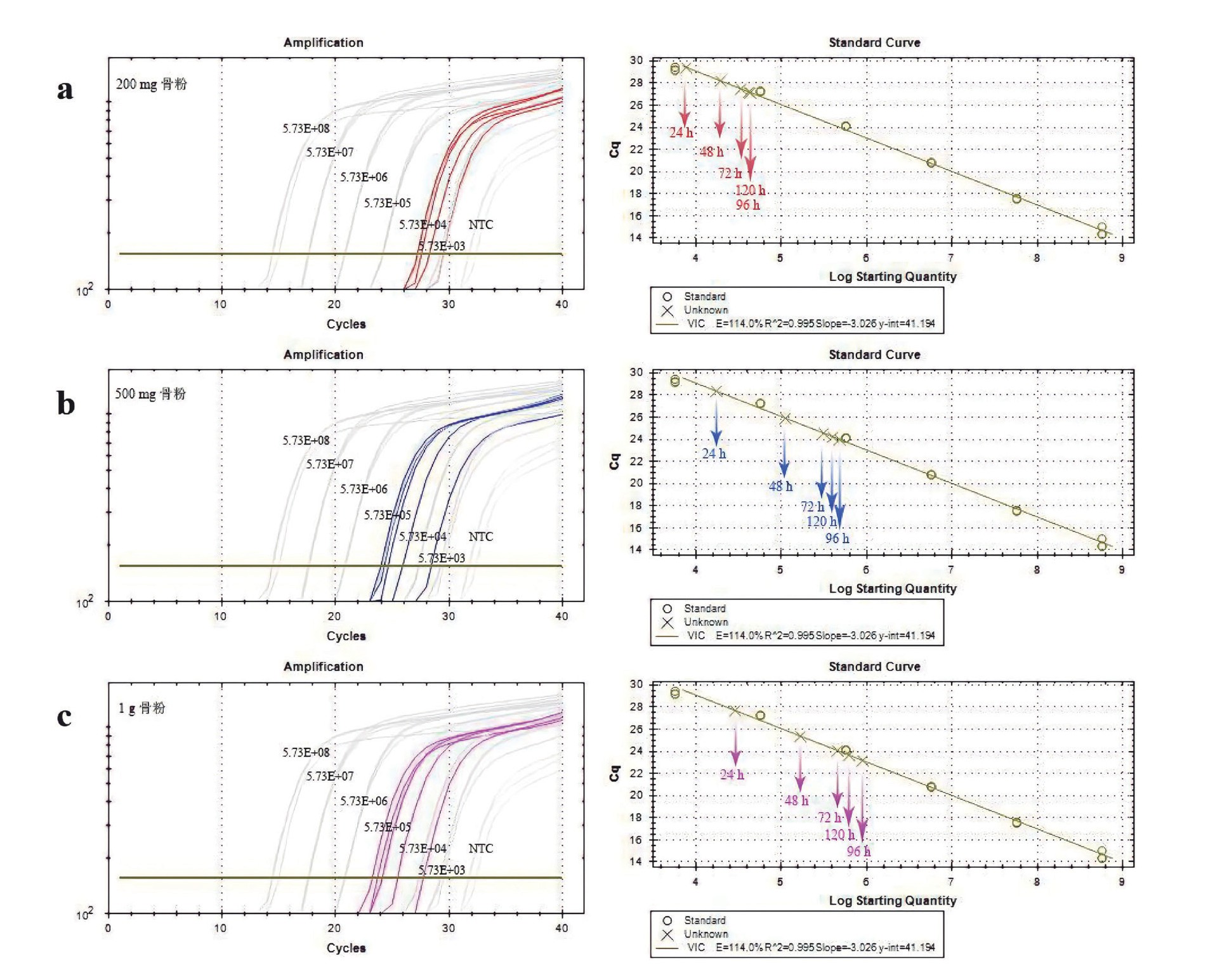
Fig.1 The effect of different incubation time on DNA extraction efficiency at room temperature
注:左侧为扩增曲线,X轴对应PCR扩增循环数,Y轴对应荧光强度值;右侧为标准曲线,X轴为模板的起始拷贝数的对数,Y轴为每个反应荧光信号到达设定阈值时所经历的循环数。a 200mg骨粉在5个不同裂解时间下的扩增曲线和标准曲线;b 500mg骨粉在5个不同裂解时间下的扩增曲线和标准曲线;c 1g骨粉在5个不同裂解时间下的扩增曲线和标准曲线。
4 、古DNA纯化方法
4.1、 基于二氧化硅(硅粒)的古DNA纯化
当溶液的p H值低于或等于二氧化硅表面电离常数时,二氧化硅表面携带的负电荷减少,因而与DNA分子带的负电荷之间的排斥力也随之减弱,会形成很多的水合离子,降低了DNA分子的水合程度,从而DNA分子集聚到二氧化硅的表面。收集DNA时,先用洗涤缓冲液洗掉吸附有DNA分子的二氧化硅表面的其他杂质,再用p H值高于电离常数的溶液洗脱吸附在二氧化硅表面上的DNA分子,从而获得DNA提取液。
在二氧化硅颗粒纯化古DNA方法中,所有的步骤都是在室温(20-23℃)下进行的,从而减少了对脆弱的古DNA造成进一步损伤,并去除了古代骨骼样本中存在的大多数不同类型的PCR抑制剂,为后续古DNA实验操作(如PCR扩增)提供稳定环境。该方案可方便地适用于不同样品量的骨粉和提取体积。该方法操作步骤较少,需要的设备简单,可在2个工作日内完成古DNA的提取。但是在实际操作中,如果操作不慎易造成吸附DNA的二氧化硅颗粒的外溅和损耗,会降低古DNA的回收率。
4.2 、硅离心柱纯化古DNA
古DNA提取中常用的二氧化硅离心柱主要分两种,一种是直接采用商业化的硅胶离心柱(Qiagen,28004)[58,59]对古DNA进行富集;另一种是通过优化商业化的色谱柱,得到更高效的富集古DNA的硅离心柱:首先将二氧化硅颗粒固定在1μm孔径的色谱膜中(Whatman,1.0μm),然后将固定有二氧化硅颗粒的色谱膜放入商业化的色谱柱中[60](Mobicols,M1002S,10μm孔径)。这种改良的二氧化硅离心柱在古DNA提取中也被广泛的应用[61,62]。
直接采用商业化的硅胶离心柱操作简便、大大减少了提取时间和人力的消耗,但是商业化的硅胶离心柱会使古DNA的回收效率受到产品本身的影响,比如受硅胶的粒径、密度、纯度和固定方式等因素的影响,会使古DNA的吸附和洗脱效率有很大差别。在实际应用中,我们发现用Qiagen品牌下的Min Elute硅胶离心柱对古DNA的回收效率明显优于国产的某些硅胶离心柱。另外,商业化的硅胶离心柱回收的DNA产量是有限的[37],且通常不适合大样本体积量的使用。用商业化的色谱柱优化的二氧化硅离心柱可以防止在离心过程中,细小的二氧化硅颗粒的丢失。但是用色谱柱制备成二氧化硅离心柱过程较为繁琐,增加操作步骤和工作量,容易将外源DNA污染再次引入古DNA提取。
4.3 、磁珠法纯化古DNA
磁珠法提取DNA常用的是硅质膜二氧化硅磁珠,是由超顺磁性内核和二氧化硅外壳组成,表面修饰大量的硅羟基。磁珠表面的硅羟基能够与溶液中的核酸通过氢键和静电作用发生特异性结合,在高盐条件下与核酸结合,而在低盐环境下被洗脱,在外加磁场作用下,磁性颗粒与溶液分离,回收磁珠颗粒(即磁珠-DNA混合物),即可以直接从复杂生物体系中迅速分离核酸[63]。
该方法也被应用于古DNA的提取[64],从新石器时期(5000年前)的遗骸材料中,成功提取了古DNA,并完成了线粒体DNA的扩增和分析。值得注意的是,该方法在整个纯化过程不需要离心,简化了古DNA提取步骤,大大缩短了提取时间,易于操作,成本低廉,设备需求简单。
4.4 、乙醇/异丙醇沉淀法纯化古DNA
根据乙醇和异丙醇能够沉淀核酸,实现DNA的分离和纯化的原理,被应用于古DNA的提取。异丙醇比乙醇沉淀DNA的效果要好,因为异丙醇比较疏水,可以较好的沉淀和分离DNA。但是乙醇比异丙醇更加亲水,能够有效的去掉盐离子,且易于挥发,对下游的实验影响较小。所以在沉淀核酸时,通常在使用异丙醇之后,再用70%的乙醇多次洗涤脱盐。这种方法快速,既不需要危险化学品,也不需要特殊的设备,可以减少耗时的分离脱钙步骤、透析、离心驱动的微浓缩等过程[65]。
乙醇/异丙醇沉淀法对于血液样本DNA提取具有很好的效果,操作简便、成本低廉、提取的DNA质量高,但是对于古DNA的提取却存在弊端。古DNA含量极低,在乙醇/异丙醇沉淀的过程中,很难看到白色絮状沉淀物,在DNA抓取中容易漏掉一定量的古DNA。古DNA高度片段化,有相当一部分片段小于100bp;对于分子量极小的古DNA片段,在乙醇/异丙醇沉淀离心后,很难回收到短片段的古DNA,这样就会造成内源性古DNA的丢失。
5、讨论与结语
从古代原始材料中提取DNA方法有很多种,样品预处理和提取过程需要考虑不同来源的现代和微生物DNA的污染。能否从古代人类遗骸中成功获取内源性古DNA并进行深入研究分析,一直是古DNA研究领域面临的实际困难和挑战。控制污染最直接的手段就是古DNA提取阶段,骨粉的预处理过程可以大幅度排除古代遗骸中微生物和现生人类DNA的污染。但在去除污染的过程中,有些预处理方法又会带来新的问题,比如次氯酸钠缓冲液预处理可以增加古DNA的释放率和序列匹配率,浓度的使用不当很容易造成内源性古DNA损伤;磷酸盐缓冲液也会增加古DNA序列的匹配率(使用磷酸盐缓冲液预处理,古DNA序列的匹配率会增加两倍,而用次氯酸钠预处理古DNA的匹配率会增加4.6倍[18]),但是内源性古DNA释放得较少[18]。虽然次氯酸钠和磷酸盐处理可有效去除微生物DNA的污染,且大大降低了古代材料基因组测序的成本,但去除人类污染方面表现着低效性[18]。需要注意的是,文库中污染的百分比并不一定反映骨粉中存在的污染水平,因为一些现代人类DNA污染是在实验室操作中引入的。次氯酸钠处理的骨粉尤其如此,内源性DNA分子的大量丢失放大了人类污染的影响,次氯酸钠处理不能保证去除人类污染[42,44],导致文库中现生人类DNA污染的百分比增加,因此不适用于不能重复取样的小而珍贵的标本。研究发现对同一骨粉样品用EDTA和蛋白酶进行预消化处理,内源性古DNA相对于微生物和现代人类污染的比例明显增加了[47,48,66]。但是此预消化过程需在50°C下孵育,应尽量减少孵育时间,通常为15-30分钟,以免长时间的预消化,内源性古DNA增加达到饱和,使得古DNA浓度下降。
骨粉的裂解温度和时间会影响古DNA的释放和回收率,为了防止孵育温度的升高对古DNA的进一步损伤。我们研究了常温环境下,200 mg、500 mg和1 g不同骨粉量,在裂解缓冲液(0.45 M EDTA和0.25 mg/ml的蛋白酶K)的作用下,不同的裂解时间对古DNA的回收率的影响,研究发现在常温环境中裂解骨粉的最佳时间为4天(96小时)。
也有研究将无损提取方法[67,68]用于古代骨骼和牙齿材料古DNA提取中,为了避免在研磨过程中对古DNA的进一步损伤,将古代样本材料直接浸泡在裂解缓冲液中,待古DNA完全释放进行纯化分析。无损古DNA提取方法也会得到较好质量的古DNA。但经过无损方法处理后的样本材料颜色变得更浅,而且出现比以前更多的空隙,这些结果似乎表明,无损提取技术虽然不会破坏原始样本材料外在的形貌,但也会损害材料内部的结构[69]。
古DNA的纯化回收方法多种多样,主要是基于二氧化硅(硅粒)、商业化的凝胶柱或自制的回收柱和醇类沉淀等方法。在使用这些方法时,古DNA的富集和回收效率会受很多外因的影响,比如DNA结合缓冲液的PH,富集DNA的硅胶或硅粒的结构、比表面积、质地,以及纯度等因素的影响。特别是购买的商业化的硅离心柱,不同厂家的产品,富集和回收效率也会存在差异,并通常不适合大样本量使用,且存在现生人群DNA污染的风险。对于较大样本体积,还需要注意的是使用的结合缓冲液和洗涤缓冲液时,需要尽量避免盐类和杂质融入古DNA提取液,以免影响后续建库、扩增和测序等过程的顺利进行。
一些人认为古材料中存在的DNA都是现代污染物造成的,所以符合古DNA研究标准的超净实验室和实验员操作过程中的严格防护,可以降低现生人群DNA的污染几率。适当的独立对照实验的引入,可以确认研究样本DNA的真实性,还可以确保不会错误地从微生物序列中进行扩增。
古DNA的研究受限于DNA保存环境,微生物DNA含量高,现生人群的DNA污染等因素的影响。虽然古DNA提取技术在不断改进,筛选内源性古DNA片段的方法不断在优化,获得可靠和足量的古DNA序列信息仍然面临着巨大考验。而降低古DNA污染最有效和方便的阶段就是在古DNA提取阶段的污染防控,本文整理了古DNA提取常用的去除污染的手段,对比分析了每种去污染方法表现出来的优缺点。总结了通常使用的骨粉裂解时间,并研究了在常温环境下,不同的裂解时间对古DNA回收效率的影响,提出了常温裂解过程最佳的孵育时间。此外,骨粉裂解完成后的纯化方法呈现多样性,列举了常用的纯化方法的基本原理,以及在古DNA研究应用中的表现进行了整理和总结。本文对古DNA提取技术进行了概述和讨论,希望为从事古DNA领域研究的学者们提供借鉴与参考。
参考文献
[1] Campos PF, Willerslev E, Sher A, et al. Ancient DNA analyses exclude humans as the driving force behind late Pleistocene musk ox(Ovibos moschatus)population dynamics[J]. Proceedings of the National Academy of Sciences of the United States of America,2010, 107(12):5675-5680
[2] Leonard JA, Wayne RK, Cooper A. Population genetics of ice age brown bears[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(4):1651-1654
[3] Pinsky ML, Newsome SD, Dickerson BR, et al. Dispersal provided resilience to range collapse in a marine mammal:insights from the past to inform conservation biology[J]. Mol Ecol, 2010, 19(12):2418-2429
[4] Shapiro B, Drummond AJ, Rambaut A, et al. Rise and fall of the Beringian steppe bison[J]. Science, 2004, 306(5701):1561-1565
[5] Stiller M, Baryshnikov G, Bocherens H, et al. Withering away--25,000 years of genetic decline preceded cave bear extinction[J].Mol Biol Evol, 2010, 27(5):975-978
[6] Ning C, Li TJ, Wang K, et al. Ancient genomes from northern China suggest links between subsistence changes and human migration[J]. Nature communications, 2020, 11(1):2700
[7] Wang K, Goldstein S, Bleasdale M, et al. Ancient genomes reveal complex patterns of population movement, interaction, and replacement in sub-Saharan Africa[J]. Science Advance, 2020, 6 eaaz0183
[8] Narasimhan VM, Patterson N, Moorjani P, et al. The Genomic Formation of South and Central Asia[J]. bio Rxiv, 2018,
[9] Green RE, Krause J, Briggs AW, et al. A draft sequence of the Neandertal genome[J]. Science, 2010, 328(5979):710-722
[10] Shapiro B, Sibthorpe D, Rambaut A, et al. Flight of the Dodo[J]. Science, 2002, 2951683
[11] Krause J, Unger T, Nocon A, et al. Mitochondrial genomes reveal an explosive radiation of extinct and extant bears near the Miocene-Pliocene boundary[J]. BMC Evol Biol, 2008, 8220
[12] Orlando L, Metcalf J, Alberdi M, et al. Revising the recent evolutionary history of equids using ancient DNA[J]. Proc Natl Acad Sci USA, 2009, 10621754-21759
[13] Barquera R, Krause J. An ancient view on host-pathogen interaction across time and space[J]. Current opinion in immunology,2020, 6565-69.
[14]吴苡婷.古DNA检测技术在抗击新冠中的特殊作用[N].上海科技报,2020.
[15] Collins MJ, Nielsen-Marsh CM, Hiller J, et al. The survival of organic matter in bone:a review[J]. Archaeometry, 2002, 44(3):383-394
[16] Hofreiter M, Paijmans JL, Goodchild H, et al. The future of ancient DNA:Technical advances and conceptual shifts[J]. Bioessays,2015, 37(3):284-293
[17] Campos PF, Craig OE, Turner-Walker G, et al. DNA in ancient bone-where is it located and how should we extract it?[J]. Ann Anat, 2012, 194(1):7-16
[18] Korlevic P, Gerber T, Gansauge MT, et al. Reducing microbial and human contamination in DNA extractions from ancient bones and teeth[J]. Bio Techniques, 2015, 59(2):87-93
[19] Lindahl T. Instability and decay of the primary structure of DNA[J]. Nature, 1993, 362709-715
[20] Brundin M, Figdor D, Sundqvist G, et al. DNA binding to hydroxyapatite:a potential mechanism for preservation of microbial DNA[J]. J Endod, 2013, 39(2):211-216
[21] Svintradze DV, Mrevlishvili GM, Metreveli N, et al. Collagen–DNA Complex[J]. Biomacromolecules, 2008, 921-28
[22] Reich D, Green RE, Kircher M, et al. Genetic history of an archaic hominin group from Denisova Cave in Siberia[J]. Nature, 2010,468(7327):1053-1060
[23] Prufer K, Racimo F, Patterson N, et al. The complete genome sequence of a Neanderthal from the Altai Mountains[J]. Nature,2014, 505(7481):43-49
[24] Gamba C, Jones ER, Teasdale MD, et al. Genome flux and stasis in a five-millennium transect of European prehistory[J]. Nature,communications, 2014, 55257
[25] Burbano HA, Hodges E, Green RE, et al. Targeted Investigation of the Neandertal Genome by Array-Based Sequence[J]. Science,2010, 328723-725
[26] Avila-Arcos MC, Cappellini E, Romero-Navarro JA, et al. Application and comparison of large-scale solution-based DNA captureenrichment methods on ancient DNA[J]. Sci Rep, 2011, 174
[27] Fu QM, Meyer M, Gao X, et al. DNA analysis of an early modern human from Tianyuan Cave, China[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(6):2223-2227
[28] Carpenter ML, Buenrostro JD, Valdiosera C, et al. Pulling out the 1%:whole-genome capture for the targeted enrichment of ancient DNA sequencing libraries[J]. Am J Hum Genet, 2013, 93(5):852-864
[29] Castellano S, Parra G, Sanchez-Quinto FA, et al. Patterns of coding variation in the complete exomes of three Neandertals[J].Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(18):6666-6671
[30] Meynert AM, Ansari M, Fitz Patrick DR, et al. Variant detection sensitivity and biases in whole genome and exome sequencing[J].BMC Bioinformatics, 2014, 15247
[31] Briggs AW, Stenzel U, Johnson PLF, et al. Patterns of damage in genomic DNA sequences from a Neandertal[J]. Proc. Natl. Acad.Sci, 2007, 10414616-14621
[32] Gansauge MT, Meyer M. Selective enrichment of damaged DNA molecules for ancient genome sequencing[J]. Genome Res, 2014,24(9):1543-1549
[33] Geigl E. On the circumstances surrounding the preservation and analysis of very old DNA[J]. Archaeometry, 2002, 44337-342
[34] H?ss M, Dilling A, Currant A, et al. Molecular phylogeny of the extinct ground sloth mylodon darwinii[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93181-185
[35] Hansen AJ, Mitchell DL, Wiuf C, et al. Crosslinks rather than strand breaks determine access to ancient DNA sequences from frozen sediments[J]. Genetics, 2006, 1731175-1179
[36] Hofreiter M, Jaenicke V, Serre D, et al. DNA sequences from multiple amplifications reveal artifacts induced by cytosine deamination in ancient dna[J]. Nucleic Acids Research, 2001, 294793-4799
[37] Rohland N, Hofreiter M. Comparison and optimization of ancient DNA extraction[J]. Bio Techniques, 2007, 42343-352
[38] Tebbe CC, Vahjen W. Interference of humic acids and DNA extracted directly from soil in detection and transformation of recombinant DNA from bacteria and a yeast[J]. Appl Environ Microbiol, 1993, 592657–2665
[39] Tuross N. The biochemistry of ancient DNA in bone[J]. Experientia, 1994, 50530-535
[40] Li R, Liriano L. A bone sample cleaning method using trypsin for the isolation of DNA[J]. Leg Med(Tokyo), 2011, 13(6):304-308
[41] Kemp BM, Smith DG. Use of bleach to eliminate contaminating DNA from the surface of bones and teeth[J]. Forensic Sci Int,2005, 154(1):53-61
[42] Barta JL, Monroe C, Kemp BM. Further evaluation of the efficacy of contamination removal from bone surfaces[J]. Forensic Sci Int, 2013, 231(1-3):340-348
[43] Salamon M, Tuross N, Arensburg B, et al. Relatively well preserved DNA is present in the crystal aggregates of fossil bones[J].PNAS, 2005, 102(39):13783-13788
[44] Malmstrom H, Svensson EM, Gilbert MT, et al. More on contamination:the use of asymmetric molecular behavior to identify authentic ancient human DNA[J]. Mol Biol Evol, 2007, 24(4):998-1004
[45] Dabney J, Knapp M, Glocke I, et al. Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013,110(39):15758-15763
[46] Hajdinjak M, Fu QM, Hubner A, et al. Reconstructing the genetic history of late Neanderthals[J]. Nature, 2018,
[47] Ginolhac A, Vilstrup J, Stenderup J, et al. Improving the performance of true single molecule sequencing for ancient DNA[J]. BMC Genomics, 2012, 13177
[48] Peter B. Damgaard AM, Hannes Schroeder, Ludovic Orlando,, Eske Willerslev MEA. Improving access to endogenous DNA in ancient bones and teeth[J]. Scientific Reports, 2015, 511184
[49] Sikora M, Pitulko VV, Sousa VC, et al. The population history of northeastern Siberia since the Pleistocene[J]. Nature, 2019,570(7760):182-188
[50] Bernardi G. Chromatography of Nucleic Acids on Hydroxyapatite[J]. Nature, 1965, 206779-783
[51] Grunenwald A, Keyser C, Sautereau AM, et al. Adsorption of DNA on biomimetic apatites:Toward the understanding of the role of bone and tooth mineral on the preservation of ancient DNA[J]. Appl Surf Sci, 2014, 292867-875
[52] Persson P. A method to recover DNA from ancient bones[J]. Ancient DNA Newsl, 1992, 125-27
[53] G?therstr?m A, Lidén K. A modified DNA extraction method for bone and teeth[J]. Laborativ Arkeologi, 1996, 953-56
[54] Bajorath J, Saenger W, Pal GP. Autolysis and inhibition of proteinase K, a subtilisin-related serine proteinase isolated from the fungus Tritirachium album Limber[J]. Biochimica et Biophysica Acta(BBA)-Protein Structure and Molecular Enzymology, 1988, 954:176-182
[55]刘杨柳,武小芳,胡树样,等.蛋白酶K的性质及其在核酸提取中的应用[J].食品研究与开发,2017, 38(10):196-199
[56] Hofreiter M, Rabeder G, Jaenicke-Despres V, et al. Evidence for reproductive isolation between cave bear populations[J]. Curr Biol, 2004, 14(1):40-43
[57] Rohland N, Hofreiter M. Ancient DNA extraction from bones and teeth[J]. Nature protocols, 2007, 2(7):1756-1762
[58] Yang DY, Eng B, Waye JS, et al. Improved DNA extraction from Ancient Bones Using Silica-Based Spin Columns[J]. American Journal of Physical Anthropology, 1998, 105(4):539-543
[59] Juras A, Makarowicz P, Chylenski M, et al. Mitochondrial genomes from Bronze Age Poland reveal genetic continuity from the Late Neolithic and additional genetic affinities with the steppe populations[J]. Am J Phys Anthropol, 2020, 172(2):176-188
[60] Rohland N, Siedel H, Hofreiter M. A rapid column-based ancient DNA extraction method for increased sample throughput[J].Molecular ecology resources, 2010, 10(4):677-683
[61] Sullivan NO, Posth C, Coia V, et al. Ancient genome-wide analyses infer kinship structure[J]. Science Advance, 2018, 4 eaao1262
[62] Gaudio D, Fernandes DM, Schmidt R, et al. Genome-Wide DNA from Degraded Petrous Bones and the Assessment of Sex and Probable Geographic Origins of Forensic Cases[J]. Sci Rep, 2019, 9(1):8226
[63]杨百全,王利君,遇长青,等.磁珠法回收纯化DNA样本[J].中国法医学杂志,2006, 21:10-11
[64] Zhao J, Liu FE, Lin S, et al. Investigation on maternal lineage of a Neolithic group from northern Shaanxi based on ancient DNA[J]. Mitochondrial DNA A DNA Mapp Seq Anal, 2017, 28(5):732-739
[65] Kalmár T, Bachrati CZ, Marcsik A, et al. A simple and efficient method for PCR amplifiable DNA extraction from ancient bones[J].Nucleic Acids Research, 2000, 28(12):E67
[66] Orlando L, Ginolhac A, Raghavan M, et al. True single-molecule DNA sequencing of a pleistocene horse bone[J]. Genome Res,2011, 21(10):1705-1719
[67] Rohland N, Siedel H, Hofreiter M. Nondestructive DNA extraction method for mitochondrial DNA analyses of museum specimens[J]. Bio Techniques, 2004, 36814-821
[68] Hervella M, Iniguez MG, Izagirre N, et al. Nondestructive methods for recovery of biological material from human teeth for DNA extraction[J]. J Forensic Sci, 2015, 60(1):136-141
[69] Gomes C, Palomo-Díez S, Roig J, et al. Nondestructive extraction DNA method from bones or teeth, true or false?[J]. Forensic Science International:Genetics Supplement Series, 2015, 5e279-e282





