非编码 RNA 在基因调控方面有着重要功能,可广泛参与基因调控的各个层面,在发育、癌症、神经功能紊乱等生物过程中都发挥重要作用[1]。在长非编码RNA 中有一重要亚类,天然反义转录本,是从已知转录序列的互补链转录的转录物[2]。有学者提出一些分子模型来解释天然反义 RNA 产生功能的分子机制,如转录干扰、RNA 遮蔽、双链 RNA 降解、翻译干扰等[3]。已经有研究表明,长非编码 RNA 与转录因子的编码基因有着更密切的联系[4]。Dlx1as 是众多天然反义 RNA 中较早被发现的,但到目前为止,关于其具体生物学功能的报导很少。Dlx1as 在基因座位上位于进化上极其保守的两个转录因子 Dlx1 与 Dlx2之间[5-6]。Evf 2 作为 Dlx1 同源蛋白 Dlx5-Dlx6 基因座位中的天然反义转录物,已被证实可以反式调节另一个同源基因 Dlx2[7]。本研究以小鼠的大脑发育为模型,检测与探讨了 Dlx1as 在发育过程中的表达,分析 了 其 对 Dlx1 编 码 基 因 产 生 影 响 的 可 能机制。
一、材料和方法
材料 pGEM-T 载体、质粒提取试剂盒、DNA 纯化试剂盒购自天根生化科技 ( 北京) 有限公司,反转录试剂盒购自全式金生物科技公司,实时定量 PCR 试剂盒、RNase-Free DNaseI 购自日本 Takara 公司,Trizol购自美国 Invitrogen 公司,T4 DNA 连接酶购自美国Promega 公 司。 E12. 5、 E14. 5、 E16. 5、 E18. 5、 P1、P7 及成年小鼠品系均为 ICR 品系,由中国医学科学院基础医学研究所实验动物中心饲养。冰冻切片机为莱卡 CM1950 ( 实验室器材) 。
组织 RNA 的提取及去 DNA 处理 取新鲜鼠脑组织,按照 1 mg 组织∶1 ml 的比例加入 Trizol,使用高速匀浆器进行破碎,之后加入 200 μl 氯仿抽提,4℃12 000 r / min离心 20 min; 吸取上清 500 μl,加入等体积异丙醇,4℃放置 1 h,4℃ 12 000 r/min 离心 20 min;弃上清,75% 乙醇 1 ml 漂洗 2 次,晾干,加入适量DEPC 水溶解。按照 10 μg 总 RNA 加入 10U DNaseⅠ与相应体积的 10 × buffer,37℃反应 30 min,70℃热失活 5 min。
RT-PCR 使用 2 μg 总 RNA 作为模版,1 μl( 50 pm) OligdT 作为引物,2 × buffer 10 μl、反转录酶1 μl、DEPC-水补足 20 μl 体系。阴性对照的 PCR 体系中未加逆转录酶。
实时定量 PCR 用等量 cDNA 作为模版,20 μl总体系,0. 5 μl-Primer-F,0. 5 μl-Primer-R,10 μl-2 ×Mix,使用 GAPDH 为内参,对 Dlx1 mRNA 与 Dlx1as 进行定量检测。实时定量 PCR 引物序列见表 1。
克隆与测序 将 RT-PCR 得到的特异条带进行切胶回收,用 DNA 纯化试剂盒纯化,将纯化的 DNA 产物连接到 pGEM-T 载体,连接产物转化至大肠杆菌 DH5α感受态细胞中,菌落 PCR 鉴定阳性克隆,接菌,送美国 Invitrogen 公司测序部进行测序。
RNA 探针制备 使用将 Dlx1as 与 Dlx1 mRNA 的片段克隆到 pGEM-3Zf 载体,分别使用 BamHⅠ与 HindⅢ进行线性化,醇沉回收线性化质粒,使用 T7 转录酶体系进行体外转录,RNase-Free DNaseⅠ消化模版,醇沉,漂洗,50μl DEPC-水溶解,加入等体积甲酰胺,标定浓度为 50 ng/μl,分装使用。
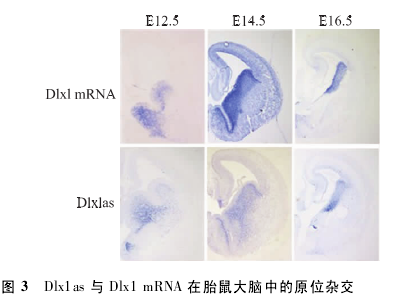
原位杂交 使用冰冻切片机,将不同天数的鼠脑进行切片,使用杂交液进行预杂交,使用0.5 ng/μl 的终浓度探针,65℃ 杂交过夜,使用 0. 2 × SSC,65℃,洗脱20 min,重复 3 次,使用地高辛抗体 1∶ 5000,4℃ ,杂交过夜,进行显色反应,中性树脂进行封闭保存。使用尼康显微镜进行拍照记录。
二、结 果
Dlx1as 分子具有多聚腺苷酸尾 使用 UCSC 公共数据库的 polyA 信号分析,在 Dlx1as 的末端有 1 个强烈的特征信号,强度比 Dlx1 mRNA 更高; 采用 OligdT和 Random primer 进行反转录,以得到的 cDNA 为模版进行 PCR 扩增,阴性对照未见条带,目的片段大小符合预期,连接 pGEM-T 载体,测序正确 ( 图 1) 。
Dlx1as 和 Dlxl mRNA 在小鼠大脑胚胎期高表达Dlx1as 和 Dlx1 mRNA 在神经发育过程中出现了一段时间 内 的 高 表 达,从 E14. 5 持 续 到 E18. 5,除 在E12. 5 和成年小鼠中有显著性差异外,其余 4 个时间点均没有显著性差异。使用曲线拟合,计算得出 R2=0. 71 ,P = 0. 19 ( 图 2) 。
Dlx1as 与 Dlx1 mRNA 在空间表达上具有互补性从 E12. 5、E14. 5 和 E16. 5 的时间点中,都能够看出二者的表达部位有明显的差别,虽然二者均较特异表达于大脑腹侧生发区,但 Dlx1 mRNA 在室周区较室周下表达量更高,而 Dlx1as 则与之相反 ( 图 3) 。
三、讨 论
越来越多的证据表明,一些长非编码 RNA 的转录使用和 mRNA 相同的转录机器,具有多聚腺苷酸尾,有可变剪切形式。本研究鉴定了经典的天然反义RNA—Dlx1as 具有多聚腺苷酸尾。在特定的时空表达的RNA 分子,很可能会参与特定的生物学过程。Dlx1as在小鼠胚胎期大脑中高表达,与成年的低表达形成鲜明的对比,揭示 Dlx1as 很可能在大脑发育过程成具有重要功能。而且 Dlx1as 与 Dlx1 mRNA 的表达趋势非常类似,笔者认为可能是整个基因座位的转录被激活所导致,当然也不能排除二者的转录分别受到调控,还需要进一步的验证转录的上游控制。
天然反义 RNA 很多都通过影响 mRNA 来产生效应,例如天然反义 RNA Star 的表达可以降低 StAR 的蛋白表达,在体敲低 Bdnf-AS 的表达水平,可以使 Bdnf 的表达水平得到大幅度的增强[8-9]。因此笔者推测 Dlx1as 很可能也是通过类似方式来实现其功能的。本研究通过原位杂交,发现 E12. 5、E14. 5 和 E16. 5 的胎脑中,在脑室室周,Dlx1as 与 Dlx1 mRNA 有着明显的位置不同,甚至是接近互补的存在,由此推测 Dlx1as 可能是通过对 Dlx1 mRNA 的负调控来发挥功能的。在笔者撰写文章的同时,有新加坡的实验室发表了关于 Dlx1as 敲除小鼠的研究,并提出了Dlx1as 通过负向调节Dlx1 mRNA的分子模型,与笔者推测相符[10]。至于更加详细的分子调控机制,还需要进行更加深入细致的研究。
参 考 文 献
[1] Chen LL,Carmichael GG. Long noncoding RNAs in mamma-lian cells: what, where, and why [J]. Wiley InterdiscipRev RNA,2010 ,1( 1) : 2-21.
[2] Lapidot M,Pilpel Y. Genome-wide natural antisense tran-scription: coupling its regulation to its different regulatorymechani- sms [J]. EMBO Rep,2006 ,7( 12) : 1216-1222.
[3] Li K,Ramchandran R. Natural antisense transcript: a con-comitant engagement with protein-coding transcript [J]. On-cotarget,2010,1( 6) : 447-452.
[4] Werner A. Natural antisense transcripts [J]. RNA Biol,2005,2( 2) : 53-62.
[5] McGuinness T,Porteus MH,Smiga S,et al. Sequence,or-ganization,and transcription of the Dlx-1 and Dlx-2 Locus[J]. Genomics,1996 ,35( 3) : 473-485.
[6] Panganiban G,Rubenstein JL. Developmental functions of theDistal-less / Dlx homeobox genes [J]. Development,2002,129( 19) : 4371-4386.
[7] Feng J,Bi C,Clark BS,et al. The Evf-2 noncoding RNA istranscribed from the Dlx-5 /6 ultraconserved region and func-tions as a Dlx-2 transcriptional coactivator [J]. Genes Dev,2006,20( 11) : 1470-1484.
[8] Castillo AF,Fan J,Papadopoulos V,et al. Hormone-depend-ent expression of a steroidogenic acute regulatory protein natu-ral antisense transcript in MA-10 mouse tumor leydig cells[J]. PLoS One,2011,6( 8) : e22822.
[9] Modarresi F,Faghihi MA,Lopez-Toledano MA,et al. Inhibi-tion of natural antisense transcripts in vivo results in gene-spe-cific transcriptional upregula-tion [J]. Nat Biotechnol,2012,30( 5) : 453-459.
[10] Kraus P,Sivakamasundari V,Lim SL,et al. Making sense ofDlx1 antisenseRNA [J]. Dev Biol,2013,376 ( 2) : 224-235.