摘 要: 利用动物线粒体12S rRNA和16S rRNA基因序列片段对动物角制品进行种属鉴别,为DNA条形码技术在角制品分子鉴定提供有效方法。收集犀牛角、水牛角、黄牛角和山羊角作为研究材料,采用优化提取步骤的商业化提取试剂盒进行基因组DNA的提取,以提高基因组DNA的浓度及质量。采用PCR扩增及测序技术获得所有样本12S rRNA和16S rRNA基因部分序列,使用Blast程序分别进行序列比对分析,通过下载Genbank相关物种的12S rRNA和16S rRNA序列,分别比较两个基因片段种内和种间距离,分别构建系统进化树。结果显示,12S rRNA序列所有样本扩增成功,16S rRNA序列仅犀牛角和黄牛角扩增成功。遗传距离分析显示各检材种间差异均大于种内差异,收集检材在12S rRNA和16S rRNA引物构建的进化树中表现出良好的单系性和较高的置信度。表明12S rRNA基因片段的DNA条形码技术对动物角制品及其它混伪品具有更好的鉴别能力,可提高物种鉴定的准确性和灵敏度。
关键词 : 动物角制品; DNA条形码技术;动物线粒体;
Abstract: Animal mitochondria 12 S rRNA and 16 S rRNA gene sequences fragment was used to identify the animal horn products,by which to explore an effective method for molecular identification of horn products by DNA barcoding.The genomic DNA of rhino horn,buffalo horn,ox horn and goat horn were extracted with a commercial extraction kit.The 12 S rRNA and 16 S rRNA sequences of GenBank related species were downloaded and the intra-specific and inter-specific genetic distances of them were compared and phylogenetic trees were constructed for the two gene fragments.It was found that the 12 S rRNA sequence was amplified successfully with all samples while the 16 S rRNA sequence was amplified only in rhino horn and cattle horn.Genetic distance analysis indicated that,the difference between species was greater than that within species and the collected samples showed good monophyletic and high confidence in the phylogenetic tree constructed by 12 S rRNA and 16 S rRNA primers.It is concluded that DNA barcoding of 12 S rRNA gene fragments is more reliable in identifying animal horn products and other counterfeits,which can improve the accuracy and sensitivity of species identification.
Keyword: animal horn products; DNA barcoding; animal mitochondria;
动物角是哺乳动物头部表皮及真皮角化的产物,在人类生活中具有广泛的应用[1]。不仅是各种手工艺品及角器的原料之一,同时大部分角制品还具有一定的药用价值,是制药工业的原料之一,因此角制品可以说是动物资源中重要的组成部分。

角类工艺品无论是在国内还是国外,都因被认为具有极大的升值空间而受到人们不断追捧和关注,而濒危动物的角制品与其替代品之间在价格上存在着巨大差异,导致市面上出现大量仿品和赝品,不法分子更是通过走私、贩卖等途径从中牟取暴利[2]。同时,角制品作为常用药材原料之一,一般具有清热、镇痛、镇静等作用,但由于入药需要,会将其制成粉状、丝状或片状等形状进行销售,导致角制品的种类难以辨识,也无完整的性状鉴定特征,执法部门和消费者更是无法鉴别药材基原物种。正因源头把控技术的缺陷,动物类药材中又隐藏着巨大的经济效益,药市中常采用其他廉价、虚假的角制品替代珍贵药材原料,以次充好、以假乱真,药材中出现伪品、造假的情况也越来越普遍[3,4,5,6,7]。
各种动物角制品原料在外观上均具有很多相似之处,需要鉴定人员具备一定的理论知识和丰富的经验来进行辩别,而采用形态学和组织学鉴定方法易出现偏差,造成鉴别工作的困难[8]。近代以来,随着分子鉴定技术的快速发展,DNA条形码技术已成为世界公认对生物物种进行精准鉴定的一种方法[9,10]。该方法不仅具有近缘物种之间的特异性及种属之间的保守性,还不会受到不同时期物种生长状态和外观形态的影响,易于标准化普及和操作,已被广泛应用于多种动物和植物物种鉴定中[11,12,13,14]。其中动物线粒体DNA不仅严格按照母系遗传,而且符合动物进化速率快的特点,即便是亲缘关系较近的种属也能被有效区分,已在分类进化等领域得到广泛的应用[15,16,17]。但目前针对动物角制品进行准确有效鉴别的研究颇少,给监管部门和执法部门在物种鉴别上造成了极大的困难,无法为仲裁提供科学依据,因此为动物角制品物种鉴定与识别提供科学准确的方法至关重要。
本研究采用线粒体12S rRNA和16S rRNA基因作为DNA条形码,对犀科和牛羊科动物角制品进行鉴别研究,以期建立适用于动物角制品鉴定的DNA条形码技术,为动物角制品分类鉴定和物种保护提供理论依据和技术支持。
1、 材料与方法
1.1、 材料
本研究选取濒危物种、濒危物种常用替代物种及掺假造假中常见的角制品作为研究对象,包括犀牛角、水牛角、黄牛角和山羊角。其中犀牛角为东北林业大学赠与,其他角制品为本研究收集样本。
1.2 试剂和设备
柱式骨骼DNA out提取试剂盒(北京天恩泽基因科技有限公司);TaKaRa Ex Taq (货号:RR001,宝日医生物技术有限公司);引物探针均由Invitrogen公司合成。
台式高速冷冻离心机(3-18K,德国Sigma公司);电子天平(AL104,瑞士梅特勒-托利多);电热恒温水浴锅(DK-8D,上海一恒);阶梯PCR仪(Veriti 96,美国ABI)。
1.3、 方法
1.3.1、 基因组DNA提取
各种动物角制品检材用超纯水清洗干净,使用75%乙醇溶液擦洗各样本表面后,放置于已进行消毒处理的通风橱内30 min备用。使用小型电磨机将犀牛角、水牛角、黄牛角和山羊角检材磨制成粉末后,使用液氮研磨至细粉。取各检材粉末60mg,参照柱式骨骼DNA out提取试剂盒的优化步骤进行基因组DNA的提取[18]:65℃水浴保温时间延长至1 h;采用0.3 m L溶液B (提取试剂盒所包含试剂)和0.2 m L氯仿抽提的操作增加至两次,以加大角质细胞的裂解效率,更好的去除蛋白质、提高基因组DNA的浓度及质量,其余提取步骤参照试剂盒说明书。
1.3.2、 PCR引物
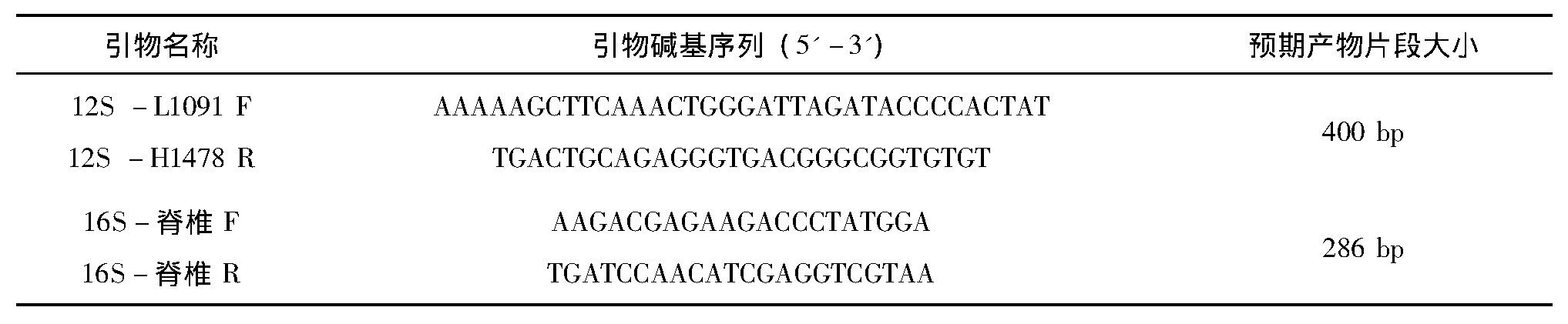
采用动物线粒体12S rRNA基因通用引物(L1091、H1478)[19]和脊椎动物线粒体16S rRNA基因通用引物对对各角制品检材进行PCR扩增反应,各通用引物对序列详见表1。
表1 PCR反应引物序列
1.3.3 、PCR扩增
以提取的各检材DNA作为模板,分别采用线粒体12S rRNA基因和16S rRNA基因的引物进行PCR扩增反应。
PCR扩增反应体系:0.25μL TaKaRa Ex Taq(5U/μL),5μL 10×Ex Taq buffer (Mg2+plus),4μL dNTP Mixture (2.5 mmol/L),1μL各相应上游引物(10 mol/L),1μL各相应下游引物(10 mol/L),2μL DNA模板,36.75μL ddH2O,总反应体系共计50μL。
PCR扩增程序:98℃变性10 s,55℃退火30 s,72℃延伸60 s,40个循环;72℃延伸10 min。
各检材的扩增产物经2%琼脂糖凝胶电泳检测,对条带清晰的扩增产物进行回收,送至生工生物工程有限公司进行双向测序,测序引物同为扩增引物。
1.3.4、 序列比对
采用DNA Star软件中的Seq Man程序对测序公司的序列结果进行拼接处理,去除引物区并对碱基进行校对。将各角制品的线粒体12S rRNA和16S rRNA基因片段序列在GenBank数据库中进行Blast程序比对和相似性分析。
1.3.5、 序列数据分析
根据Blast程序比对结果,自NCBI数据库中获得相关物种序列。应用CLC Genomics Wotkbench软件基于Kimura 80模型计算出各基因种内、种间遗传距离。最后,基于邻接法(Neighbor-joining,NJ)分别构建12S rRNA和16S rRNA基因片段的系统发育进化树,Bootstrap自展检测1 000次以检验各分支的置信度。
2 、结果分析
2.1 、基因序列分析
对各检材的基因组DNA分别采用12S rRNA和16S rRNA基因片段进行PCR扩增反应,扩增片段目标条带清晰,得到约480 bp大小的12S rRNA基因序列和约320 bp大小的16S rRNA基因序列,各片段大小分别与预期目标片段大小相符合。测序结果在GenBank数据库中分别进行Blast比对,结果如表2所示。其中犀牛角的12S rRNA基因片段与GenBank数据库中非洲白犀(Ceratotherium simum)的一致性达99.50%~100.00%,与黑犀(Diceros bicornis)的一致性为96.00%~96.25%;16S rRNA基因片段与GenBank数据库中非洲白犀(Ceratotherium simum)的一致性高达100.00%,与黑犀(Diceros bicornis)的一致性为96.05%。现世界残存的五种犀牛中白犀和黑犀同属非洲,两者本身就存在较近的亲缘关系,应是造成犀牛角测序比对后两个物种同时存在的原因,但比对结果中黑犀的一致性只有96.00%,相对较低,犀牛角的所属物种应判定为非洲白犀(Ceratotherium simum)。从比对结果看,在采用16S rRNA基因片段扩增时水牛角和山羊角未出现目标条带,无法进行后续测序等试验。其它物种测序结果与GenBank数据库中参考序列比对后一致性均较高。
表2 动物角制品的12S rRNA和16S rRNA基因片段比对结果

注:“—”表示无目标扩增条带。
2.2 、遗传距离分析结果
依据序列比对鉴定结果,自NCBI下载相关物种序列,物种参考序列信息见表3。采用CLC Genomics Wotkbench软件根据Kimura 80模型分别计算出基于12S rRNA和16S rRNA基因的种内种间遗传距离,结果见表4和表5。结果表明,对于12S rRNA基因片段,犀牛角所属物种的种内遗传距离为0.003,种间距离范围为0.02~0.07;黄牛角和水牛角的种内遗传距离为0.003和0.02,种间遗传距离范围为0.07~0.08;山羊角种内遗传距离为0.00 (见表4的13行与11列的交汇处),种间遗传距离为0.04;犀科、牛羊科科间遗传距离在0.08~0.18之间。对于16S rRNA基因片段,犀牛角所属物种的种内遗传距离为0.00 (见表5的5行与3列的交汇处),种间距离范围为0.04~0.09;黄牛角种内遗传距离为0.01,种间遗传距离为0.07;水牛角及山羊角无扩增及测序结果,种间遗传距离分别为0.07和0.08;犀科、牛羊科科间遗传距离在0.11~0.24之间。所涉及的物种种内遗传距离均小于种间遗传距离,同时各种间遗传距离值满足高于Hebert推荐的种间最小遗传距离值0.020[9]。
表3 线粒体12S rRNA、16S rRNA物种鉴定参考序列信息

表4 基于12S rRNA基因片段的Kimura 80遗传距离
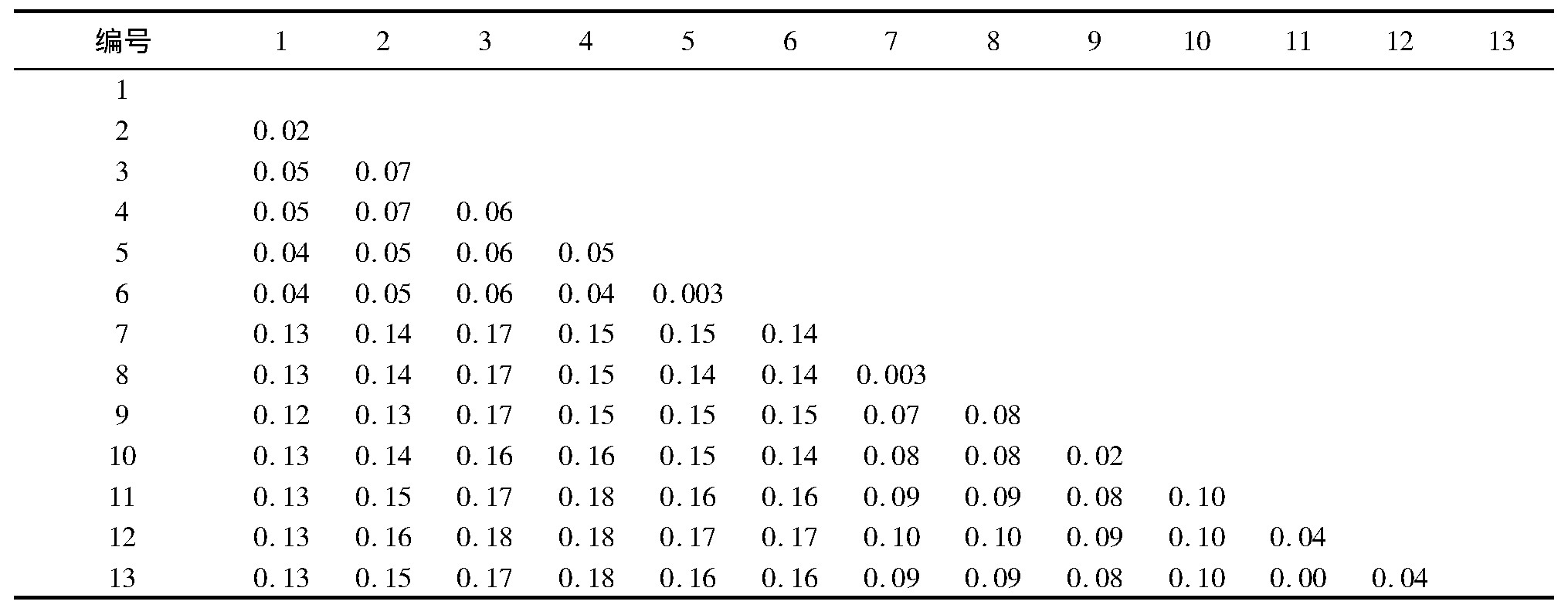
注:1.印度犀(AY739618.1);2.爪哇犀(AJ245724.1);3.苏门达腊犀(AJ245722.1);4.黑犀(AJ245721.1);5.白犀(X86942.1);6.犀牛角(12S rRNA);7.黄牛(AJ885201.1);8.黄牛角(12S rRNA);9.水牛(AJ846850.1);10.水牛角(12SrRNA);11.山羊(AJ885199.1);12.绵羊(AJ885200.1);13.山羊角(12S rRNA)。
表5 基于16S rRNA基因片段的Kimura 80遗传距离
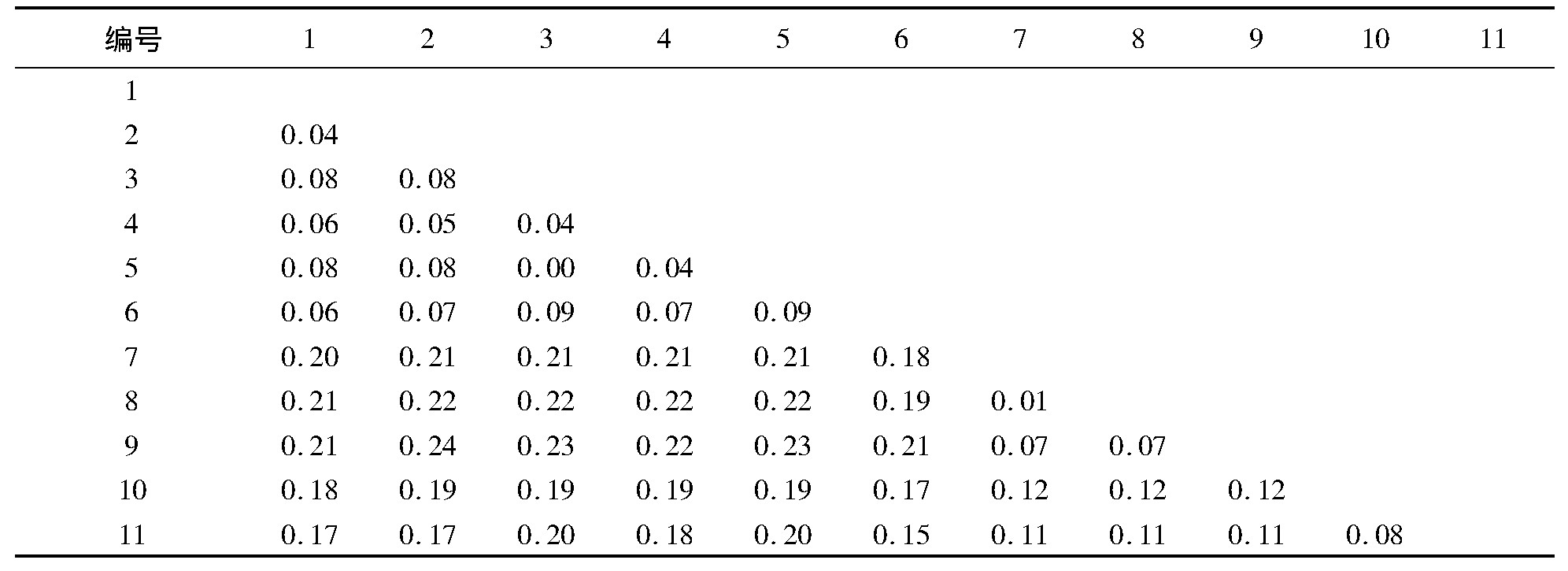
注:1.印度犀(X97336.1);2.爪哇犀(KY117574.1);3.白犀(NC_001808.1);4.黑犀(FJ905814.1);5.犀牛角(16SrRNA);6.苏门达腊犀(MF066643.1);7.黄牛(NC_006853.1);8.黄牛角(16S rRNA);9.水牛(NC_049568.1);10.山羊(NC_005044.2);11.绵羊(NC_001941.1)。
2.3 、系统发育进化树构建分析
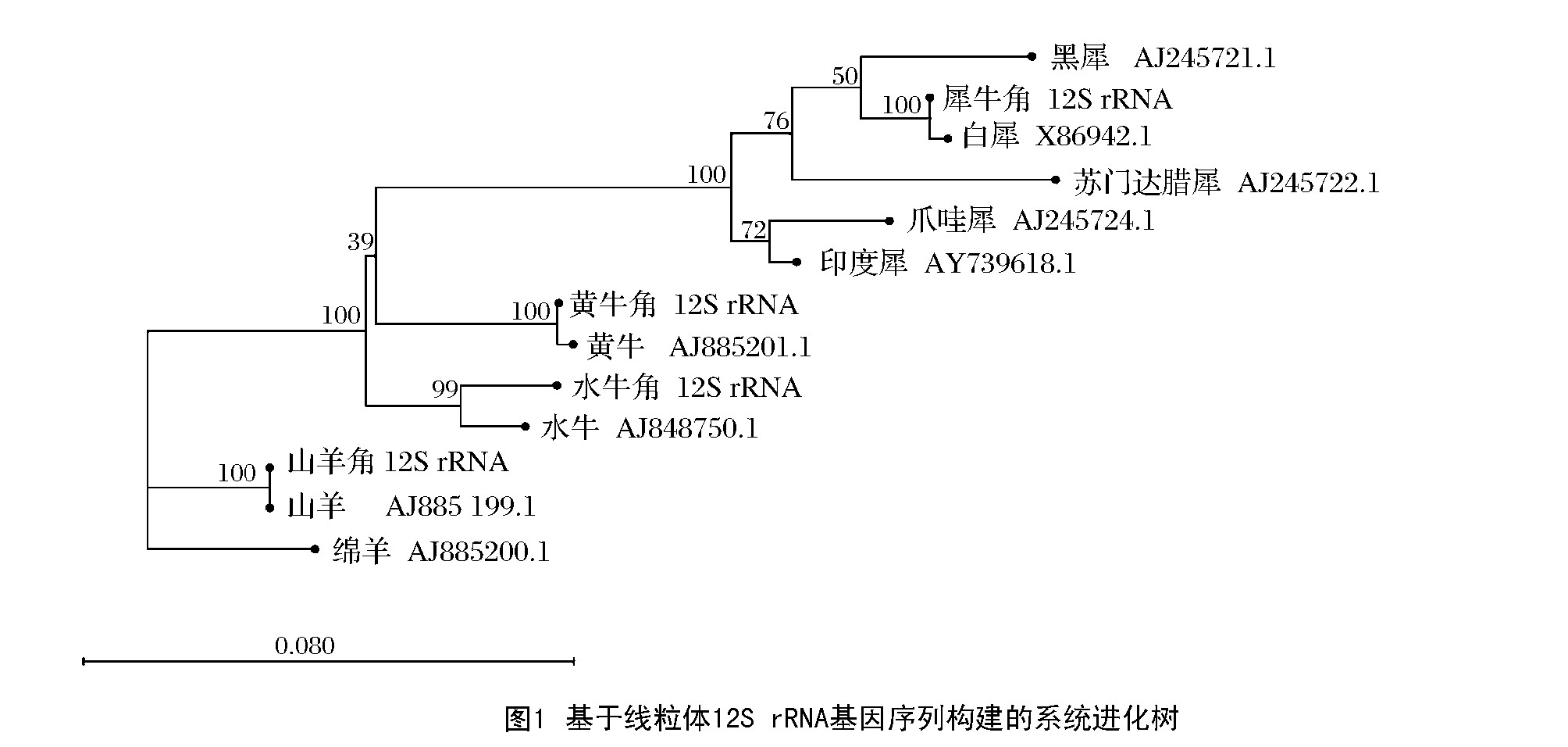
运用邻接法将试验所得各种角制品的线粒体12S rRNA和16S rRNA基因片段序列与GenBank上下载的参考序列分别构建系统发育进化树,结果如图4和图5所示。从发育进化树上可以看出,两个片段的犀科、牛科及羊科各自形成独立的大分支,不同物种之间界限也十分清晰;犀科中的五种物种形成独立的进化分支,试验测定的犀牛角序列与非洲白犀(Ceratotherium simum)聚在一支,12S rRNA和16S rRNA片段的置信度分别为100.00%和99.00%;测定黄牛角序列与黄牛(Bos taurus)聚为一支,12S rRNA和16S rRNA片段的置信度分别为100.00%和97.00%;12S rRNA片段中水牛角和山羊角分别与水牛(Bubalus bubalis)和山羊(Capra hircus)聚为一支,置信度分别为100.00%和99.00%。表明两个片段能够较好的对物种进行区分和鉴别。同时在两个序列构建的系统发育进化树中,犀牛角和非洲白犀先聚为一支,再与黑犀聚为一支,置信度均在可信范围内,验证了两个物种具有较近的亲缘关系。
3 、结论
近年来,基于DNA条形码技术在基因水平上的快速发展,使其在动物物种鉴定方面得到了广泛的应用。线粒体DNA作为动物起源进化分子水平的研究对象,最常用的标记中包含12S rRNA和16S rRNA基因,两者片段保守的同时却又具有一定的进化速率,其中线粒体12S rRNA基因主要用于动物种属的鉴定,而作为进化分子钟的线粒体16S rRNA基因则主要应用于群体结构和系统发育领域[20,21]。其中陈云霞等基于12S rRNA条形码序列对涉案犀牛角制品进行比对分析及鉴定[22];刘海情等应用12S rRNA和16S rRNA序列研究龟鳖类系统进化关系[17];陈宏等采用线粒体COI、16S rRNA和12S rRNA 3种微型条形码相结合的方法研究出16S rRNA的微型条形码技术可有效鉴别炮山甲及其混伪品[23]。但动物角制品系统鉴别方法未见相关报道,针对动物角制品自身角质化、质地坚韧的特点造成基因组DNA提取困难和含量偏低的问题,本研究结合参考文献及自身试验材料特点,采用有针对性的商业化提取试剂盒进行基因组DNA的提取并优化其提取步骤,以提高基因组DNA的提取效率,为动物角制品在法律仲裁及药物鉴定中提供技术支撑。
为了提高动物角制品物种鉴定的准确性,本研究基于线粒体12S rRNA和16S rRNA基因片段分别构建DNA条形码,结果显示在动物角制品中12S rRNA基因引物的DNA扩增效果要高于16S rRNA基因引物,12S rRNA基因引物将收集的全部角制品检材扩增成功,而16S rRNA基因引物仅在部分角制品检材中扩增成功,其主要原因为DNA模板问题或引物不适用造成的扩增失败,但16S rRNA基因引物扩增失败的样本DNA,12S rRNA基因引物却能扩增成功,排除DNA模板的因素,认为此16S rRNA基因引物不完全适用动物角制品鉴别,原因可能是所设计的引物未完全或是仅有部分落在动物角制品基因的保守序列内,造成部分检材最终扩增反应的失败。根据遗传距离和系统发育进化树的分析结果表明:收集的犀牛角、黄牛角、水牛角和山羊角检材各自种内遗传距离明显小于种间遗传距离,说明能将各角制品进行有效的区分和鉴别;进化树中各科角制品均形成独立的分支,各属种的所有个体都能很好的聚集在一起并形成较高的置信值,确定收集检材和研究结果相一致。通过综合分析以上鉴定方法,12S rRNA基因片段的DNA条形码技术对动物角制品及其它混伪品具有更好的鉴别能力,在分子水平上提高了动物角制品物种鉴定的准确性和灵敏度,可作为DNA条形码技术在动物角制品物种鉴定上的依据,具有重要的使用价值和应用前景。
参考文献
[1]李圣清.犀牛角及其仿制品的研究[D] .昆明:昆明理工大学, 2011:1-67.
[2]周晶梅.犀牛角及其制品鉴定识别方法的研究[D] .哈尔滨东北林业大学, 2010:1-41.
[3]黎跃成.中国药用动物原色图鉴[M] .上海: 上海科学技术出版社, 2010.
[4]张红印,刘冬,刘侗,等. DNA条形码鉴定技术在动物类中药材鉴定领域的研究进展[] .吉林中医药, 2014,37(4)-:378-381.
[5]王亮,陈万生.山羊角研究概况[J] .江苏中医药, 2019,51(12):86-89.
[6]刘旭朝,周丽思,刘金欣,等.基于COI序列的水牛角及其易混伪品DNA条形码鉴定研究[J] .药学学报, 2017,52(3):494-499.
[7] COGHLAN M L,HAILE J,HOUSTON J,et al. Deep sequencing of plant and animal DNA contained within traditional Chinese medicines reveals legality issues and health safetyconcerns[J]. PLo S Genetics, 2012, 8(4):1002657.
[8]赵竹,宋云,许瑾,等.基于C0I基因对犀角工艺品真伪的鉴别[J] .植物检疫, 2013,27(2):68-71.
[9]李婵,谢雪那,蔡炫,等.穿山甲属动物的DNA条形码及在穿山甲商品鉴定中的应用[J] .中国现代中药, 2019,21(9):1221-1228.
[10]岳巧云,邱德义,胡佳,等. DNA条形码医学媒介生物快速准确鉴定的利器[J] .检验检疫学刊, 2013,23(5);60-63.
[11] KHAN S A,BAESHEN M N,RAMADAN H A,et al.ITS2:An ideal DNA barcode for the arid medicinal plant rhazya stricta[J]. Pharmaceut Medicine ,2019,33(1):53-61.
[12] SHI R,HU Z,LU H.et al. Hierarchical nanostructuring array enhances mid-hybridization for accurate herbal identification via ITS2 DNA barcode[J]. Analytical Chemistry,2020.92(2):2136-2144.
[13] YU M,JIAO L C,GOU J,et al. DNA barcoding of vouchered xylarium wood specimens of nine endangered Dalbergia species[J]. Planta,2017.,246.1165-1176.
[14] HOFMANN T,KNEBELSBERGER T,KL OPPMANN M,et al. Egg identification of three economical important fish species using DNA barcoding in comparison to a morphologicaI determination[J]. Journal of Applied Ichthyology,2017,33(5):925-932.
[15]李笑,谭朝阳,徐德宏,等. DNA条形码技术在中药材鉴定中的应用[J] .安徽农业科学, 2020,48(2):16-19.
[16]蒙晓平,白晓军,梅燕,等. COX1、12S rRNA、16S rRNA基因复合扩增种属鉴定研究[J] .中国法医学杂志, 2015,30(6):560-566.
[17]刘海情,刘楚吾, 刘丽.基于12S rRNA和16S rRNA序列的龟鳖类系统进化特征研究[J] .南方农业学报, 2012 ,43(11):1758-1764.
[18]刘旭朝,周丽思,刘金欣,等. 于C0I序列的水牛角及其易混伪品DNA条形码鉴定研究[J].药学学报, 2017,52(3):494-499.
[19] FOLMER O,BLACK M.HOEC W,et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit | from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology, 1994,3(5):294-299.
[20]韦健红.地龙类药材DNA条形码分子鉴定的研究[D].广州:广州中医药大学, 2012:1-80.
[21]宋洁,张小芳,刁雪淘,等.线粒体DNA 12S rRNA、16S rRNA研究进展[J] .安徽农业通报, 2008, 14(9)-42-43.
[2]陈云霞,郭海涛.基于12S rRNA的犀牛角制品DNA条形码鉴定分析[J].中国法医学杂志, 2016,31(5):473-475.
[23]陈宏,王红雨,何铭琪,等.炮山甲及其混伪品的DNA微型条形码鉴定研究[J] .中药材. 2019,1(10):2268-2273.





